理论上我前面提到的GEOquery包就可以根据一个GSE索引号来获取NCBI提供的所有关于这个GSE索引号的数据了,包括metadata,表达矩阵,soft文件,还有raw data
但是很多时候,那个metadata并不是很整齐,而且一个个下载太麻烦了,所以就需要用R的bioconductor的另一个神奇的包了GEOmetadb
它的示例:http://bioconductor.org/
里面还是很多数据库基础知识的
代码托管在github,它的示例代码是这样连接数据库的:
library(GEOmetadb)
if(!file.exists('GEOmetadb.sqlite')) getSQLiteFile()
file.info('GEOmetadb.sqlite')
con <- dbConnect(SQLite(),'GEOmetadb.sqlite')
dbDisconnect(con)
但是一般不会成功,因为这个包把它的GEOmetadb.sqlite文件放在了国外网盘共享,在国内很难访问,推荐大家想办法下载到本地
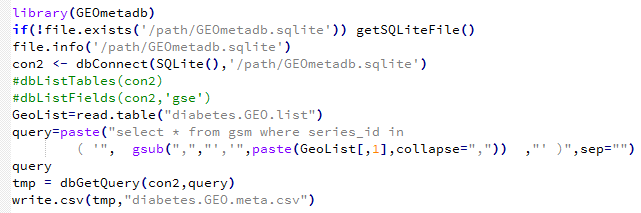 用这个代码就会成功了,需要自己下载GEOmetadb.sqlite文件然后放在指定目录:/path/GEOmetadb.sqlite 需要自己修改
我们的diabetes.GEO.list文件内容如下:
GSE1009
GSE10785
GSE1133
GSE11975
GSE121
GSE12409
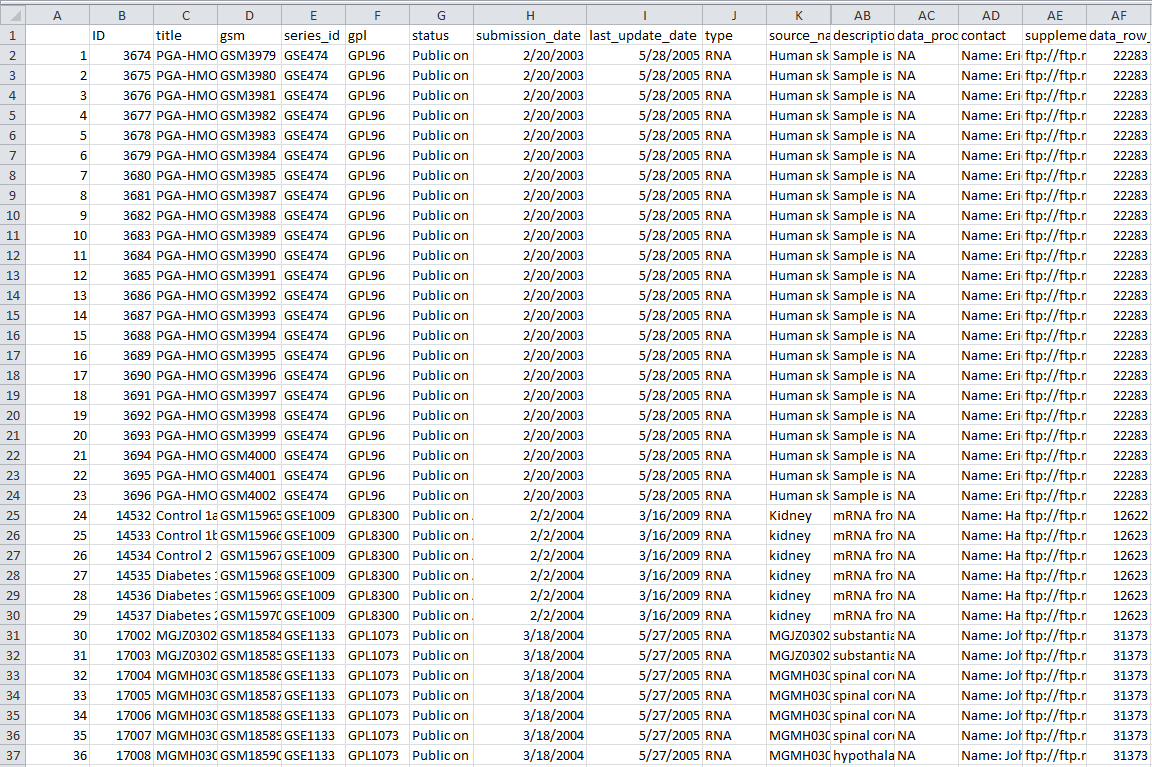
那么会产生的表格文件如下:共有32列数据信息,算是蛮全面的了
用这个代码就会成功了,需要自己下载GEOmetadb.sqlite文件然后放在指定目录:/path/GEOmetadb.sqlite 需要自己修改
我们的diabetes.GEO.list文件内容如下:
GSE1009
GSE10785
GSE1133
GSE11975
GSE121
GSE12409
那么会产生的表格文件如下:共有32列数据信息,算是蛮全面的了