这个也是基于bam文件来的,判断chrY独有区域的覆盖度及测序深度
首先下载chrY独有区域的记录文件,https://www.familytreedna.com/documents/bigy_targets.txt
然后用samtools depth来统计测序深度,samtools depth $i |grep 'chr[XY]'
depth统计结果文件如下:
mzeng@ubuntu:/home/jmzeng/gender_determination$ head Sample3.depth
chrX 60085 1
chrX 60086 1
chrX 60087 1
chrX 60088 1
chrX 60089 1
chrX 60090 1
chrX 60091 1
chrX 60092 1
chrX 60093 1
chrX 60094 1
chrX 60085 1
chrX 60086 1
chrX 60087 1
chrX 60088 1
chrX 60089 1
chrX 60090 1
chrX 60091 1
chrX 60092 1
chrX 60093 1
chrX 60094 1
然后我随便写了一个脚本来对测序深度文件进行再统计,统计覆盖度及测序深度
[perl]
open FH,"bigy_targets.txt";
while(<FH>){
chomp;
@F=split;
$all+=$F[2]-$F[1]+1;
foreach ($F[1]..$F[2]){
$h{$_}=1;
}
}
close FH;
open FH,$ARGV[0];
while(<FH>){
chomp;
@F=split;
next unless $F[0] eq 'chrY';
if (exists $h{$F[1]}){
$pos++;
$depth+=$F[2];
}
}
close FH;
$average=$depth/$pos;
$coverage=$pos/$all;
print "$pos\t$average\t$coverage\n" ;
[/perl]
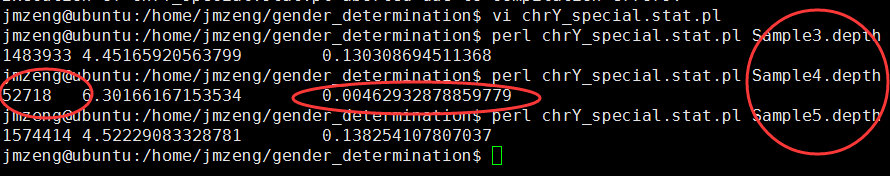
这样对那三个样本结果如下:
可以看到只有sample4,是覆盖率极低的,而且记录到的pos位点也特别少,所以她是女性!
这里测序深度没有意义。
chrY独有区域文献:https://www.familytreedna.com/learn/wp-content/uploads/2014/08/BIG_Y_WhitePager.pdf
try the GATK "DepthOfCoverage" ?http://www.broadinstitute.org/gsa/wiki/index.php/Depth_of_Coverage_v3.0
or you can run 'samtools pileup' and calculate the mean value for the coverage column.