我们知道了生存分析,就是随着时间的流逝,死亡率是如何增加的,一般是用KM法来估计生存函数,然后画个图即可!而根据某些因子把样本分组,可以看到他们死亡率的变化趋势显著的不同,这就说明了我们的这个因子是非常有效的分类方式,这个因子可以是一个biomarker,也可以某些其它指标!
甚至,我们还可以用cox模型来分析这个因子是如何影响生存函数的,那个稍后再讲
这里,我们就简单讲一个例子,是TCGA里面卵巢癌的数据,根据甲基化数据分成了4个组,那么我们就下载这四个组样本的临床数据,
看看这样分组后,他们的死亡率变化趋势是不是有显著区别!
数据是用cgdsr下载的:
这个包的说明见:http://www.bio-info-trainee.com/?p=1257
library(cgdsr)
mycgds <- CGDS('http://www.cbioportal.org/public-portal/')
test(mycgds)
all_TCGA_studies <- getCancerStudies(mycgds)
all_tables <- getCaseLists(mycgds, 'ov_tcga_pub')
all_dataset<- getGeneticProfiles(mycgds, 'ov_tcga_pub')
#BRCA1 <- getProfileData(mycgds, my_gene, my_dataset, my_table)
ov_tcga_pub_meth1<- getClinicalData(mycgds, all_tables[8,1])
ov_tcga_pub_meth2<- getClinicalData(mycgds, all_tables[9,1])
ov_tcga_pub_meth3<- getClinicalData(mycgds, all_tables[10,1])
ov_tcga_pub_meth4<- getClinicalData(mycgds, all_tables[11,1])
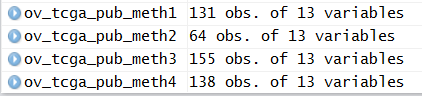
下载之后的数据如下:
根据甲基化数据,把癌症病人分成了4组,我们的临床数据记录了13项,但是我们只需要用到OS_MONTHS和OS_STATUS就可以来估计KM生存函数,画出生存曲线啦!
无病生存期(Disease-free survival,DFS)的定义是指从随机化开始至疾病复发或由于疾病进展导致患者死亡的时间。该指标也常作 为抗肿瘤药物III期临床试验的主要终点。某些情况下,DFS与OS相比,作为终点比较难以记录,因为它要求认真随访,及时发现疾病复发,而且肿瘤患者的 死亡原因也很难确定。肿瘤患者常有合并症(如,心血管病),这些合并症可能会干扰对DFS的判断。并且,肿瘤患者常死于医院外,不能常规进行尸检。
总生存期(Overall survival,OS)的定义是指从随机化开始至因任何原因引起死亡的时间。该指标常常被认为是肿瘤临床试验中最佳的疗效终点。如果在生存期上有小幅度的提高,可以认为是有意义的临床受益证据。作为一个终点,生存期应每天进行评价,可通过在住院就诊时,通过与患者直接接触或者通过电话与患者交谈,这些相对比较容易 记录。确认死亡的日期通常几乎没有困难,并且死亡的时间有其独立的因果关系。当记录至死亡之前的失访患者,通常截止到最后一次有记录的、与患者接触的时间。
library(survival)
attach(ov_tcga_pub_meth1)
## 估计KM生存曲线
my.surv <- Surv(OS_MONTHS,OS_STATUS=='DECEASED')
kmfit1 <- survfit(my.surv~1)
summary(kmfit1)
plot(kmfit1)
##我们很容易看到,随着感染癌症的时间延长,病人的死亡率到了一定程度就不增加了,有些病人熬过了这个癌症,或者说,到我们拿到数据为止,他们还没有死亡!
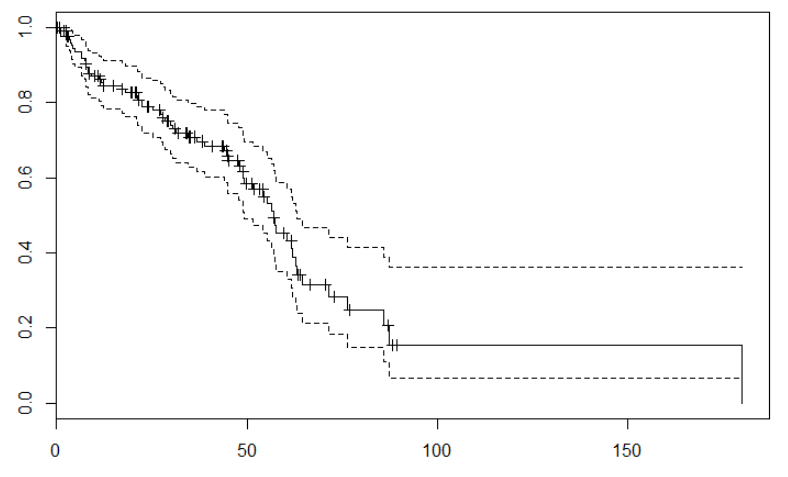
## 随便取一根因子来分组TUMOR_STAGE_2009估计KM生存曲线
kmfit2 <- survfit(my.surv~TUMOR_STAGE_2009)
summary(kmfit2)
plot(kmfit2,col = rainbow(length(unique(ov_tcga_pub_meth1[,13]))))
detach(ov_tcga_pub_meth1)
##可以看到,我们根据病人的TUMOR_STAGE_2009把他们分成了这些组,不同组的生存曲线不一样,但是我们肉眼无法看出它们这些组直接的生存率是否有显著差异!我们需要做统计检验!
我们就不对这个进行检验了,我们还是用下面的代码来对甲基化数据的分组来做检验,看看我们的分组是否有效!
ov_tcga_pub_meth1$sample<- rownames(ov_tcga_pub_meth1)
ov_tcga_pub_meth2$sample<- rownames(ov_tcga_pub_meth2)
ov_tcga_pub_meth3$sample<- rownames(ov_tcga_pub_meth3)
ov_tcga_pub_meth4$sample<- rownames(ov_tcga_pub_meth4)
ov_tcga_pub_meth1$type<- 'meth1'
ov_tcga_pub_meth2$type<- 'meth2'
ov_tcga_pub_meth3$type<- 'meth3'
ov_tcga_pub_meth4$type<- 'meth4'
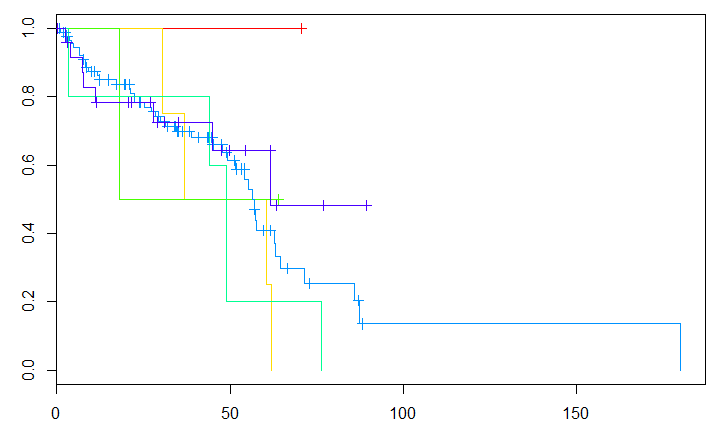
dat=rbind(ov_tcga_pub_meth1,ov_tcga_pub_meth2,ov_tcga_pub_meth3,ov_tcga_pub_meth4)
attach(dat)
## 根据分组type估计KM生存曲线
my.surv <- Surv(OS_MONTHS,OS_STATUS=='DECEASED')
kmfit3 <- survfit(my.surv~type)
summary(kmfit3)
plot(kmfit3,col = rainbow(4))
##由图中可以看到甲基化数据分成的4个组,生存率差异还是蛮大的
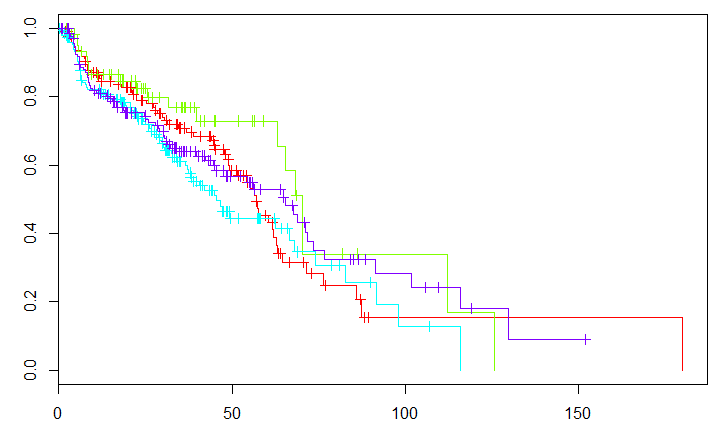
##用图形方法检验PH假设
plot(kmfit3,fun='cloglog',col = rainbow(4))
# 检验显著性
survdiff(my.surv~type, data=dat)
# 用strata来控制协变量的影响
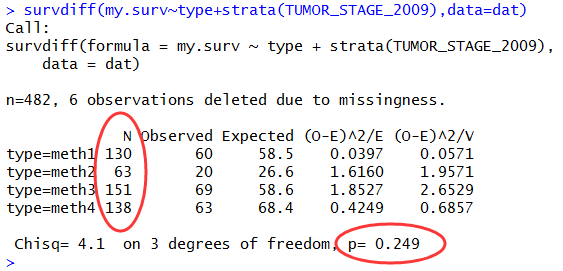
survdiff(my.surv~type+strata(TUMOR_STAGE_2009),data=dat)
##然后我们用这个代码来检验,是否显著,结果发现,p值并没有小于0.05,只能说是可以这样分组,但是分组的效果没有我们想象中的那么好!
names(dat)
detach(dat)
博主,想请教一下,TCGA生存数据里面的DFS该怎么取呢?只发现了最后联络时间和死亡时间,分别作为截尾时间和死亡事件处理的。不知道怎么取DFS时间呢~
TCGA提供的临床数据里面有DFS呀
我下的是biotab文件,只有:vital_status last_contact_days_to death_days_to和生存时间有关系,其他并没有找到的说,请问您一般用哪部分数据呢?