再次强调一下,R里面实现生存分析非常简单!
用my.surv <- surv(OS_MONTHS,OS_STATUS=='DECEASED')构建生存曲线。用kmfit2 <- survfit(my.surv~TUMOR_STAGE_2009)来做某一个因子的KM生存曲线。用 survdiff(my.surv~type, data=dat)来看看这个因子的不同水平是否有显著差异,其中默认用是的logrank test 方法。用coxph(Surv(time, status) ~ ph.ecog + tt(age), data=lung) 来检测自己感兴趣的因子是否受其它因子(age,gender等等)的影响。
我们还是拿TCGA的数据来做例子,卵巢癌的那篇文章里面根据甲基化数据和mRNA表达数据都可以把癌症样本分成四组,我们前面看了甲基化分组方法的确是有差异,但是还没有达到0.05的统计学意义的显著性。我们接下来把mRNA分组也检验一下,然后把两个分组方法当初一个影响生存率的因子,然后用cox方法看看这两个因子的权重!
Cox比例风险回归模型(Cox’s proportional hazards regression model),简称Cox回归模型。该模型由英国统计学家D.R.Cox于1972年提出,主要用于肿瘤和其它慢性病的预后分析,也可用于队列研究的病因探索。
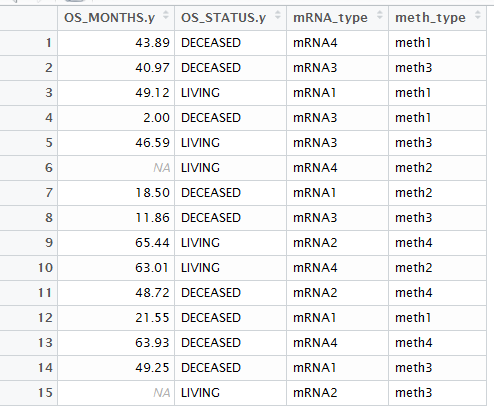
首先,我们从TCGA里面下载得到的做生存分析的数据如下(代码见末尾):
很明显看到,两种分组方式不一致的地方非常多,所以说癌症的分组仍然是一个没有解决的问题,我们可以把这两种分组方式当做因子来探究它们对生存率的影响!
my.surv=Surv(sur_dat$OS_MONTHS.y,sur_dat$OS_STATUS.y=='DECEASED')
#plot(survfit(my.surv~1))
survfit(my.surv~1)
kmfit=survfit(my.surv~1)
plot(kmfit)
summary(kmfit)
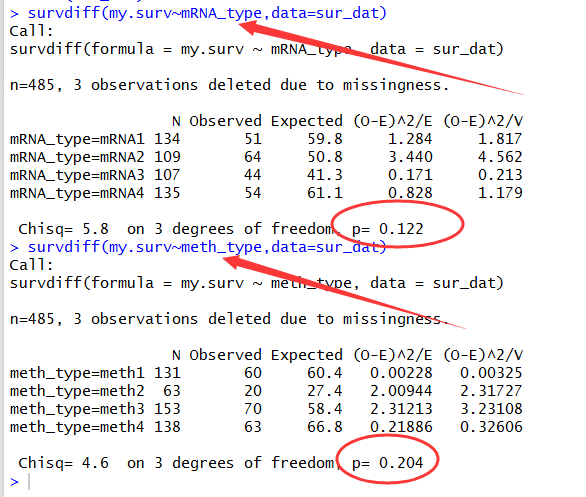
用survdiff检验分组的显著性,结果如下:
可以看出,这里两种分类方式都挺好的,虽然没有达到0.05的统计学意义上的显著。
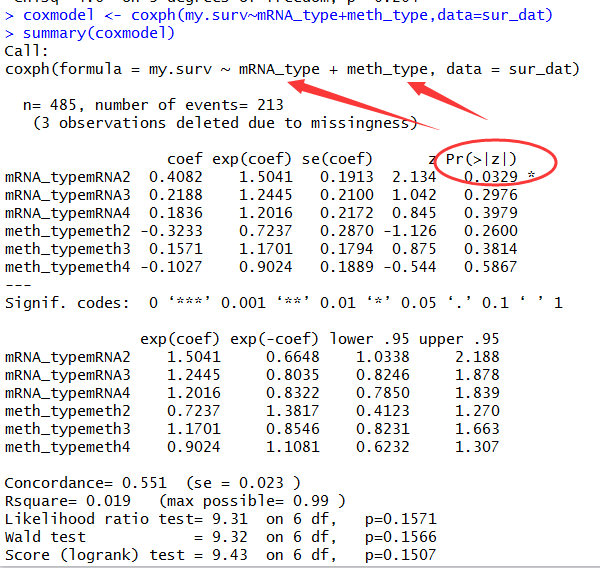
如果用cox模型回归分析如下:
所以R里面做生存分析是非常简单的,就寥寥几个函数即可,当然,你得知道这些函数的输入数据是什么。函数的结果很容易看懂,就是各种分组因子的显著性,还有风险比(HR)-hazard ratio(输出里面,coef就是beta值,相应的exp(coef)就是HR了) 而已,这也是大多数人关心的。
我记得用cox模型还可以得到预测函数,根据一些因子的值来预测病人还有多少天生存期