阅读文献并下载原始数据知illumina的Chip-seq数据
目录
一:阅读文献找到总实验项目
二:在根据实验项目地址找到所有实验数据的下载地址
三:构造脚本并下载
四:用sra-toolkit工具解压
正文
一:阅读文献找到总实验项目
该chip-seq数据其实隶属于一个大的实验项目组,其下载地址如下http://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE52964

二:在根据实验项目地址找到所有实验数据的下载地址
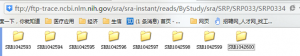
这里面的测序数据有八个,下载地址分别如下,都是单端50bp的测序策略
三:构造脚本并下载
用脚本 对它们进行批量下载,根据它们的命名方式 ,只需要构造普通的循环来下载
- for ((i=593;i<600;i++))
- do
- echo $i
- wget ftp://ftp-trace.ncbi.nlm.nih.gov/sra/sra-instant/reads/ByStudy/sra/SRP/SRP033/SRP033492/SRR1042$i/SRR1042$i.sra
- done
四:用sra-toolkit工具解压
我随便挑选其中一个下载给大家看看
http://www.ncbi.nlm.nih.gov/sra/SRX386762[accn]

SRX386762: GSM1278648: Xu_WT_rep2_Input; Homo sapiens; ChIP-Seq
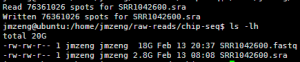
已经下载好了,2.7个GB的大小
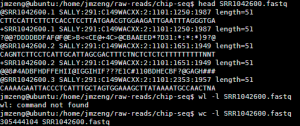
/home/jmzeng/bio-soft/sratoolkit.2.3.5-2-ubuntu64/bin/fastq-dump --split-3 SRR1042600.sra
把sra文件加压出原始reads,这个比较小,两分钟就搞定啦
解压后好像太大了一点,还是单端测序,共3亿的reads