发表这个算法的文章是:Clinical drug response can be predicted using baseline gene expression levels and in vitro drug sensitivity in cell lines 发表时间是:Genome Biology 2014 https://doi.org/10.1186/gb-2014-15-3-r47
了解 Cancer Genome Project (CGP) 数据库
主页: CGP website 是 Genomics of Drug Sensitivity of Cancer (GDSC)计划的数据
-
The CGP gene expression data are available from ArrayExpress under accession number E-MTAB-783.
-
The IC50 data for the drugs is available from the CGP website
Curve fitting code is now available as an R package – gdscIC50 – available on GitHub.
所有的关于这些癌症细胞系的各种数据,都可以在 ftp://ftp.sanger.ac.uk/pub4/cancerrxgene/releases 下载到。
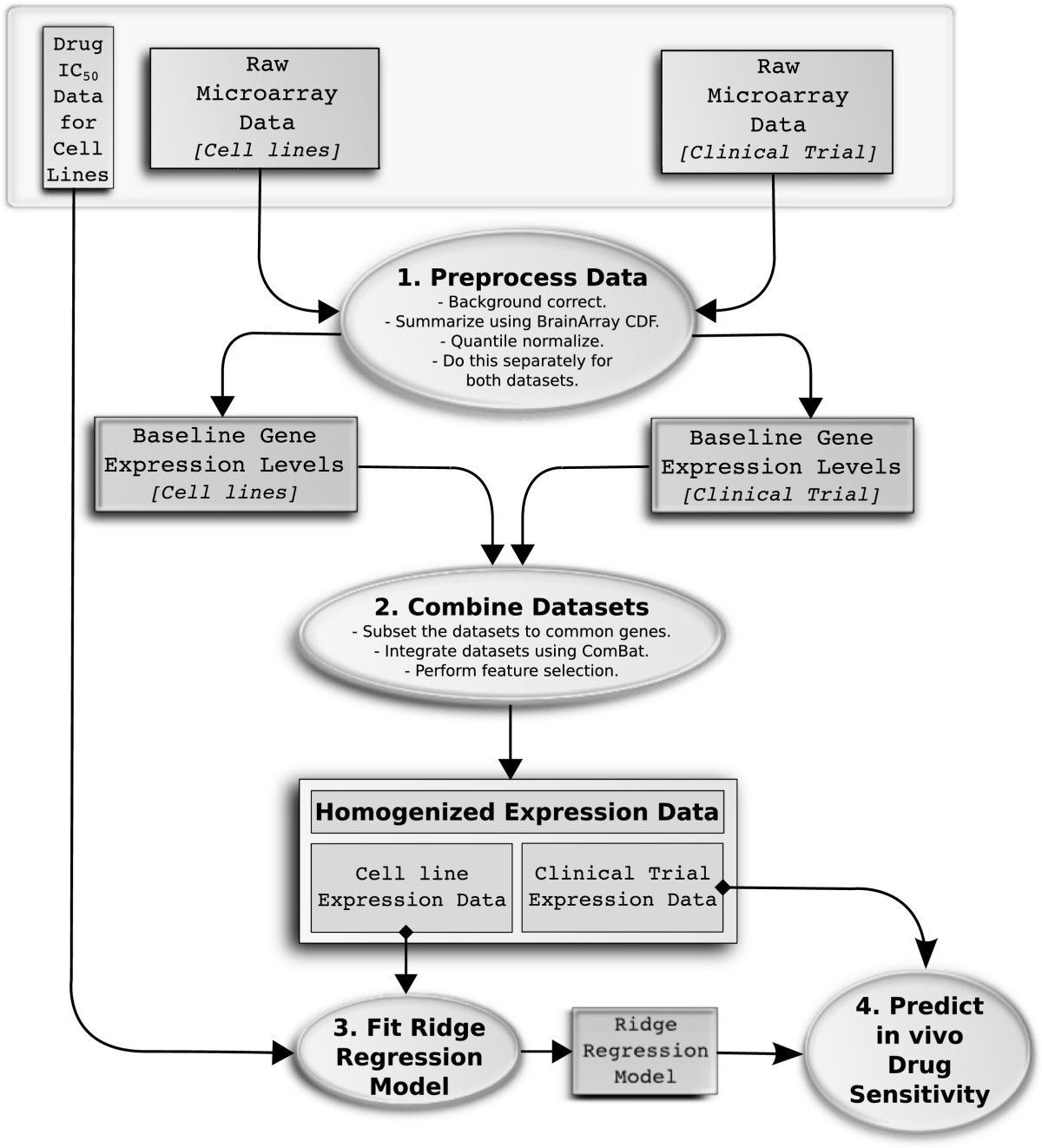
病人对药物的反应情况通常是一个很复杂的现象,由遗传因素和环境共同决定着。所以研究者通常认为我们要想预测药物作用就得收集尽可能的的信息,比如使用全基因组范围的snp信息来预测复杂性状,但是癌症患者有个特性,就是他们的染色体通常是非整倍体,所以从肿瘤样本里面测序得到可靠的基因型其实是比较困难的。相反,量化所有基因表达情况是很容易的事,问题在于,通过芯片来量化基因表达情况的重复性不太好,尤其是不同公司的芯片。
作者选取了Cancer Genome Project (CGP) 数据库里面收录的 700多种细胞系的 138 种药物的作用情况,开发了算法,而且在 4 个符合要求的数据集里面验证了可靠性。
第一步,把两个表达矩阵合并,就是Training (cell lines) and test (clinical trial) datasets ,通过sva包的ComBat()函数,去除低表达量基因以及低变化量基因。
第二步,使用 ridge包的linearRidge()函数做岭回归分析,其中药物敏感性的IC50值需要用car包的powerTransform函数进行转换,根据训练集的数据把模型构建成功就可以使用 predict.linearRidge() 来预测测试集的病人的药物反应情况了。
第三步,留一交叉验证,每次假装不知道一个细胞系的药物反应情况,用其它的所有的细胞系数据来预测它。最后把预测值和真实值做相关性分析。
第四步,使用glmnet包做ElasticNet and Lasso 回归
第五步,药物敏感性分成sensitive (15 samples) or resistant (55 samples) 两个组别,做 logistic ridge 回归。
作者的数据处理代码
代码见:http://genemed.uchicago.edu/~pgeeleher/cgpPrediction/
wget http://genemed.uchicago.edu/~pgeeleher/cgpPrediction/paper.zip
## (161M)
unzip paper.zip
cd paper/docetaxelAnalysis/
R
Sweave("docetaxelBreastCancer.Snw")
上面的代码就会生成信息的文档,描述作者的数据处理步骤。
首先需要R, RStudio, Latex相应组件,RStudio里的kintr包。 File-> New File -> R Sweave 里创建rnw文件
在4个公共数据里面验证
4个有着明确药物处理记录的数据;
-
The docetaxel data are available from GEO under accession numbers [GEO:GSE349] and [GEO:GSE350].
-
The cisplatin data are available from ArrayExpress under accession number E-GEOD-18864.
-
The bortezomib data are available from GEO under accession number [GEO:GSE9782].
-
The erlotinib data are available from GEO under accession number [GEO:GSE33072].