物种:cynomolgus monkey (Macaca fascicularis)
新闻都很劲爆,比如:
- 祝贺!中国团队成果登Nature,颠覆“长寿基因”认知
- Nature:中国科学家颠覆“长寿基因”认知
- NATURE重大突破:世界首例长寿基因编辑猴模型在中科院诞生
也许我的生物学背景不够,所以理解不了这个突破,在我看来就是一个基因编辑而已,如下:
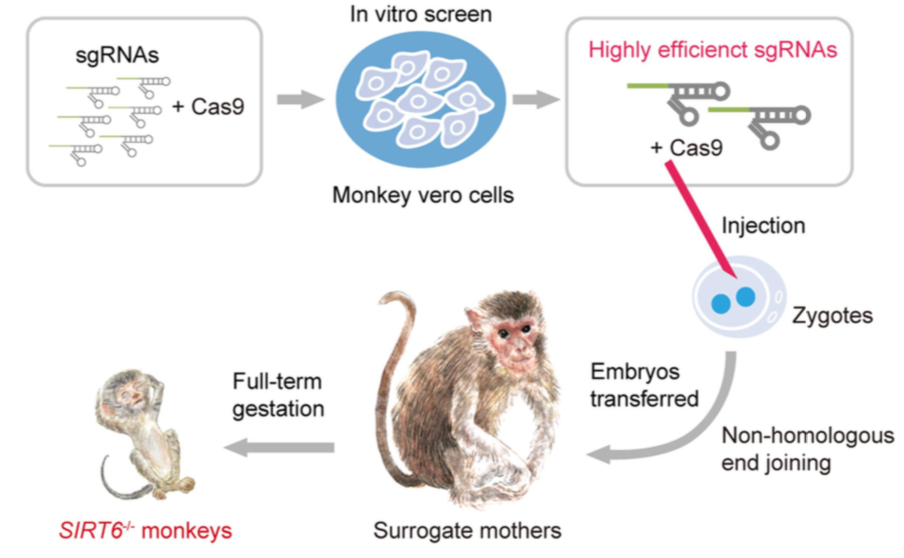
看起来就是他们团队使用CRISPR-Cas9技术创造了一个雄性猴子胚胎和三个雌性猴子胚胎,它们均不表达SIRT6。结果发现,雌性猴子在出生后不久就死亡了,而雄性胚胎在怀孕中期死亡。然后就使用转录组测序手段探索了基因编辑的猴子的表达变化情况。数据下载
总共测序得到
241.16 G数据,表达矩阵在 GSE102830, WGS验证crispr的原始测序数据在 SRP149748
值得注意的是作者同时把数据上传到了北京基因组所: (http://gsa.big.ac.cn/) under accession number PRJCA000909.
参考基因组用的是: M. fascicularis reference genome (macFas5)
作者公布的表达矩阵是以FPKM作为归一化手段,可能需要重新处理该数据,如果想走自己的下游分析。
| Supplementary file | Size | Download | File type/resource |
| —————————————— | ———— | —————————————————————————————— | ——————————— |
| GSE102830_FPKM_2_4M.txt.gz | 750.3 Kb | (ftp)(http) | TXT |
| GSE102830_FPKM_tissue.txt.gz | 4.3 Mb | (ftp)(http) | TXT |
涉及到7个器官,总共是6只 食蟹猴,分别是:organs brain heart kidney liver lung muscle thymus KO2 2 2 2 2 2 2 2 KO3 2 2 2 2 2 2 2 T2M 2 0 0 0 0 2 0 T4M 2 0 0 0 0 2 0 WT2 2 2 2 2 2 2 2 WT3 2 2 2 2 2 2 2其实还有全基因组甲基化数据,不过作者并没有上传该数据,或者我没有查找仔细。
CRISPR–Cas9 脱靶效应分析
使用Cas-OFFinder软件, 预测可能的脱靶位点。3,891 possible off-target regions were identified, including 983 sites without NAG or NGG PAM sequences.
所有关于全基因组的数据分析,都只是为了在正文提到:Whole-genome DNA sequencing further confirmed the precise targeting of the SIRT6 locus in all three monkeys, without detectable off-target effects across the genome,也就是说没有明显的脱靶效应,IGV截图基因编辑效果如下:
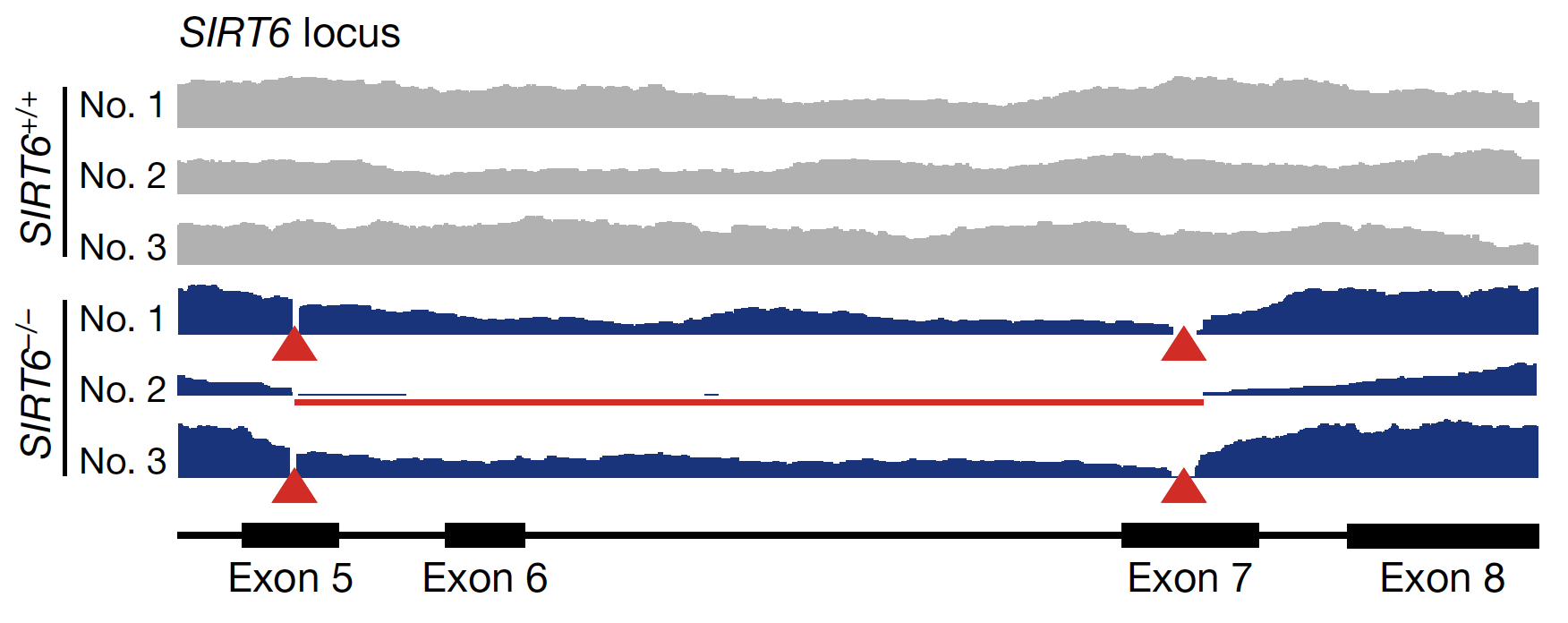
同时人工检测部分预测脱靶位点并且截图展示。

再整理表格说明那些全部的(3,891个)位点情况;
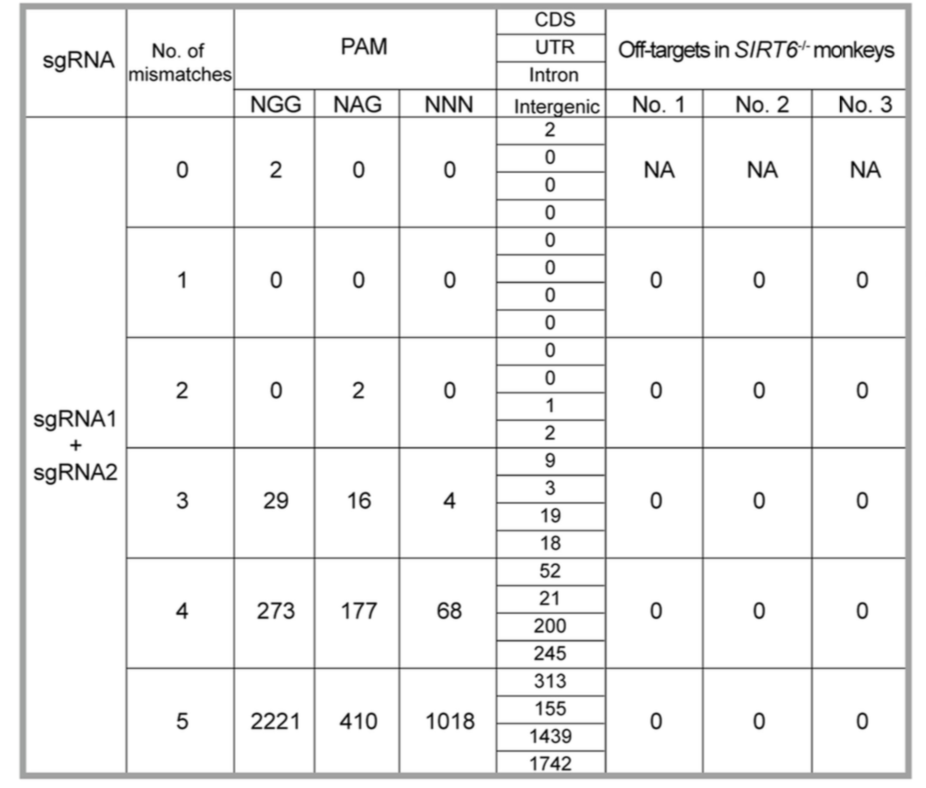
查看crispr是否明显造成indel
因为物种是monkey,所以作者没有走标准的人类相关indel分析流程,也没有使用成熟的indel相关软件,结构变异更没有触及。
从文中方法描述来看,找indel的方法太依赖主观经验,几乎不可能重复出来这个分析流程:

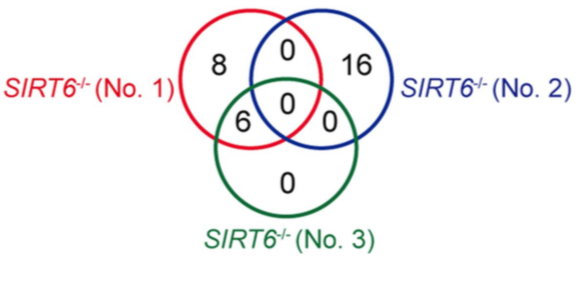
作者使用这样的in house 流程,分析得到的indel结果如下:
转录组数据看看差异表达
这个分析倒是中规中矩,如前面提到了,作者涉及到的表达器官有点多,而主要分析要点是sirt6敲掉后造成的全身基因表达变化,而不是器官特异性的,所以作者做了大量的两两相比的差异分析得到了大量的显著差异基因列表,然后取交集来缩小范围。
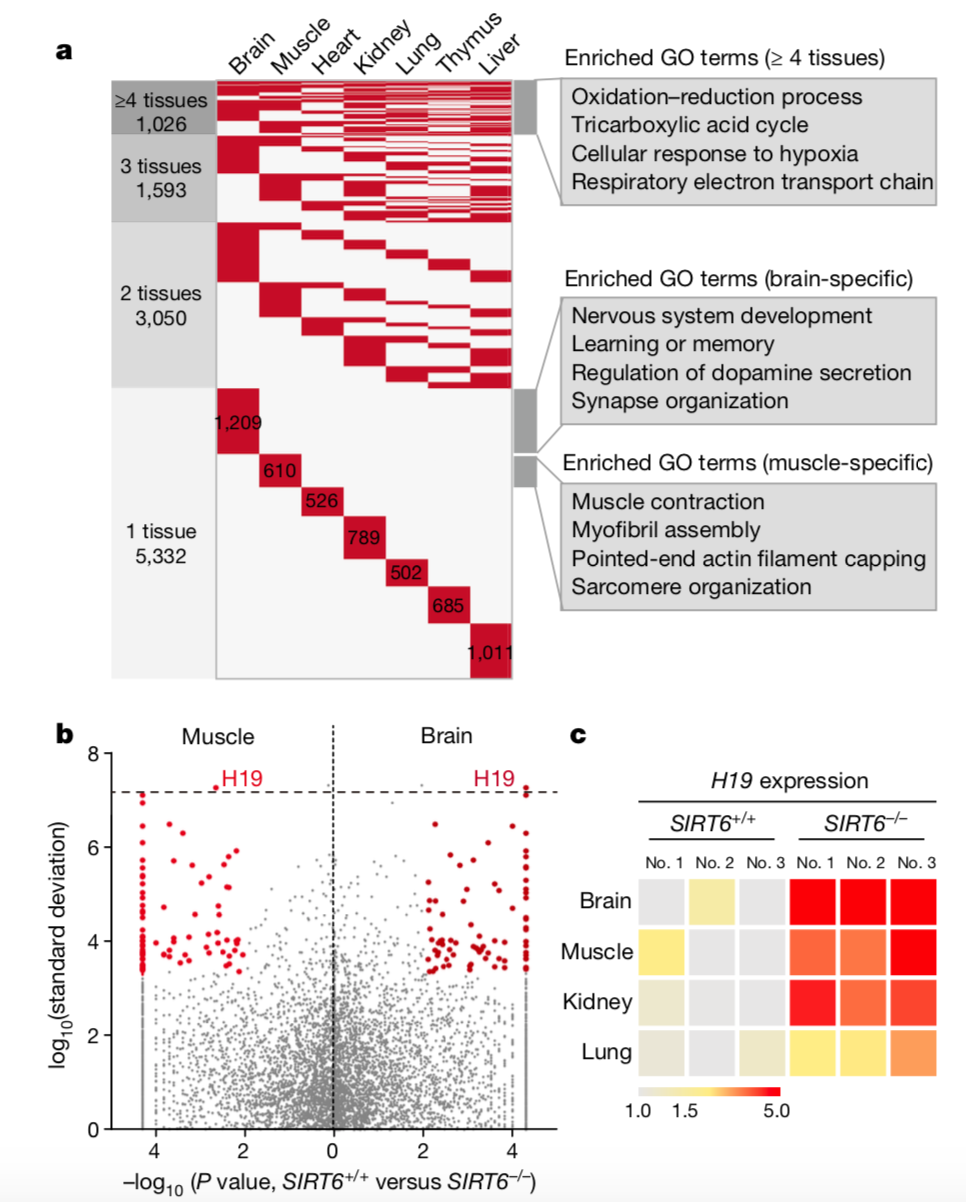
值得注意的是,作者简单粗暴的选择了表达变化最大的那个长非编码基因H19作为下游实验验证的靶标。
看完,还是不明白这个新闻劲爆在哪里。