你现在看到的是文献俱乐部2019年笔记
大名鼎鼎的TCGA计划回顾一下咯,关于AML研究发表在 N Engl J Med. 2013 May , 算是很厉害了,附件一百多页的PDF详尽的描述了当时的数据分析方法,而且这个研究非常受重视,仅仅是关于 它的评论就有:
- The beginning of the end of the beginning in cancer genomics. [N Engl J Med. 2013]
- Genetic and epigenetic map of acute myeloid leukemia. [Pharmacogenomics. 2013]
- Cancer: Mutations close in on gene regulation. [Nature. 2013]
- A panoramic view of acute myeloid leukemia. [Nat Genet. 2013]
- Genetics: the AML mutational landscape. [Nat Rev Clin Oncol. 2013]
- Genomic landscapes and clonality of de novo AML. [N Engl J Med. 2013]
- Genomic landscapes and clonality of de novo AML. [N Engl J Med. 2013]
纳入了 200 个clinically annotated adult cases of de novo AML - 其中50人测WGS, 150人测WES
- 同时伴有 RNA and microRNA sequencing and DNA-methylation 数据
这些数据都可以在TCGA官网下载了 https://portal.gdc.cancer.gov/projects/TCGA-LAML :
| Experimental Strategy | Cases | ( n=200 ) | Files | ( n=4,434 ) |
| ——————————- | —————————————————————————————— | —————————————————————————————— | —————————————————————————————— | —————————————————————————————— |
| WXS | 149 | | 1,785 | |
| RNA-Seq | 151 | | 604 | |
| miRNA-Seq | 188 | | 564 | |
| Genotyping Array | 200 | | 795 | |
| Methylation Array | 140 | | 280 | |
另外一种展现方式:https://tcga-data.nci.nih.gov/docs/publications/laml_2012/急性骨髓性白血病(Acute myeloid leukemia,AML)
急性髓系白血病(AML)是最常见的髓系血液肿瘤,也是成人白血病中最常见的类型,大约占所有白血病的50-60%,我国每年新发AML大约有30,000例。
急性白血病:是一类造血干祖细胞来源的恶性克隆性血液系统疾病。临床以感染、出血、贫血和髓外组织器官浸润为主要表现,病情进展迅速,自然病程仅有数周至数月。一般可根据白血病细胞系列归属分为急性髓系白血病(AML)和急性淋巴细胞白血病(ALL)两大类。
其中AML还可以进行FAB分型,最初的诊断是完全基于形态学(Morphology)的,即1976年法英美科学家制定FAB标准 - AML-M1 Acute myeloblastic leukemia without maturation
- AML-M2 Acute myeloblastic leukemia with differentiation
- AML-M3 Acute promyelocytic leukemia
- AML-M4 Acute myelomonocytic leukemia
- AML-M5 Acute monocytic leukemia
- AML-M6 Erythroleukemia
- AML-M7 Acute megakaryocytic leukemia
- AML-M0 Acute myeloblastic leukemia, minimally differentiated
而与之对应的慢性白血病常见有慢性粒细胞性白血病(CML)、慢性淋巴细胞性白血病(CLL)。
Hartmut Dohner医生2015年9月在新英格兰医学杂志发表的一篇AML综述,详细介绍了AML的功能性基因改变,值得相关方向研究者研读。主要发现
显著突变基因
首先必须秀出来的当然是somatic mutation的基因咯,毕竟在那个年代,人们对AML的热点突变所知甚少,任何突变基因的更新,都是一个进步,尤其是这样的人群大队列,一下子释放出来这么多突变基因,如下,有23个基因显著性的突变,还有237个基因不止一个样本突变。
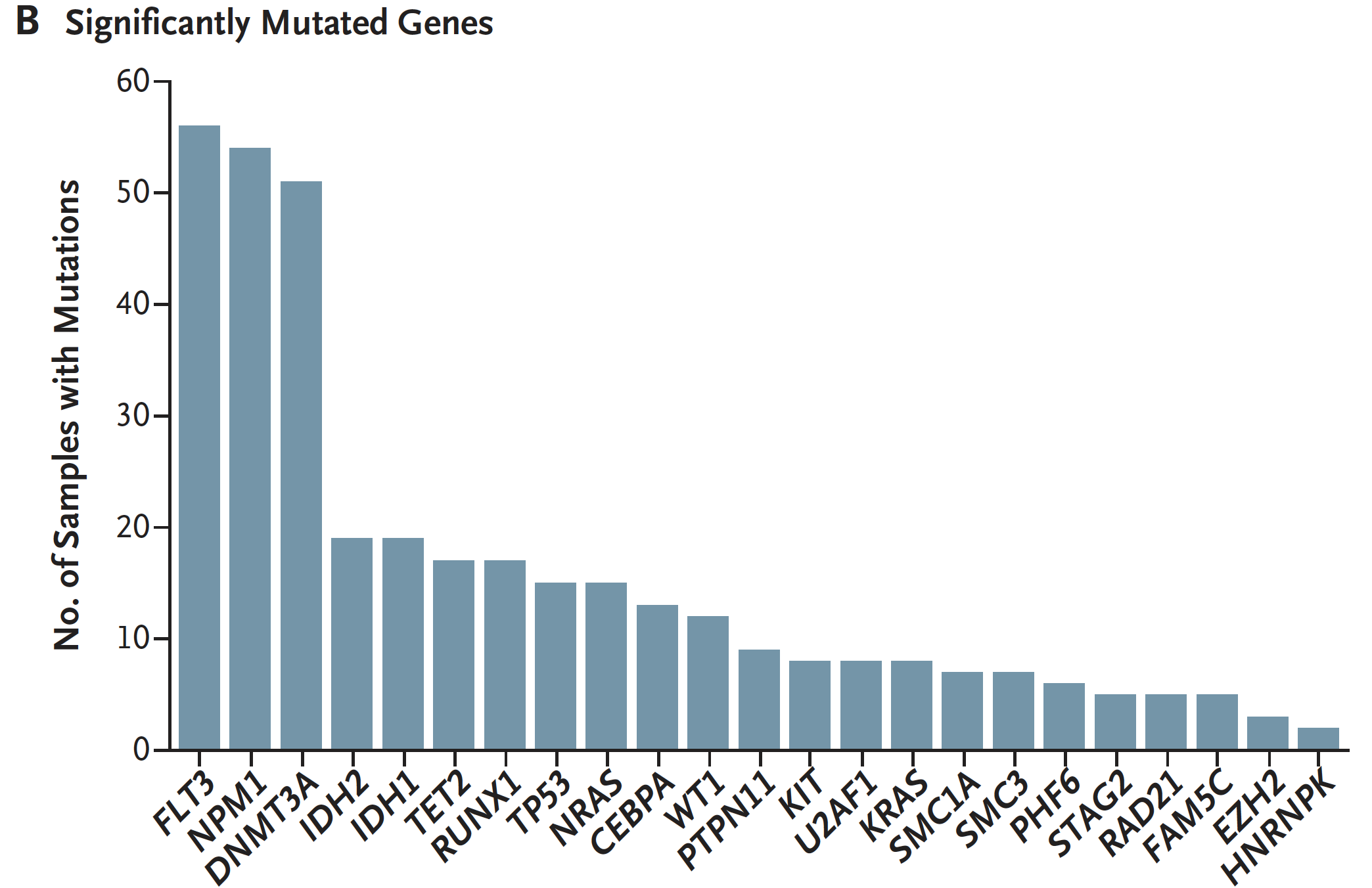
作者发表这个数据集的那个年代,确定某个基因是否显著突变,用的是 MuSiC 软件,现如今这样的工具算法已经超10个了,大家可以下载突变信息,走一波新的工具,也可以结合2016年sanger研究所的一千多AML病人的突变信息一起来找显著突变基因。
研究者感觉这些基因的生物学功能,尤其是致病性,着重点出来了9类: - transcription-factor fusions (18% of cases)
- the gene encoding nucleophosmin (NPM1) (27%)
- tumor-suppressor genes (16%)
- DNA-methylation-related genes (44%)
- signaling genes (59%)
- chromatin-modifying genes (30%)
- myeloid transcription-factor genes (22%)
- cohesin-complex genes (13%)
- spliceosome-complex genes (14%).
突变全景图
现在做这样的图已经非常容易了,标准的GATK流程,找到可靠的somatic突变,然后maftools进行可视化即可。
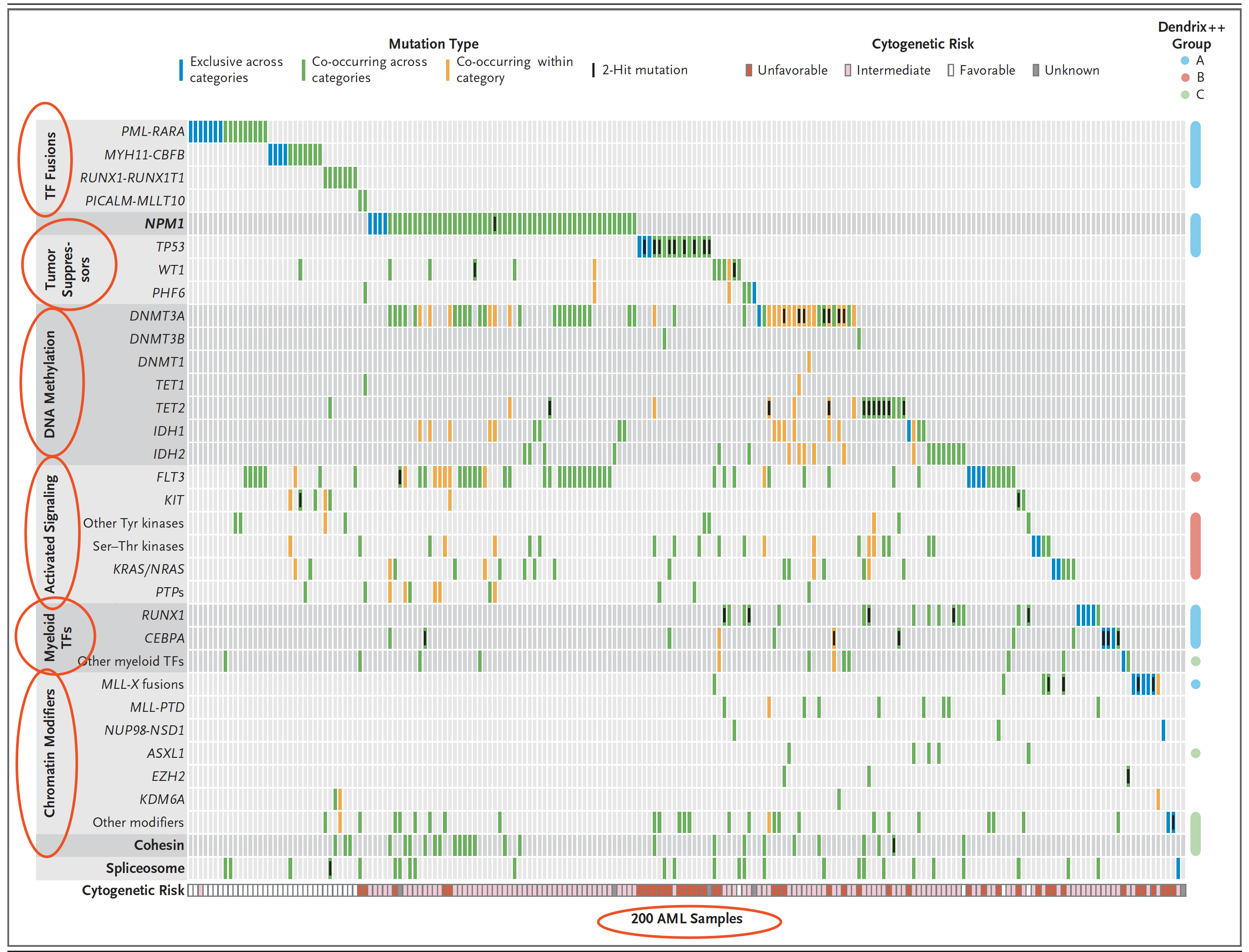
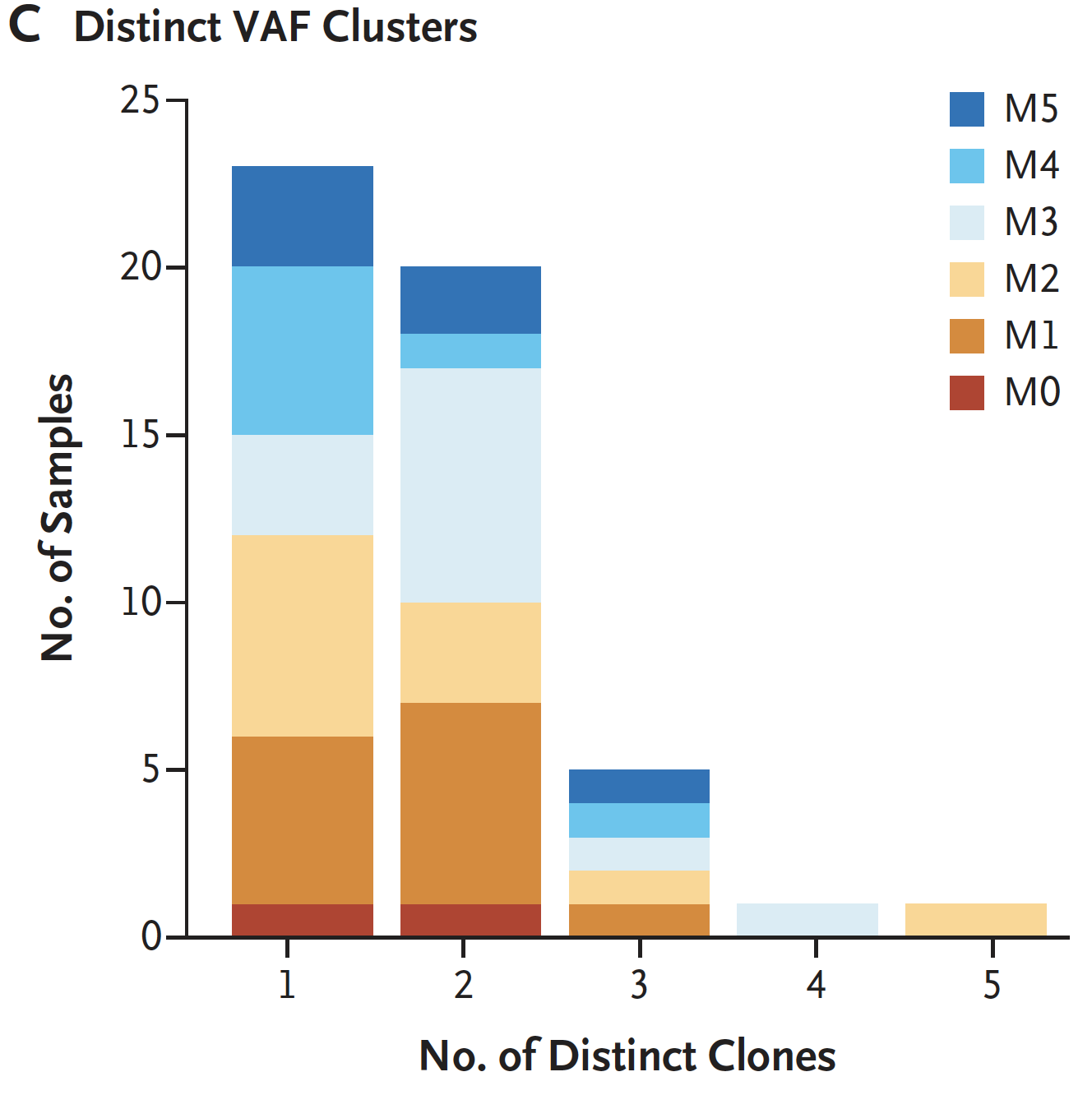
根据每个病人的somatic突变的所有VAF值的可以判断该病人有多少个肿瘤克隆,因为AML病人的突变数量不多,所以很难检测到太多克隆,大部分就一两个克隆而已,还可以根据一些临床信息进行分类,比如下图的法英美科学家制定FAB分类。
表达量分类
前面提到过除了WGS和WES这样的DNA测序外,还有:
- RNA-expression profiling on the Affymetrix U133 Plus 2 platform for 197 samples
- RNA sequencing for 179 samples
- microRNA (miRNA) sequencing for 194 samples
- Illumina Infinium HumanMethylation450 BeadChip profiling for 192 samples
- Affymetrix SNP Array 6.0 for both tumor and normal skin samples from all 200 patients.
既然有表达矩阵,就可以进行分类,这个是当年的常规分析了,这里使用的是 ConsensusClusterPlus (Tutorial) - Bioconductor 包,使用的是NMF算法。
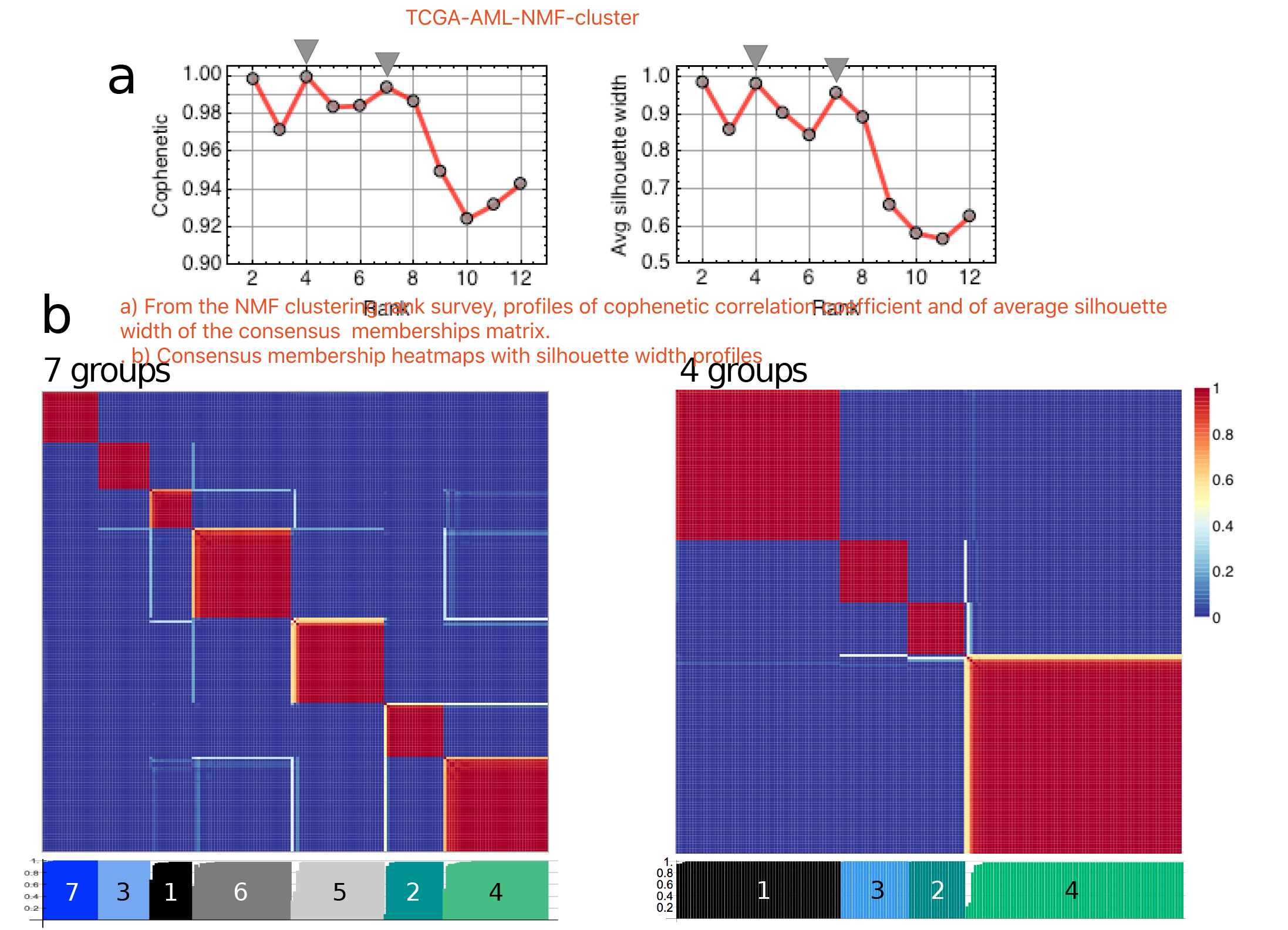
可以看到200个AML病人的bulk转录组测序可以稳定的分成7组,研究人员也在附件耗费大量笔墨描写了各个分组的生物学意义。
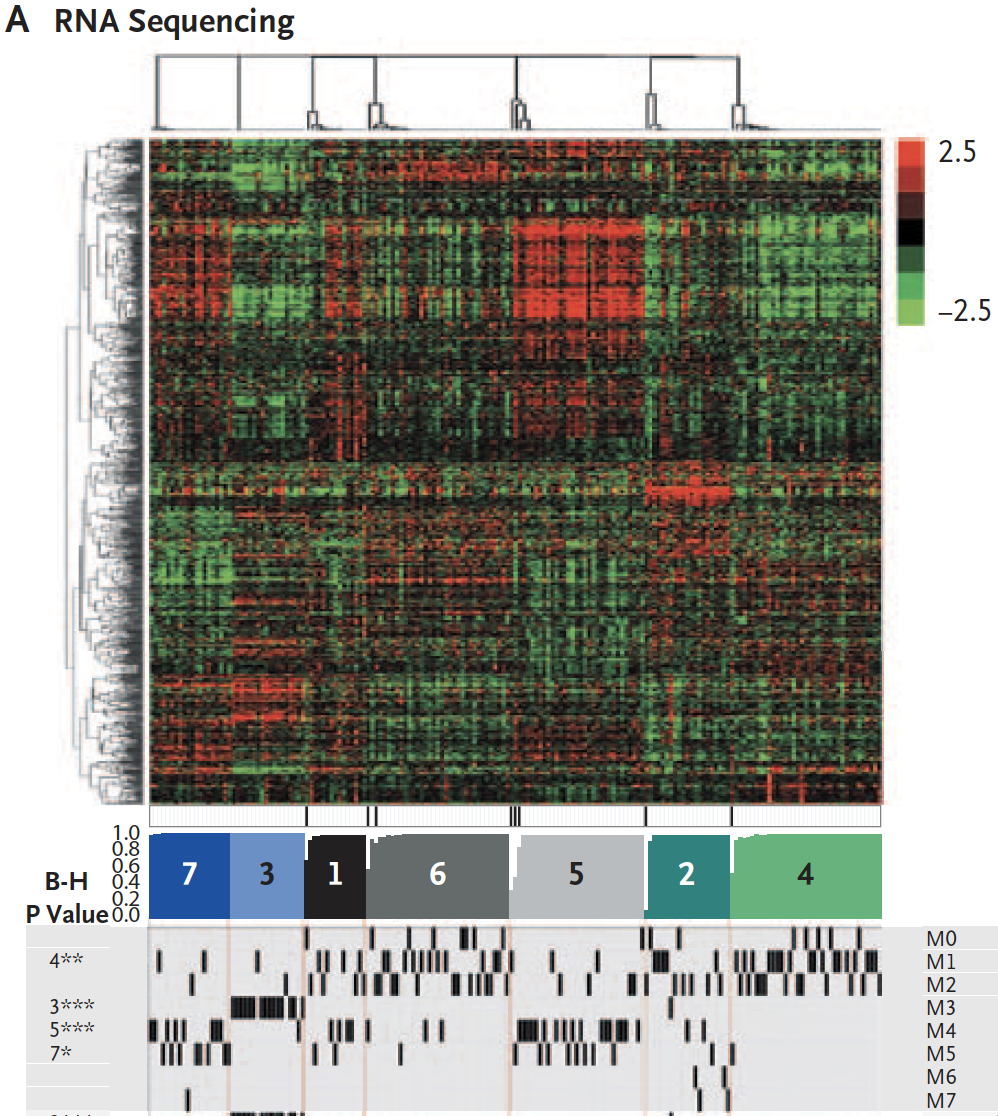
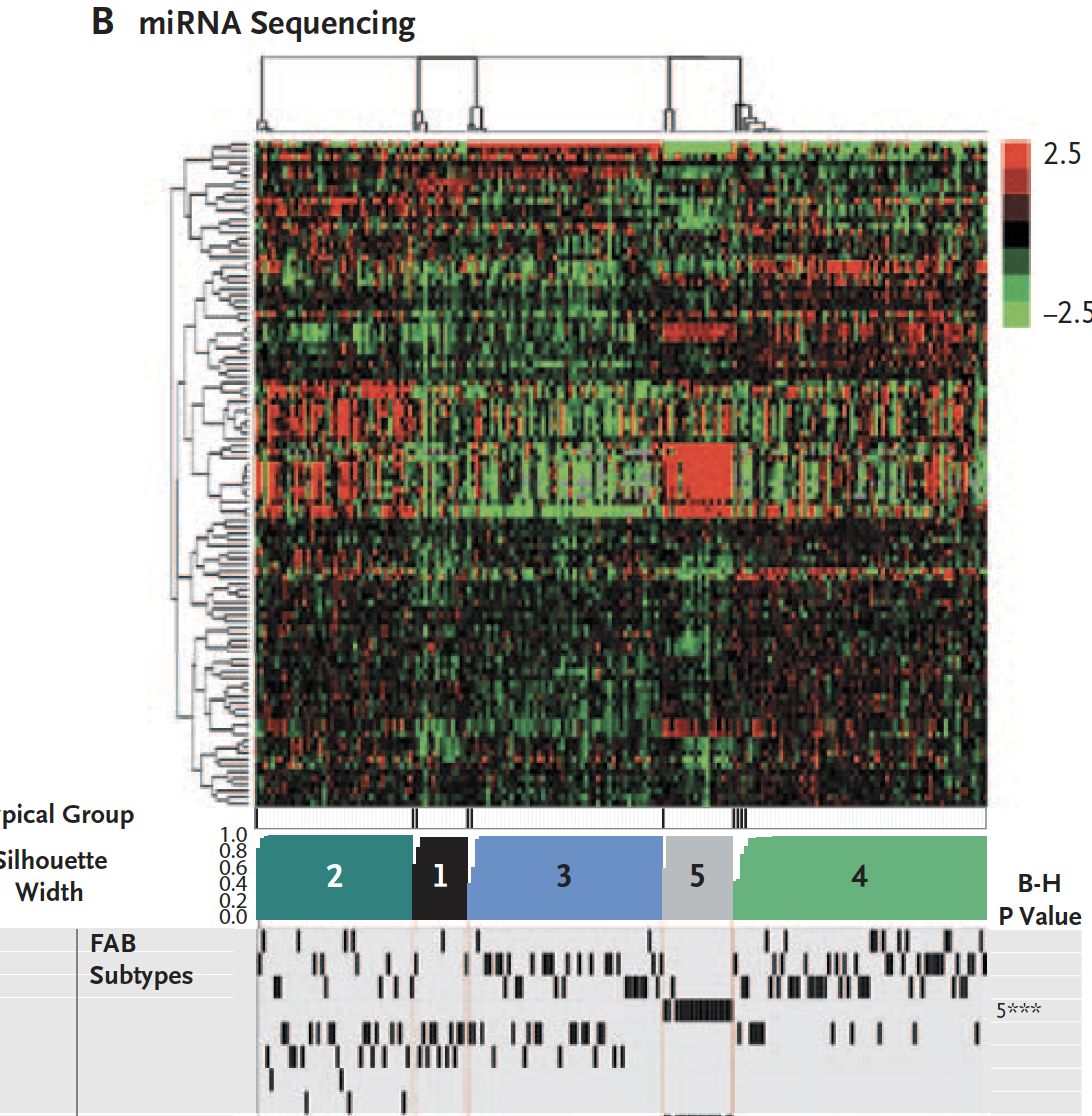
可以看到200个AML病人的bulk miRNA-seq测序可以稳定的分成5组,研究人员也在附件耗费大量笔墨描写了各个分组的生物学意义。
甲基化信号值分类
CpG island 是表观遗传学领域很重要的概念,通常全基因组可以分成gene promoters associated with dense-CGIs, sparse-CGIs and non-CGIs ,也就是说致密的CpG区域的甲基化情况和稀疏的CpG甲基化情况的生物学意义不一样,所以需要分开进行聚类。
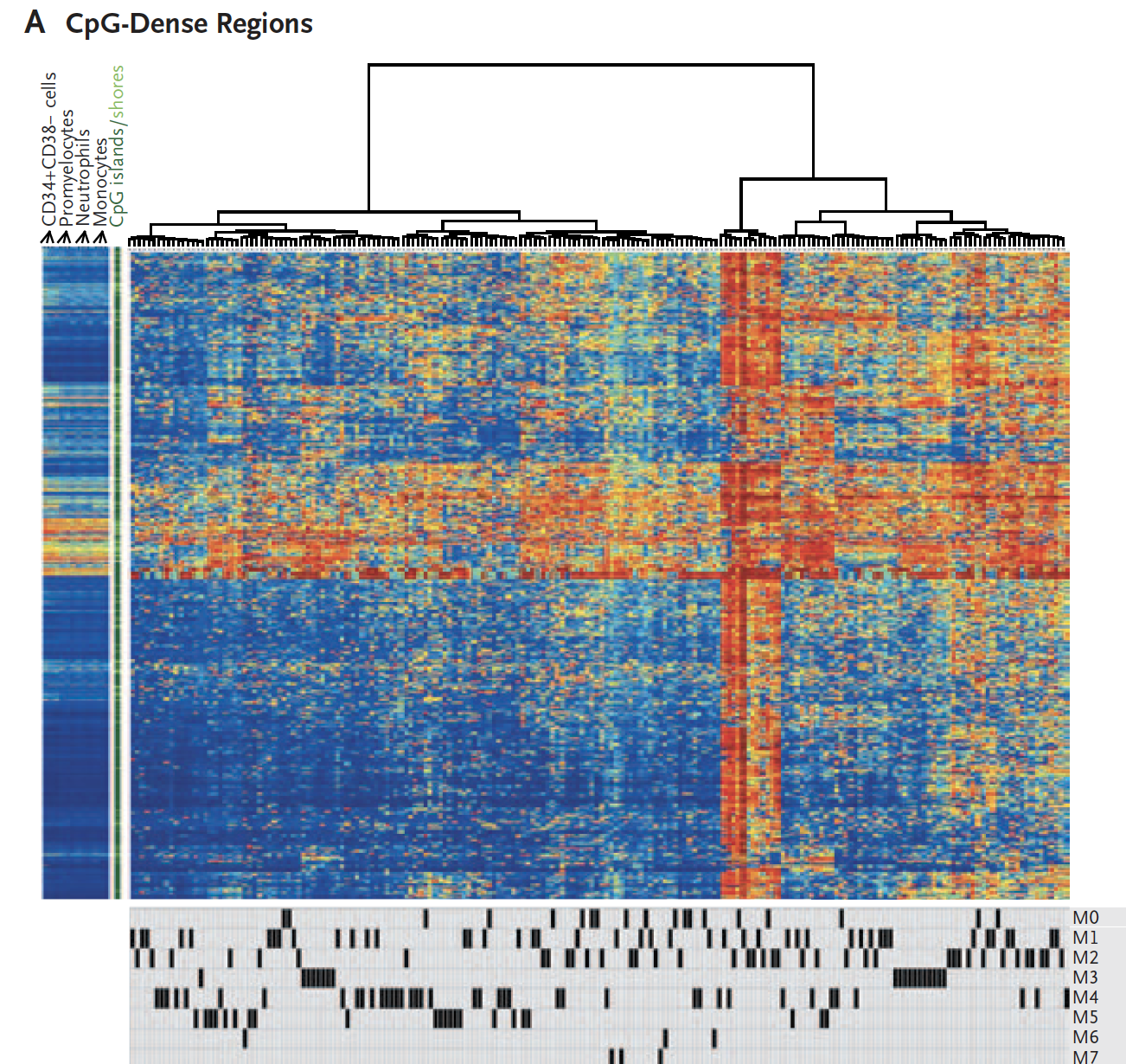
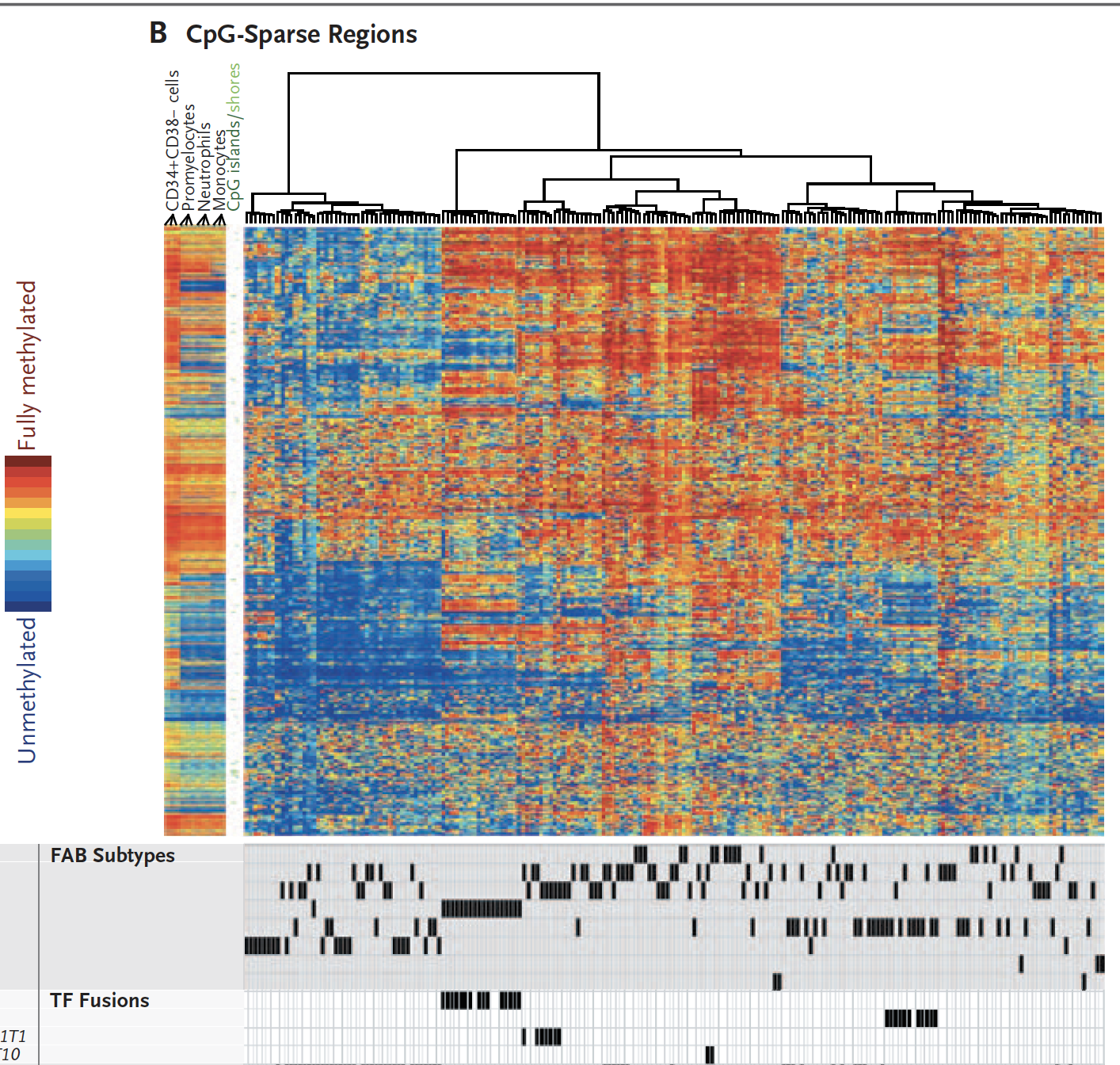
可以在TCGA数据库下载AML数据集的甲基化芯片信号值矩阵重复这篇文献的研究。
DNA methylation - Illumina Human Methylation 27 (n=140) GDC Hub
- Illumina Human Methylation 450 (n=140) GDC Hub
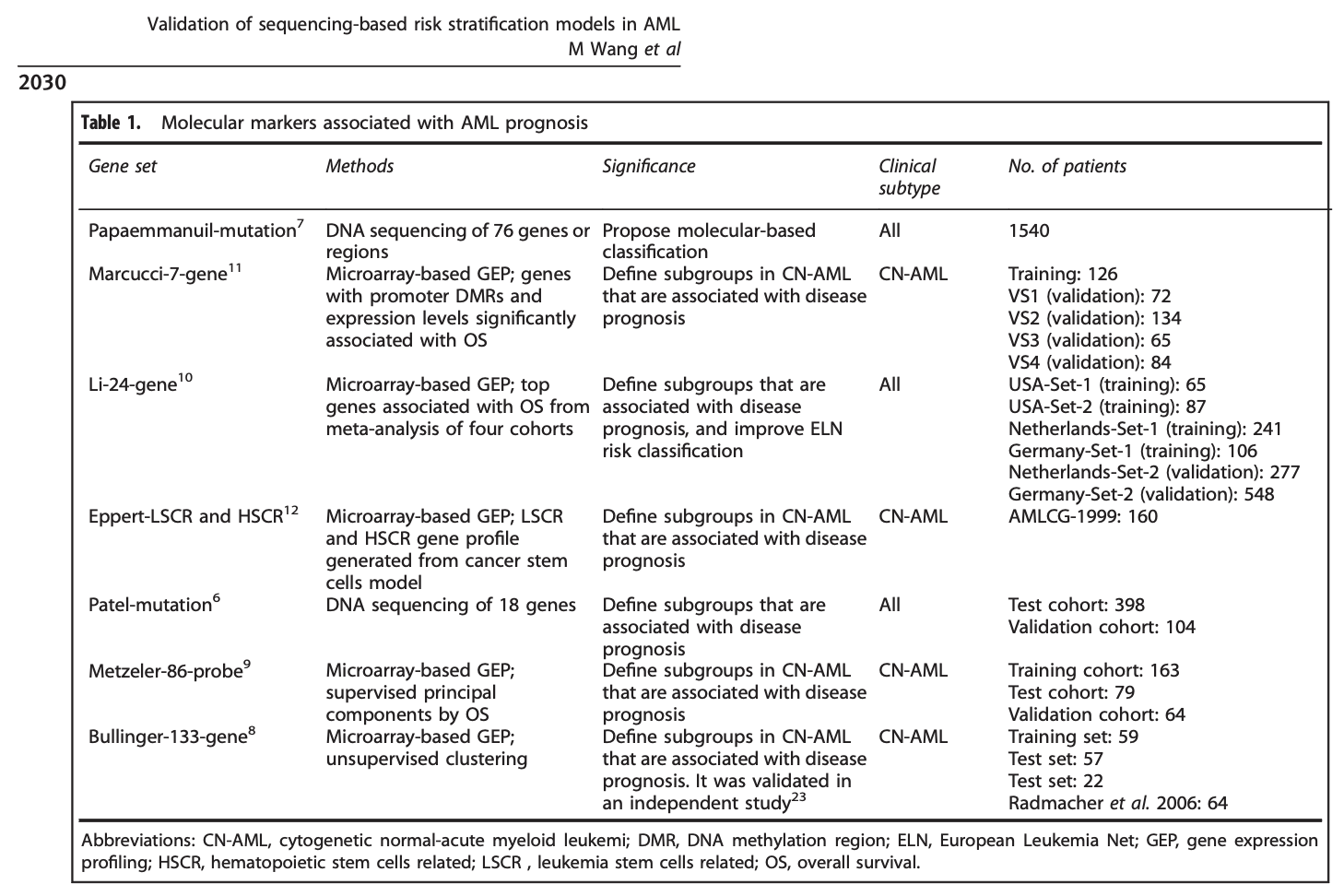
AML的一些分子标记物研究
Leukemia (2017) 的文章Validation of risk stratification models in acute myeloid leukemia using sequencing-based molecular profiling 进行了总结,如下:
其它公共数据库
另外一个大队列是桑格研究所对1500多名急性骨髓性白血病(AML)患者进行测序,检测其癌症基因,最终鉴定出11种基因亚型,也是发表在《New England Journal ofMedicine》杂志上 Genomic Classification and Prognosis in Acute Myeloid Leukemia
还值得一提的是:347 AML patients recruited in the National Taiwan University Hospital (NTUH) ,虽然表达矩阵并没有发出来。
当然还有GEO数据集:various lineages of normal hematopoiesis and AML marrow (GSE12662, GSE24006, and GSE24759) GSE12417 等
还有2018年的 Clinseq-AML cohort, consist of 274 AML patients, treated according to the national guidelines in Sweden,好像也没有看到表达矩阵。公共数据库挖掘文献举例
- J Cell Biochem. 2019 Mar; 是 A 4-microRNA signature(IF不到3分)
- Leukemia. 2015 May;(IF超过10分)
拷贝数变异结果的解释
来源于 xseq包的cna.call数据集,是 TCGA AML SNP6.0 GISTIC copy number alteration calls
A matrix containing the GISTIC copy number calls of 454 genes in 197 patients: - Row names are patient identifiers
- Colume names are official HGNC gene symbols
Each element of the matrix is coded: - -2, homozygous deletions
- -1, hemizygous deletions
- 0, neutral
- 1, gain
- 2, amplifications
其它相关癌症研究
在发表AML数据集之前已经有了几个其它TCGA癌症研究结果发表:
- glioblastoma multiforme (http://cancergenome.nih.gov/newsevents/newsannouncements/news_9_4_2008)
- ovarian serous adenocarcinoma (http://cancergenome.nih.gov/newsevents/newsannouncements/ovarianpaper)
- colorectal adenocarcinoma (http://www.cancer.gov/news-events/press-releases/2012/TCGAcolorectal)
- lung squamous cell carcinoma (http://www.cancer.gov/news-events/press-releases/2012/lungsquamoustcga)
- invasive breast cancer (http://cancergenome.nih.gov/newsevents/newsannouncements/breastserovca)