为了分析不同类型、组织起源肿瘤的共性、差异以及新课题。TCGA于2012年10月26日-27日在圣克鲁兹,加州举行的会议中发起了泛癌计划。参考:https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6000284/ 为此我也录制了系列视频教程在:TCGA知识图谱视频教程(B站和YouTube直达)
发表于 Nat Genet. 2014 Nov, 文章是 Genome-wide analysis of noncoding regulatory mutations in cancer. 本研究专注于分析那些有全基因组测序的肿瘤样本,不到一千个。采取3个研究策略:
- hotspot analysis focused on small regions that frequently contained mutations;
- regional recurrence analysis identified annotated regions that contained numerous mutations;
- transcription factor analysis nominated regions with ETS transcription factor binding sites that were disrupted or created by mutation.
大多数的研究都集中在肿瘤用来产生蛋白的基因组部分,即蛋白编码基因。蛋白就像细胞机器一样发挥作用。当它们正常地运作时,蛋白执行良好的健康所需的细胞任务。但是当蛋白不能正常地工作时,包括癌症在内的疾病可能就会产生。然而,表达蛋白的基因仅占整个基因组的1%至2%,更多未知的信息都存储在剩余的98%基因组区域,只有真正的全基因组测序数据才能探索到。
文献解读属于100篇泛癌研究文献系列,首发于:http://www.bio-info-trainee.com/4132.html病人数量及癌症肿瘤
分布如下:
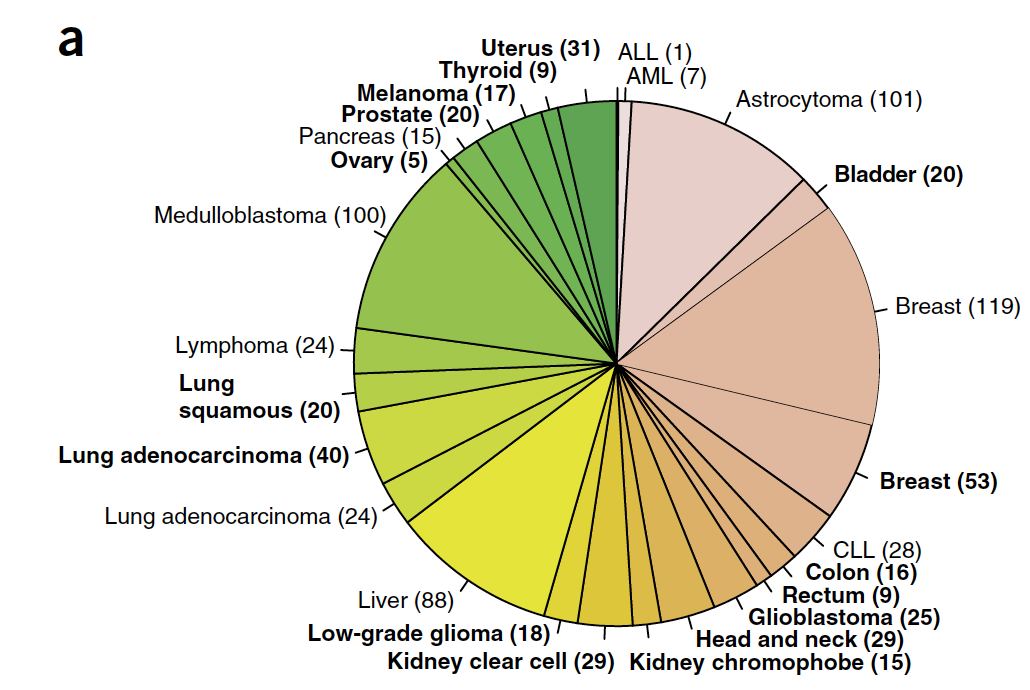
ROIs的突变数量分布
关于genomic regions of interest (ROIs)
包括:
- 经典生物学包括: gene,intron,exon,CDS,UTR
- 表观生物学包括: CpG,promoter,enhancer
- 其它: insertions/deletions, copy number variants, differentially methylated regions, transcription factor binding sites (TFBSs), and 3D interacting regions
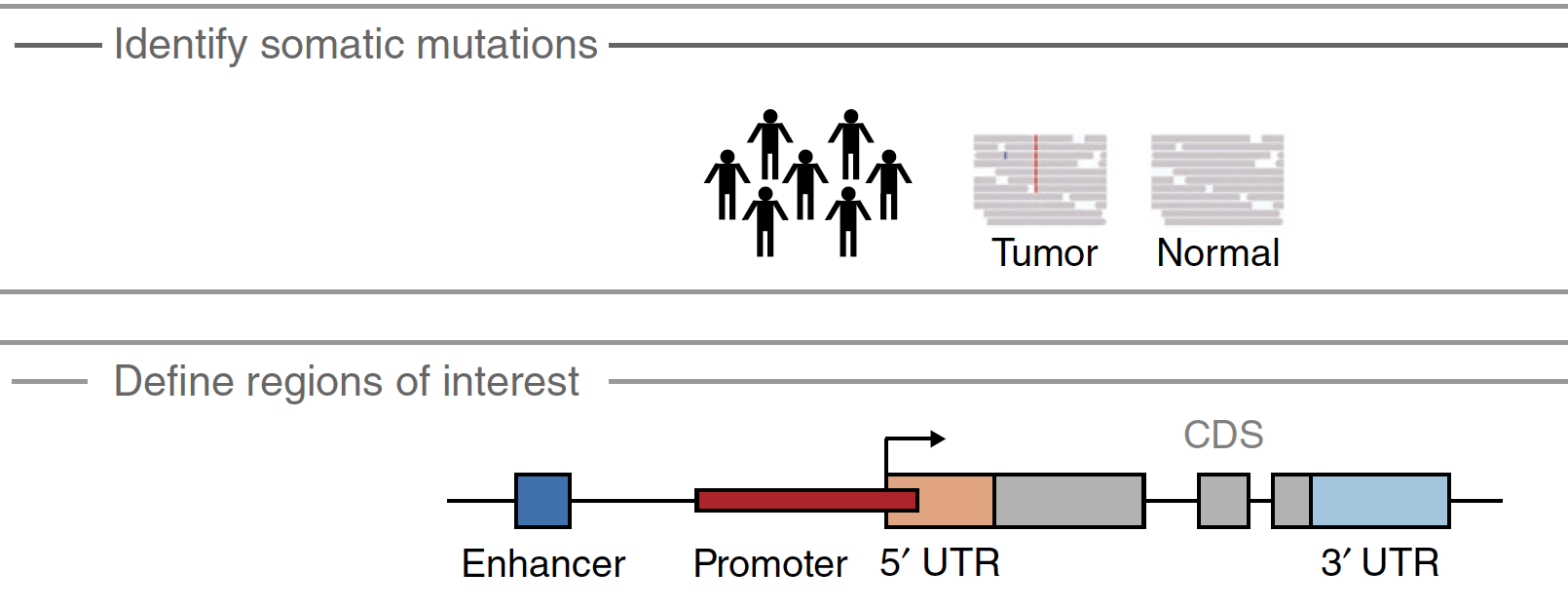
国际著名的 International Human Epigenome Consortium (IHEC) 包括:ENCODE, NIH Roadmap Epigenomics, FANTOM5, BLUEPRINT 通常喜欢把ROIs链接到邻近基因来代表它的功能,基于一个很简单的理论,这个ROIs在基因附近,那么它自然而然的可以影响该基因的行为。熟悉命令行工具的研究者可以用bedtools等工具,不熟悉的也有ColoWeb这样的网页工具。
本文采用Ensembl v70 数据库定义CDS,UTR区域,然后启动子区域是TSS的上游2Kb和下游200bp,还有 66 944 enhancer-region 也来源于数据库。
如下:
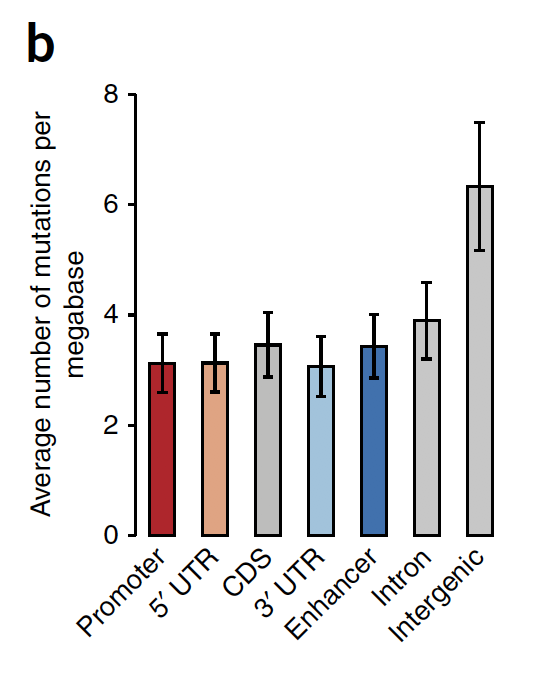
Transcription factor analysis.
在ENCODE数据可以得到 3个EST酶( EHF, ELF1 and ETS1) 都是可以结合 SDHD 启动子区域的, 所以SDHD 启动子区域突变会影响它被ELF1的调控。
SDH是一个四聚体酶(4个亚基分别由SDHA, SDHB, SDHC, SDHD基因编码) ,本文重点描述的是SDHD基因,它的启动子区域的突变可以把melanoma 分组,mutations (n = 13; red) 和 wild-type (WT) SDHD (n = 42; blue) ,表达量差异很显著:
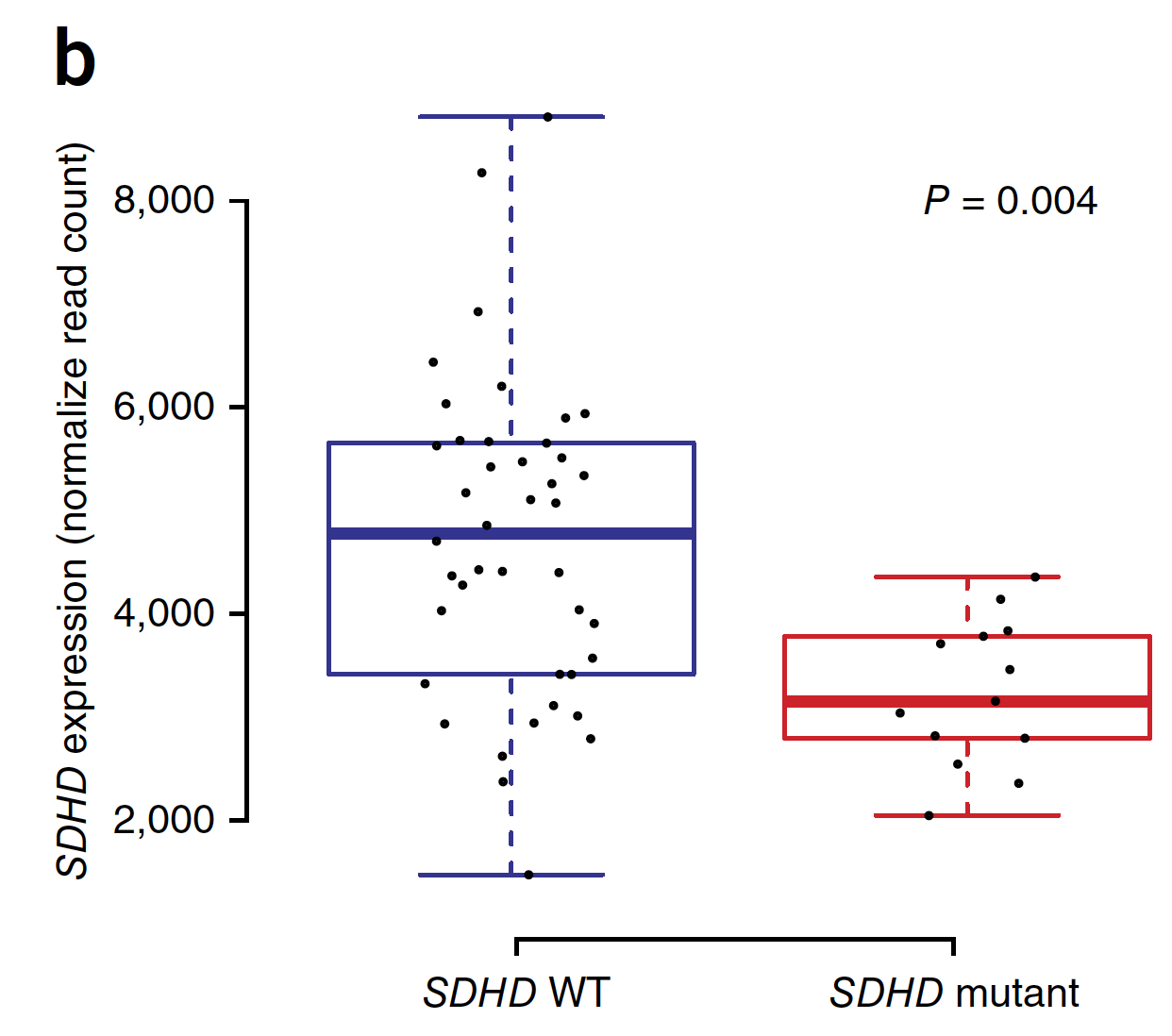
而且突变与否,完全决定了它与ELF1基因的表达量相关性,这个 ETS transcription factor (ELF1)
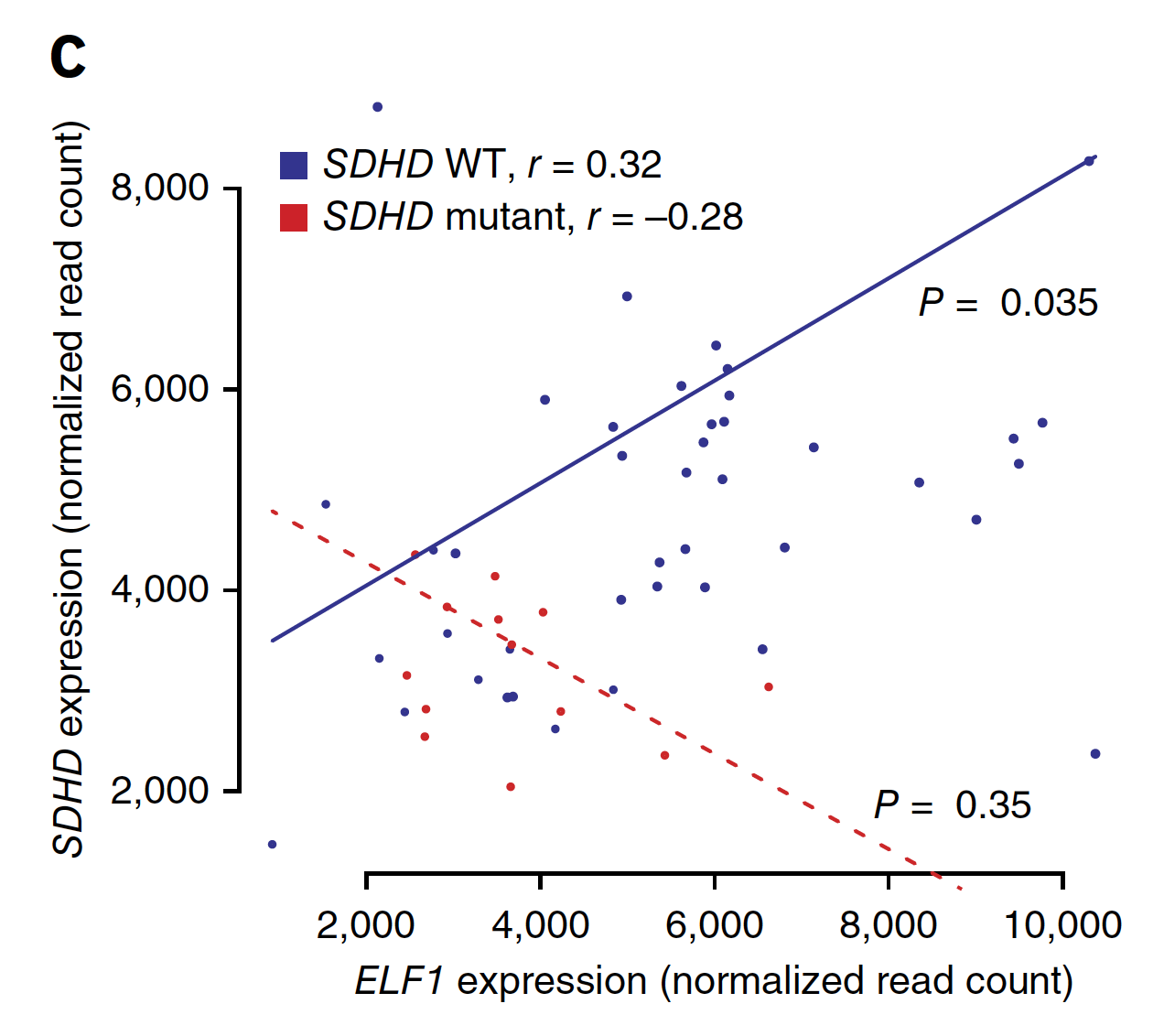
而且具有临床意义:
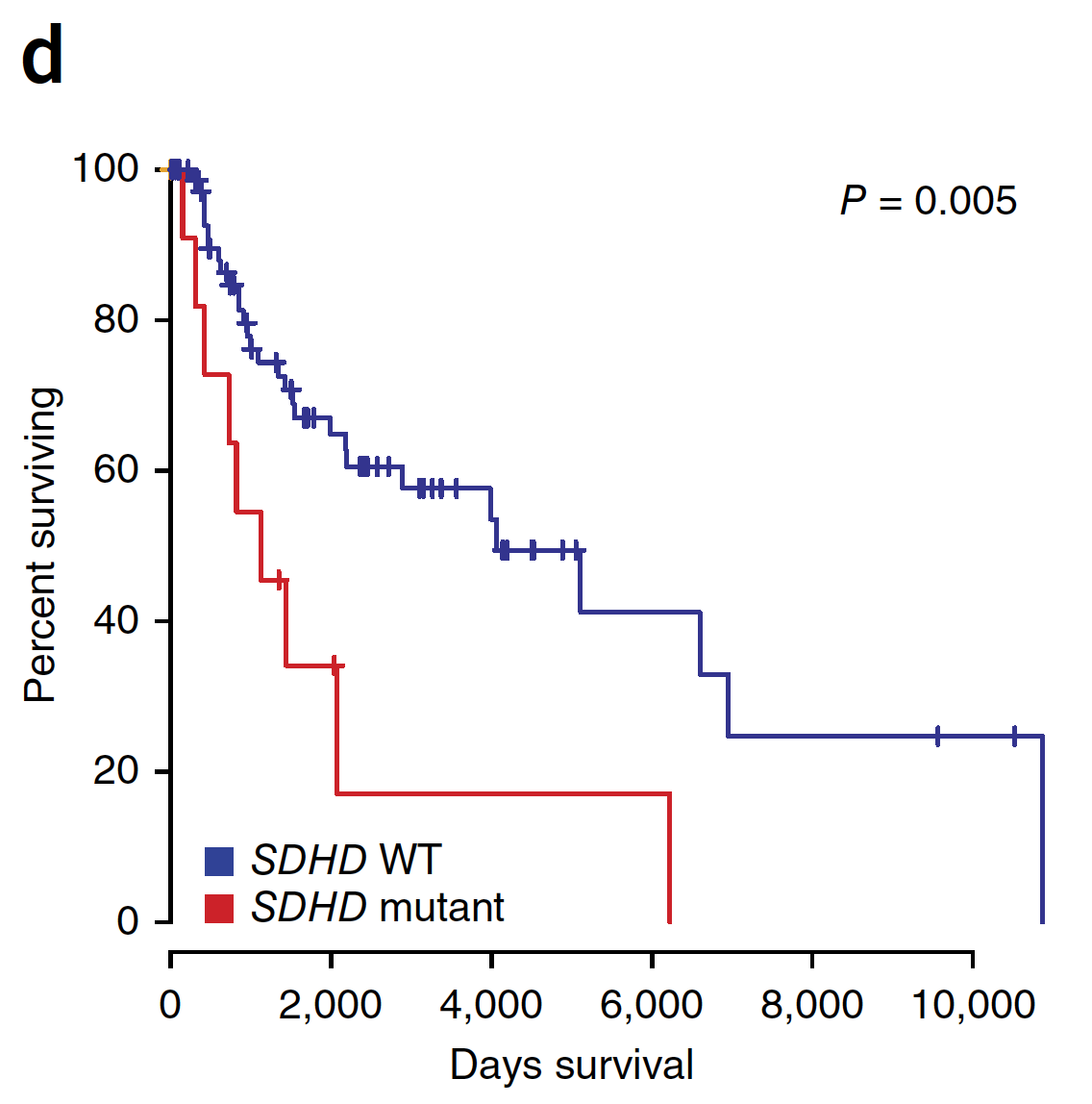
后记
本研究比较容易重复,只使用1000多个WGS数据的突变信息和表达量信息,关键是作者如何重海量数据里面定位到SDHD 和 ELF1 这个关系。
本文献解读属于100篇泛癌研究文献系列,首发于:http://www.bio-info-trainee.com/4132.html