前面我们介绍过卵巢癌领域的第一个类器官研究,发表于 September 13, 2018,题目是:Prediction of DNA Repair Inhibitor Response in Short Term Patient-Derived Ovarian Cancer Organoids 研究者共成功制备了33 organoid cultures derived from 22 HGSC patients ,但是做的数据分析很少,常规的WES+RNA测序数据,而且做的是短期培养,最后研究者从IHC结果还有SNV/CNV全景图来说明病人的肿瘤样品与其培养的类器官匹配情况。数据在phs001685.v1.p1需要申请才能下载。
这次我们要介绍的是卵巢癌领域的第二个类器官研究,发表于 Nat Med. 2019 Apr ,是类器官领域大名鼎鼎的Hans Clevers研究组,建立卵巢癌病人的组织进行类器官培养体系,作者们总共从32个不同病人组织建立了56个类器官品系。并且通过对于类器官细胞的形态学以及免疫组化染色,确认了类器官与病人组织切片的特征有着很好的一致性。
卵巢癌完整分类
参考: April 2019 https://doi.org/10.1038/s41591-019-0422-6
- borderline tumors (BTs; non-carcinoma) 约占15%
- primarily of serous BT (SBT)
- mucinous BT (MBT) subtypes
- type I (carcinomas)
- low-grade serous (LGS)
- mucinous(MC)
- endometrioid (END)
- clear cell (CCC) carcinomas
- type II tumors (carcinomas)
- high-grade serous (HGS) tumors
HGS的来源:
- high-grade serous (HGS) tumors
- fimbria of the fallopian tube (FT)
- surface epithelium (OSE)
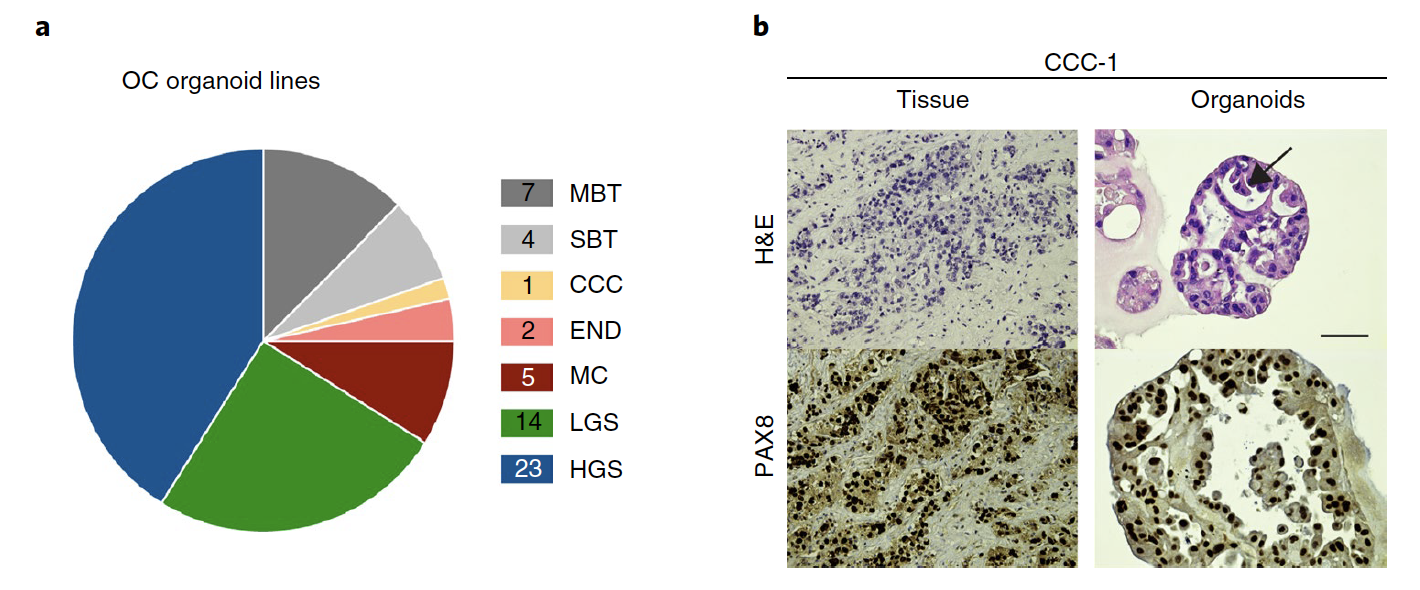
本研究纳入的卵巢癌样本分类情况
涵盖了几乎全部的卵巢癌亚型,如下:
相似性问题
一般类器官研究都需要证明类器官与病人组织切片的特征有着很好的一致性,如下表:
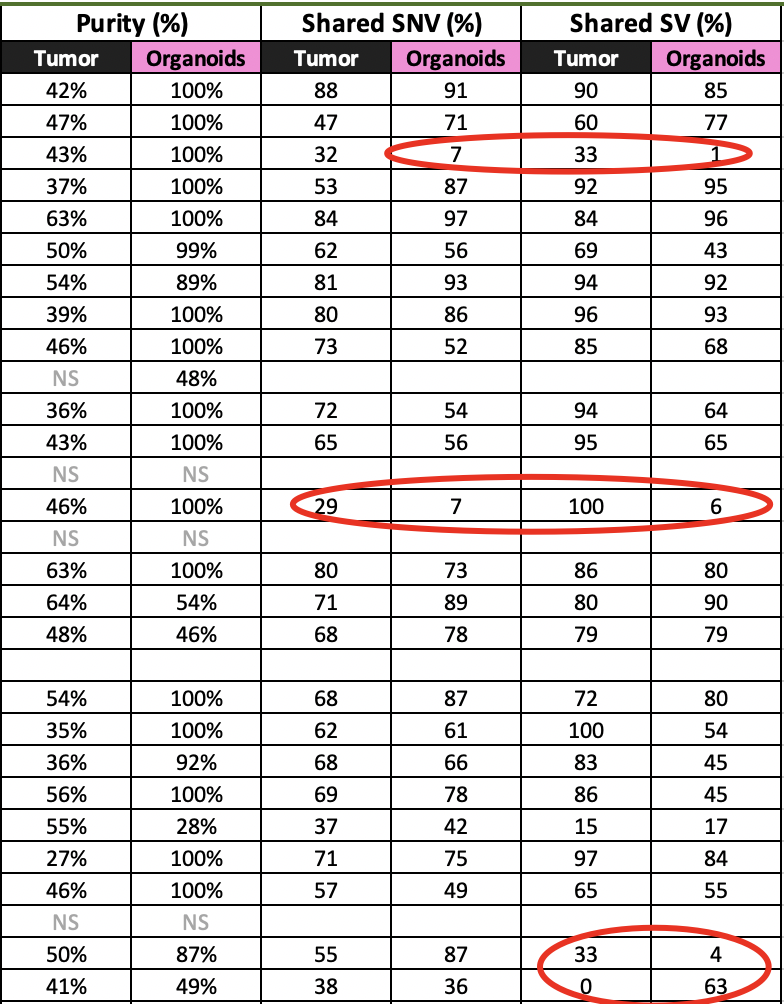
虽然有少部分病人的类器官跟其原位癌症的突变对不上,但是总体上来说,一致性是非常的好:
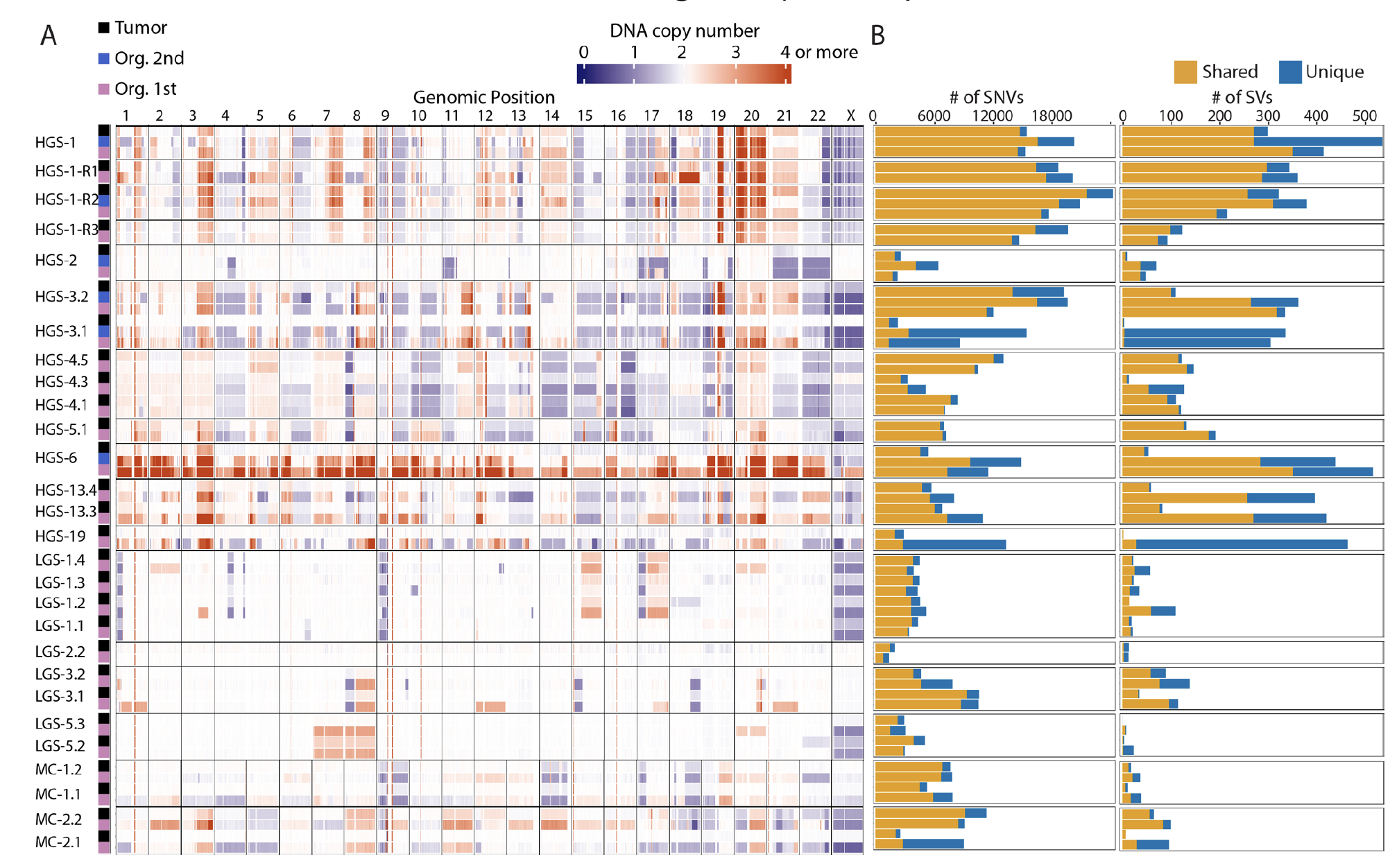
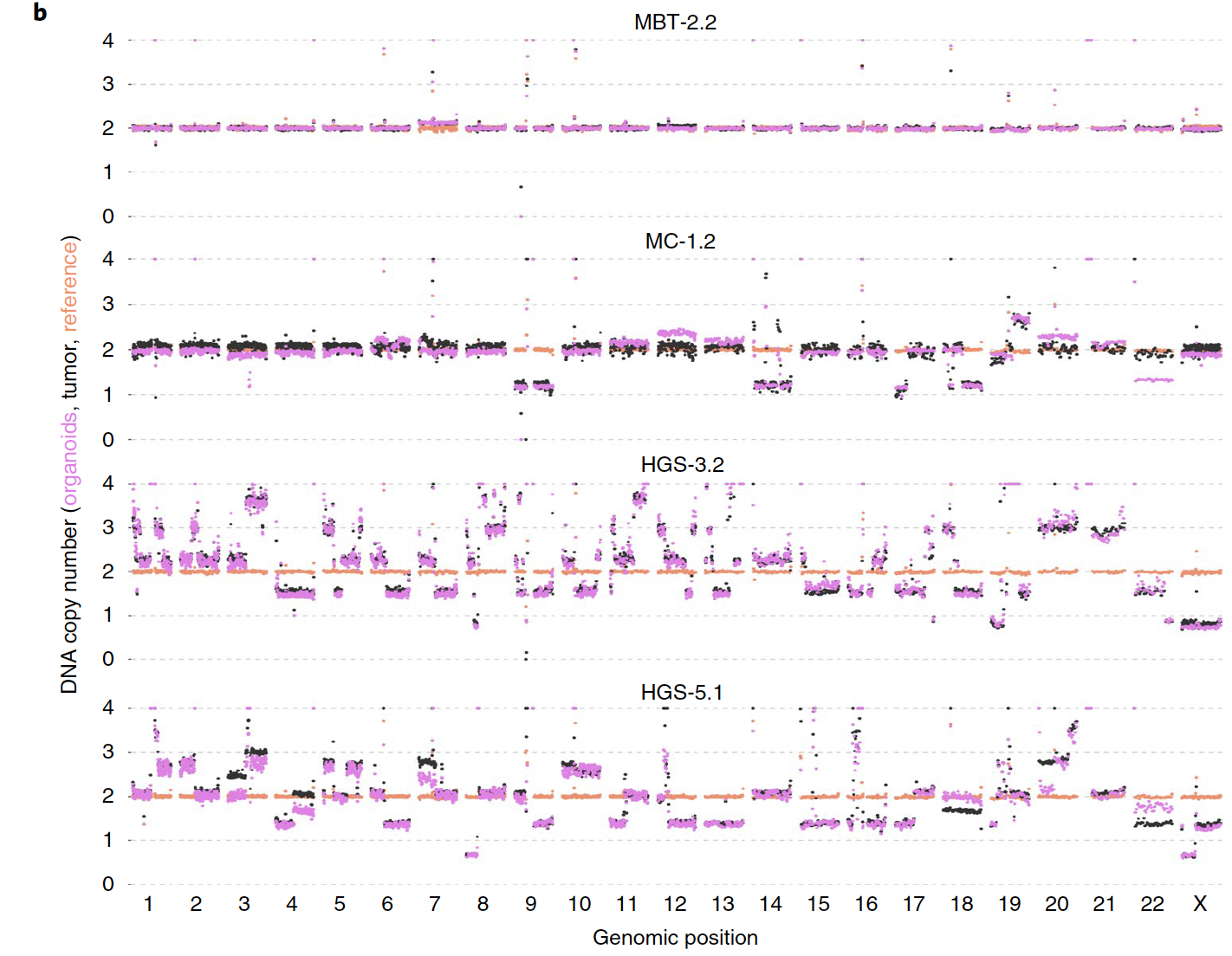
同样的,拷贝数变异也是这样的结果。首先是CNV相似性
使用control-free-C软件对WGS数据找CNV,相似性很明显:
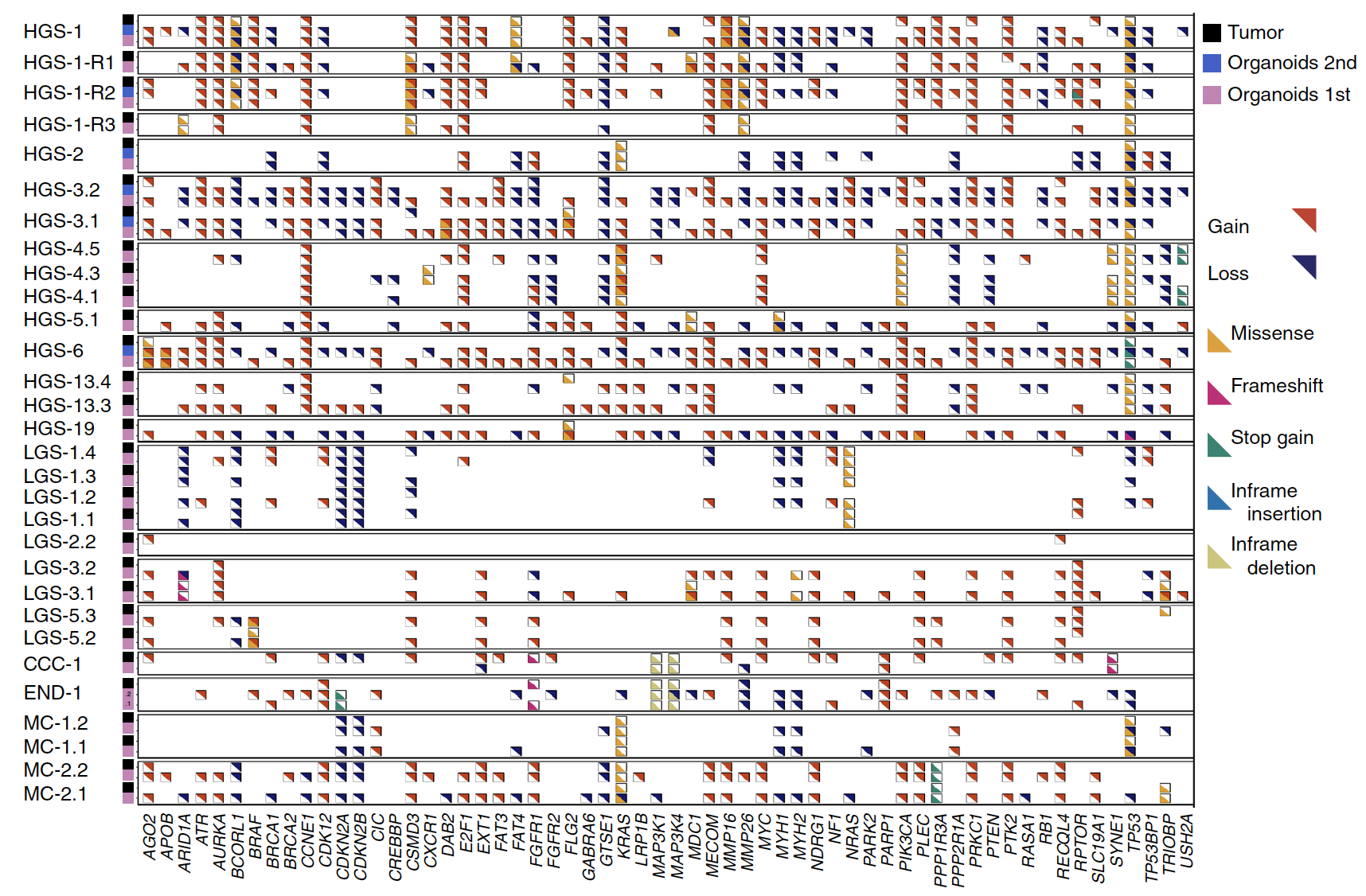
然后是SNV的相似性
跟他们实验室2018的乳腺癌文章一样,仅仅是展示感兴趣基因的突变一致性。
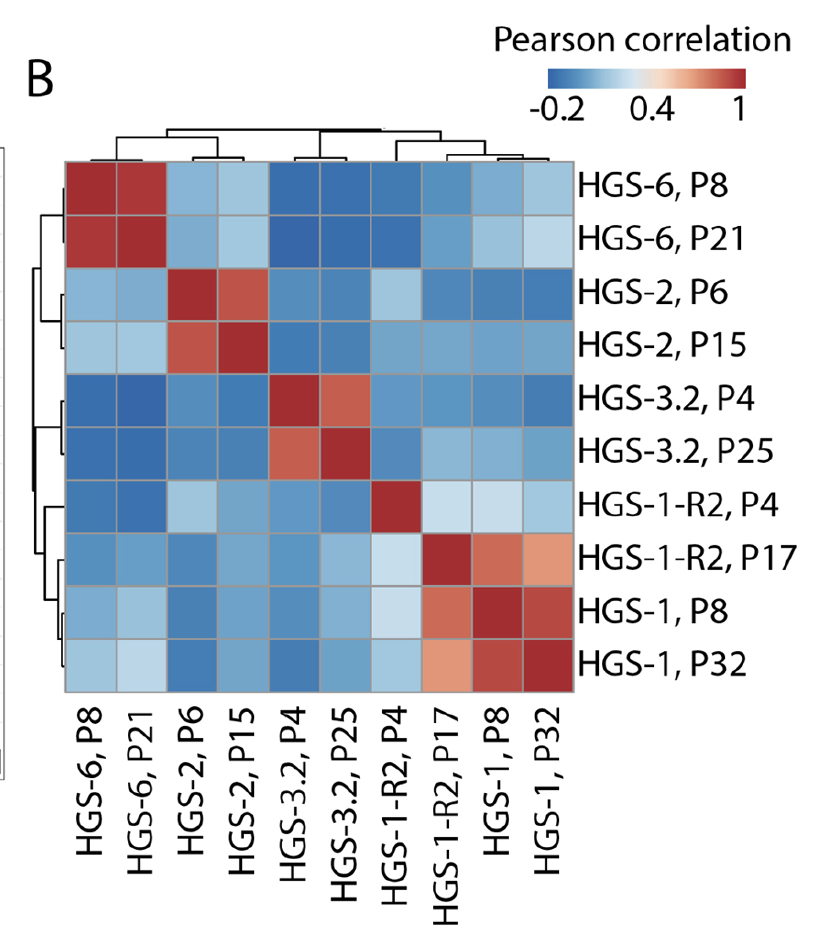
转录组数据看相似性
全文就放了一个相关性热图:
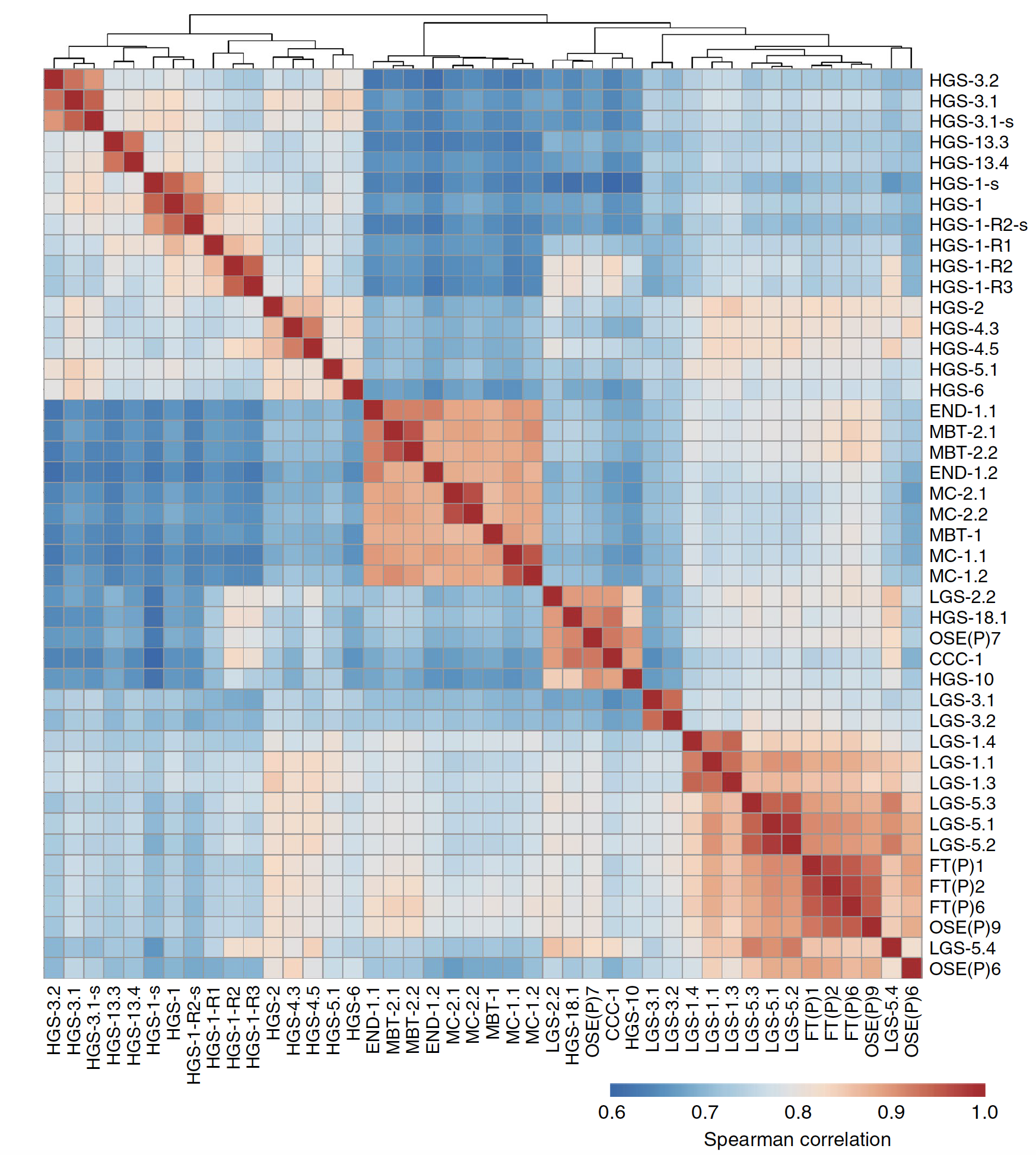
感觉Hans Clevers研究组一直把NGS数据当做是一个辅助其科研的实验手段,仅仅是做最基本的分析而已,就是为了说明类器官及其来源的病人的癌症样本的一致性而已。药物处理情况
既然是药物处理,必然有敏感人群和不敏感的, 根据IC50聚类如下:
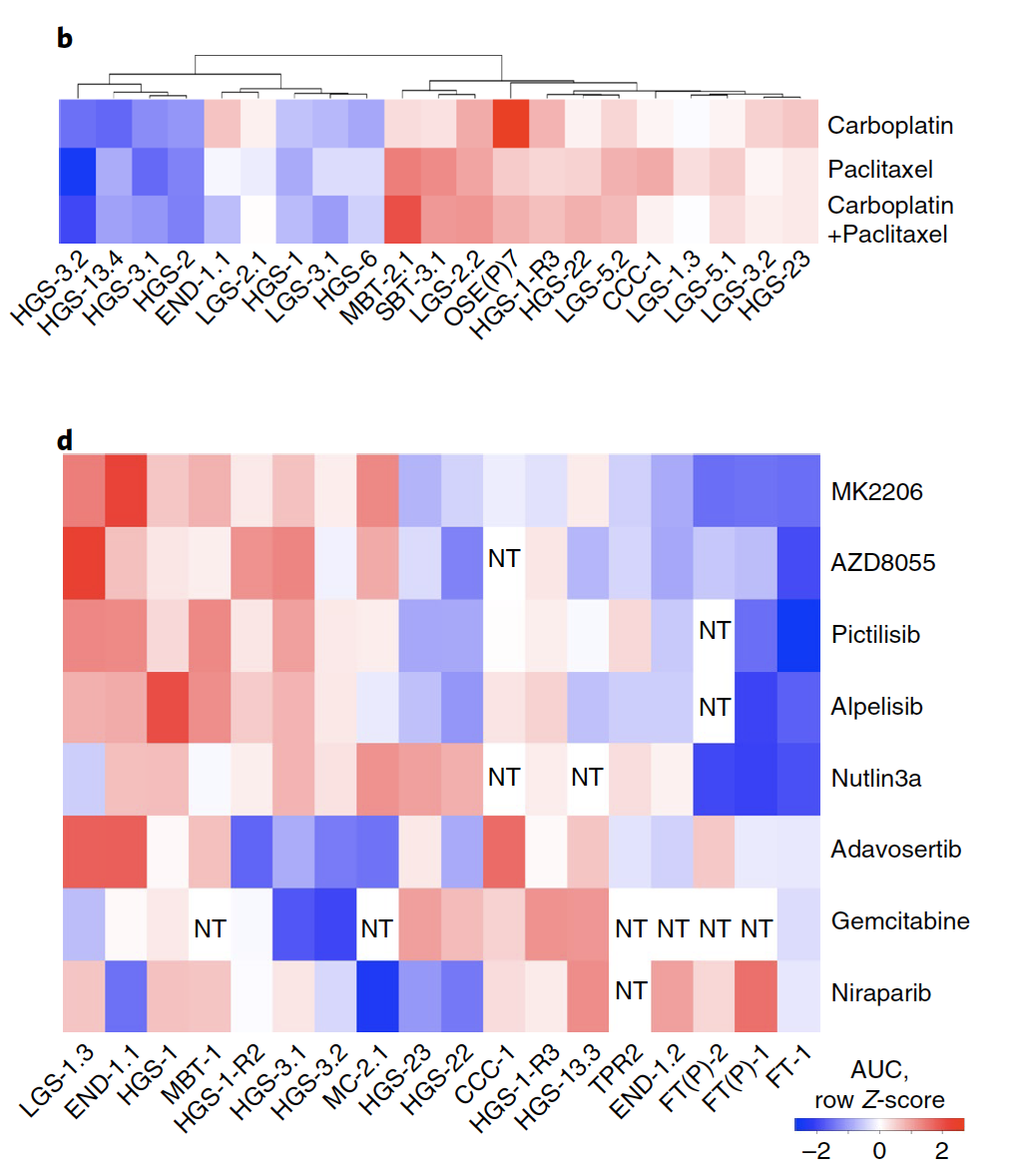
可以看到,药物处理反应情况可以比较好的分成两组,按照道理,这个时候可以对这两个组别进行差异分析等等,来寻找药物相关基因集,不过这个很显然不是作者的重点。多部位取样
有趣的是,这里作者涉及了一点肿瘤内部异质性的概念,可以看到对同一个病人的不同癌症部位取样测序,它们的异质性存在,相似性也有。
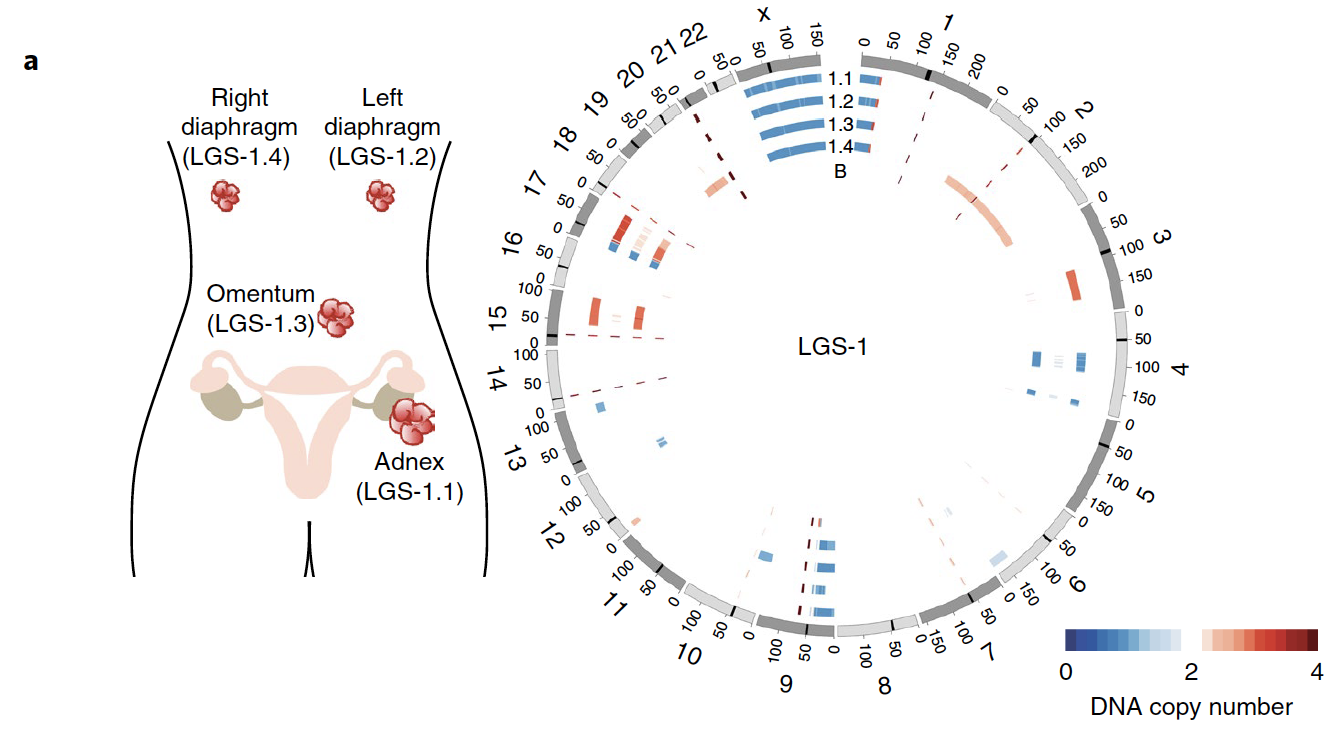
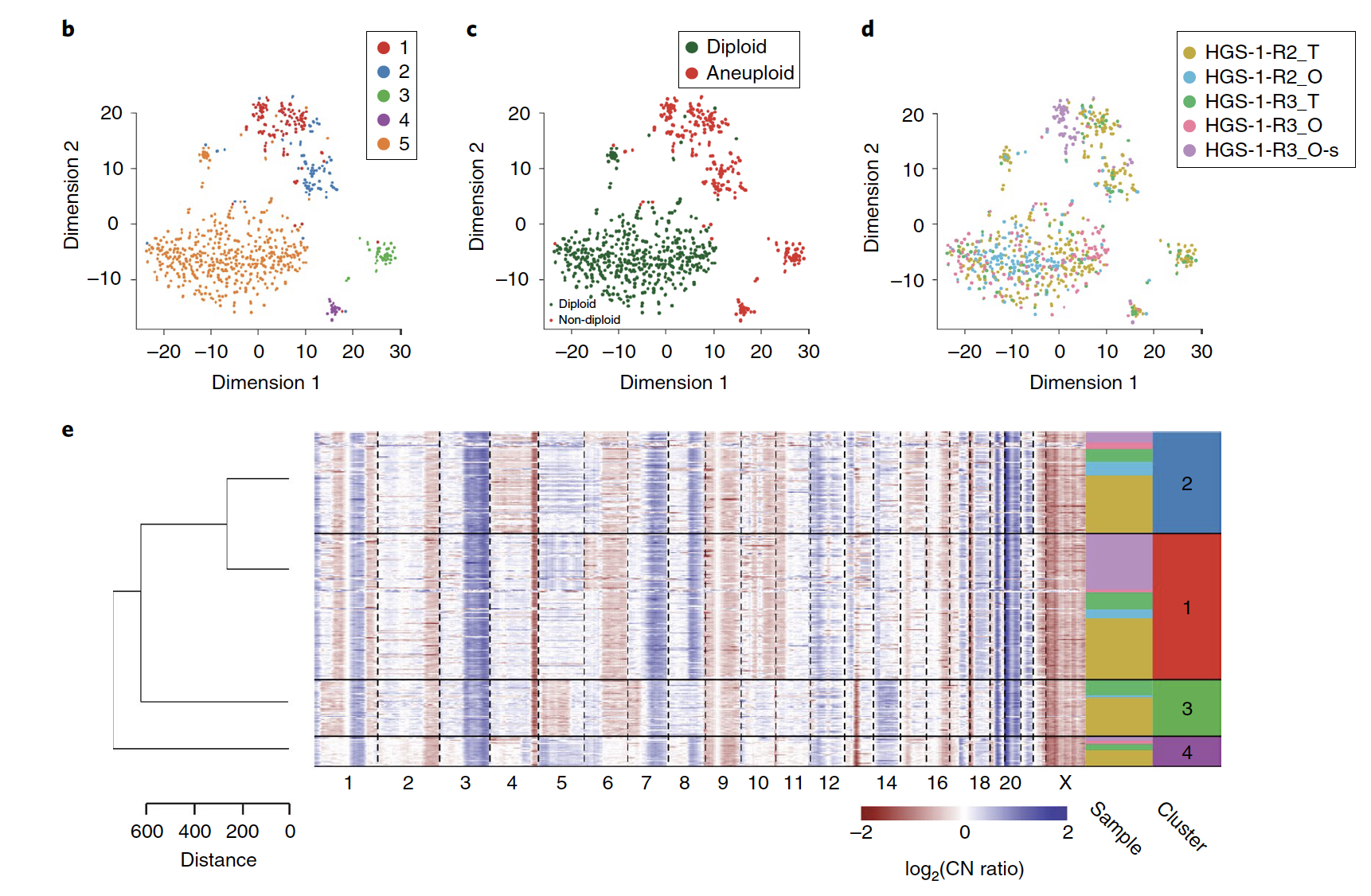
单细胞全基因组
细胞数量不多,因为单细胞研究还是比较前沿的,看起来Hans Clevers研究组也是才开始这方面建设。
不同病人的癌症部位和类器官都有二倍体细胞和非整倍体细胞。
850K甲基化芯片数据
这里也很简单,仅仅是挑选那些在样本中变化比较大的一万多个探针的信号值矩阵进行计算相关性而已。
Heat map of five independent organoid lines from both early and late passages based on 11,720 methylation probes. The heat map colors represent Pearson correlation values, as calculated from the methylation beta-values
看相关性: