这里我选取酵母基因组来组装,以为它只有一条染色体,而且本身也不大!
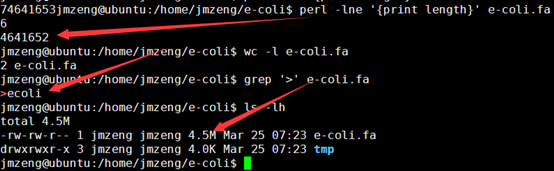
这个文件就4.5M,然后第一行就是序列名,第二列就是序列的碱基组成。共4641652个碱基。
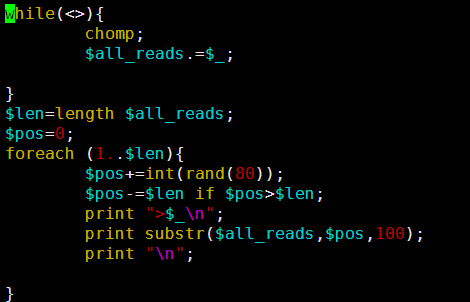
我写一个perl程序来人为的创造一个测序文件
这样我们的4.5M基因组就模拟出来了486M的单端100bp的测序数据,而且是无缝连接,按照道理应该很容易就拼接的。
/home/jmzeng/bio-soft/SOAPdenovo2-bin-LINUX-generic-r240/SOAPdenovo-127mer
all -s config_file -K 63 -R -o graph_prefix 1>ass.log 2>ass.err
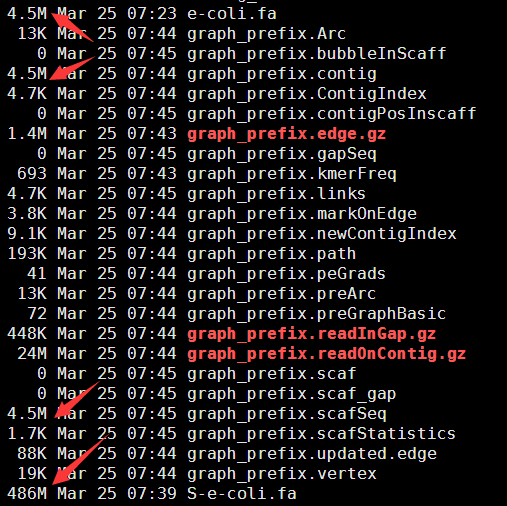
可以看到组装效果还不错哦,然后我模拟了一个测试数据,再进行组装一次,这次更好!
其实还可以模拟双端测序,应该就能达到百分百组装了。
但是由于我代码里面选取的是80在随机错开,所以我把kmer的长度设置成了81来试试看,希望这样可以把它完全组装成一条e-coli基因组。
/home/jmzeng/bio-soft/SOAPdenovo2-bin-LINUX-generic-r240/SOAPdenovo-127mer
all -s config_file -K 81 -R -o graph_prefix 1>ass.log 2>ass.err
但是也没有什么实质性的提高,虽然理论上是肯定可以组装到一起!
那我再模拟一个双端测序吧,中间间隔200bp的。