每月一生信流程栏目灵感来自于《铁汉1991》博客的《每日一生信》,他那个时候介绍的主要是生信基础知识,包括数据结构,数据格式,数据库资源,计算机基础等等,所以每天都可以进步,每天都有成果。这些基础知识已经被分享的七七八八了,所以我这里推陈出新,来一个每月一生信流程,陪生信技能树的粉丝们一起进步!
前面我们推荐3个值得用一个月时间来学习的流程,包括
但是转录组研究不仅仅是RNA-seq这样的NGS技术可以得到表达矩阵,比较传统的芯片技术也积累了海量的数据,值得我们挖掘,现在我们就一起学习一个表达芯片数据处理流程吧:
We will start from the raw data CEL files, show how to import them into a Bioconductor ExpressionSet, perform quality control and normalization and finally differential gene expression (DE) analysis, followed by some enrichment analysis.
今天学习maEndToEnd
流程的原标题是:An end to end workflow for differential gene expression using Affymetrix microarrays
这个同样的也没有中文版,但实际上表达芯片数据处理流程,我已经分享过太多太多了,并不比bioconductor少。
在R里面安装这个bioconductor流程
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install("maEndToEnd")
因为原文写的实在是太详细了,我这里就不拷贝粘贴了,大家直接去阅读即可: 流程代码
全部目录如下;
- 1.Introduction
- 2.Workflow package installation
- 3.Downloading the raw data from ArrayExpress
- 4.Background information on the data
- 5.Import of annotation data and microarray expression data as “ExpressionSet”
- 6.Quality control of the raw data
- 7.Background adjustment, calibration, summarization and annotation
- 8.Relative Log Expression data quality analysis
- 9.RMA calibration of the data
- 10.Filtering based on intensity
- 11.Annotation of the transcript clusters
- 12.Linear models
- 13.Gene ontology (GO) based enrichment analysis
- 14.A pathway enrichment analysis using reactome
流程代码
同样的需要熟练掌握ExpressionSet这个对象,以及它配套的3个函数:
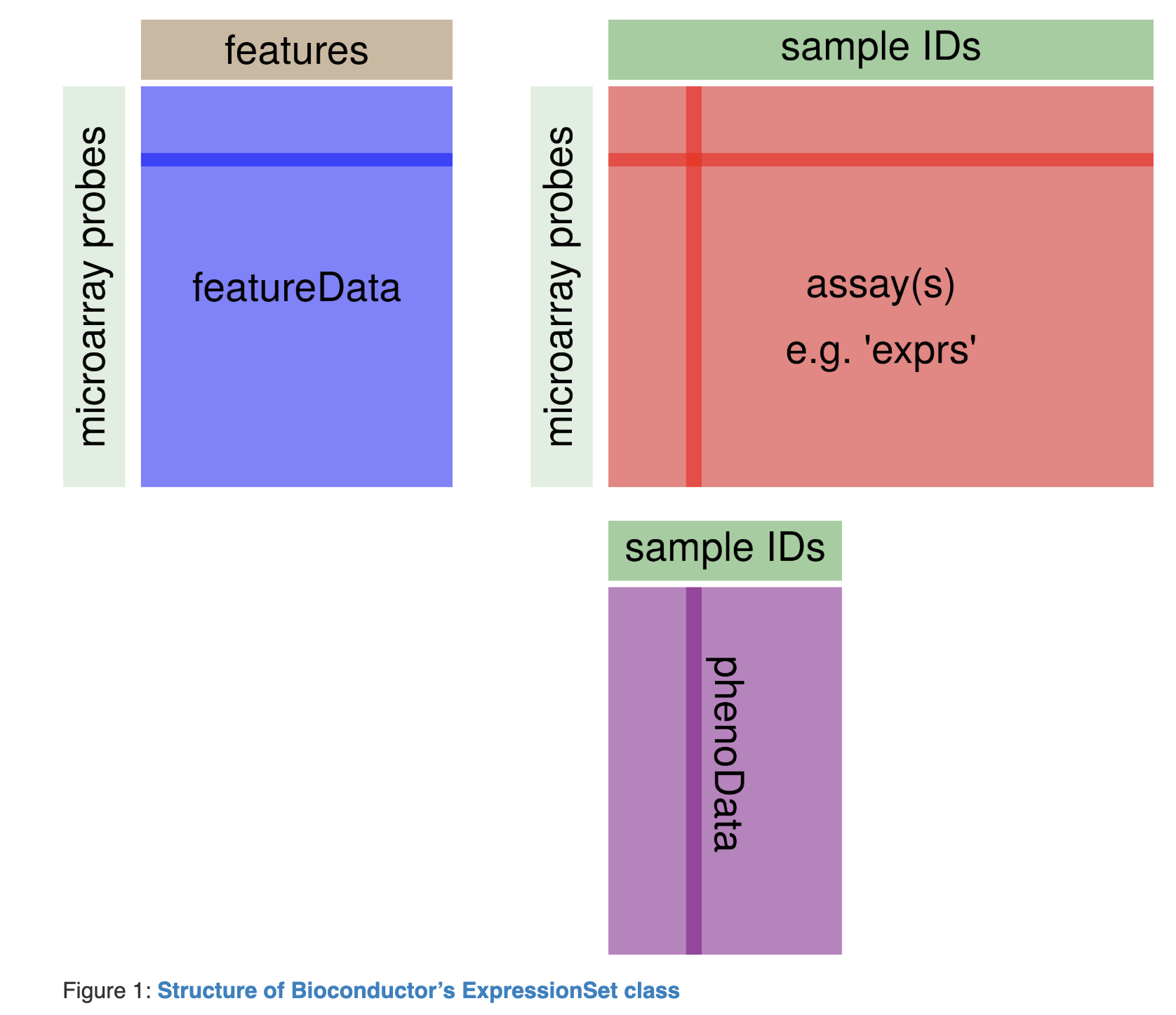
You can use the functions pData and fData to extract the sample and feature annotation, respectively, from an ExpressionSet. The function exprs will return the expression data itself as a matrix.
以及为什么需要对原始的表达芯片进行一定程度的预处理:
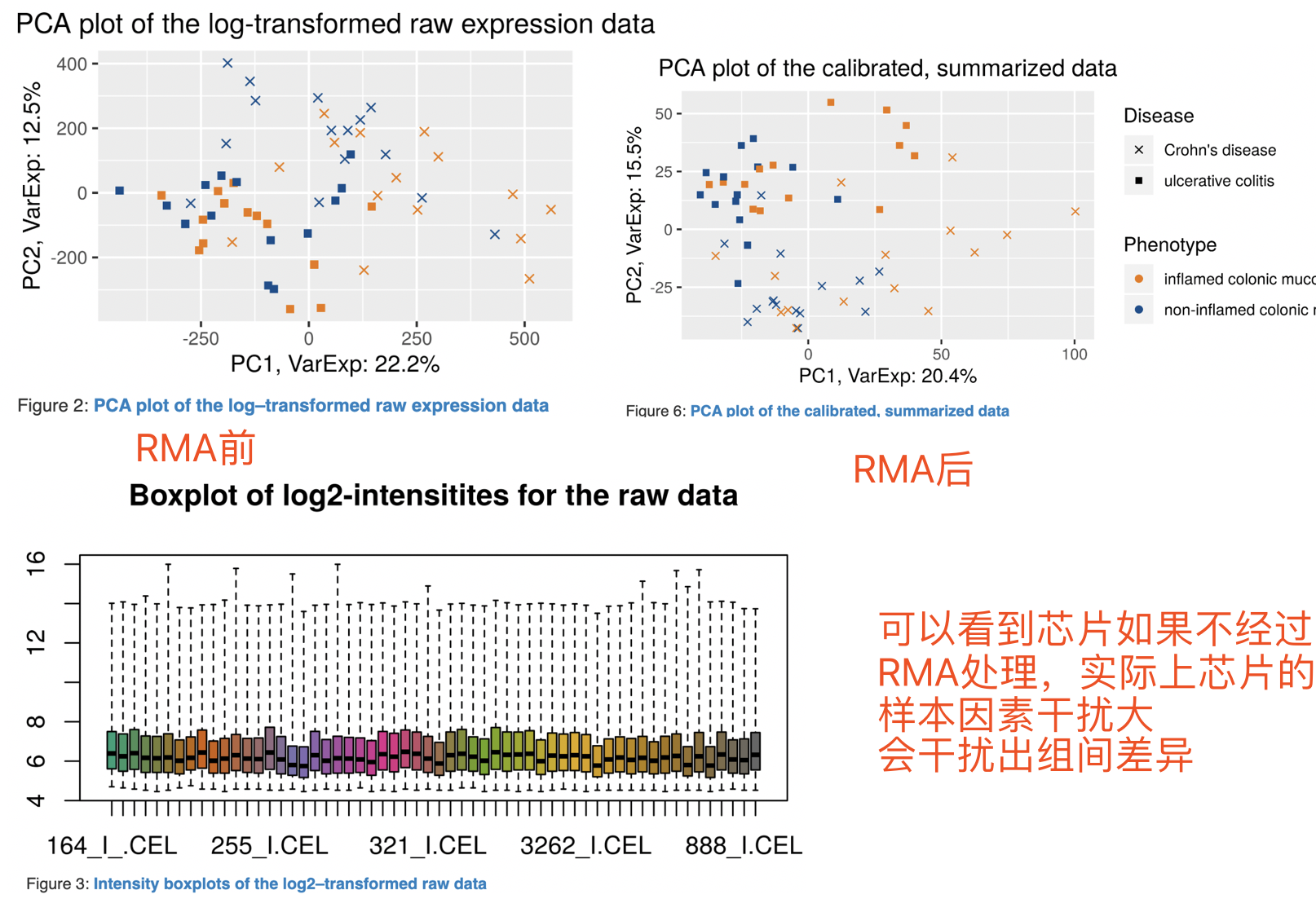
有了芯片表达矩阵,后续分析其实等同于RNA-seq的表达矩阵分析流程,只不过是里面的一些R包需要替换一下。
学习这样的流程是需要一定背景知识的
首先是LINUX学习
我在《生信分析人员如何系统入门Linux(2019更新版)》把Linux的学习过程分成6个阶段 ,提到过每个阶段都需要至少一天以上的学习:
- 第1阶段:把linux系统玩得跟Windows或者MacOS那样的桌面操作系统一样顺畅,主要目的就是去可视化,熟悉黑白命令行界面,可以仅仅以键盘交互模式完成常规文件夹及文件管理工作。
- 第2阶段:做到文本文件的表格化处理,类似于以键盘交互模式完成Excel表格的排序、计数、筛选、去冗余,查找,切割,替换,合并,补齐,熟练掌握awk,sed,grep这文本处理的三驾马车。
- 第3阶段:元字符,通配符及shell中的各种扩展,从此linux操作不在神秘!
- 第4阶段:高级目录管理:软硬链接,绝对路径和相对路径,环境变量
- 第5阶段:任务提交及批处理,脚本编写解放你的双手
- 第6阶段:软件安装及conda管理,让linux系统实用性放飞自我
然后是R学习
我在生信分析人员如何系统入门R(2019更新版) 里面给初学者的知识点路线图如下:
- 了解常量和变量概念
- 加减乘除等运算(计算器)
- 多种数据类型(数值,字符,逻辑,因子)
- 多种数据结构(向量,矩阵,数组,数据框,列表)
- 文件读取和写出
- 简单统计可视化
- 无限量函数学习
必备书籍及视频
书籍贪多不烂,下面2本必买,读5遍以上
视频必须强推生信技能树近30万学习量的基础合辑:
生信技能树关于表达芯片的公共数据库挖掘教程的确不少了
因为做目录确实很浪费时间,差不多就下面这些,生信技能树B站系列教学视频,读者需要细读表达芯片的公共数据库挖掘系列推文 ;
- 解读GEO数据存放规律及下载,一文就够
- 解读SRA数据库规律一文就够
- 从GEO数据库下载得到表达矩阵 一文就够
- GSEA分析一文就够(单机版+R语言版)
- 根据分组信息做差异分析- 这个一文不够的
- 差异分析得到的结果注释一文就够
然后看B站的GEO数据挖掘技巧,基本上该分享的都在B站和GitHub了,目录如下:
- 第一讲:GEO,表达芯片与R
- 第二讲:从GEO下载数据得到表达量矩阵
- 第三讲:对表达量矩阵用GSEA软件做分析
- 第四讲:根据分组信息做差异分析
- 第五讲:对差异基因结果做GO/KEGG超几何分布检验富集分析
- 第六讲:指定基因分组boxplot指定基因list画热图
- 第七讲:根据差异基因list获取string数据库的PPI网络数据
- 第八讲:PPI网络数据用R或者cytoscape画网络图
- 第九讲:网络图的子网络获取
- 第十讲:hug genes如何找
后记
听说隔壁openbiox团队在组织翻译这个bioconductor流程系列,而且还是由我们生信技能树元老-思考问题的熊领头,希望他们的翻译成果早日出版!