最开始分享过芯片探针注释到基因名的3种方法:
- 1金标准当然是去基因芯片的厂商的官网直接去下载
- 2一种是直接用bioconductor的包
- 3一种是从NCBI里面下载文件来解析
见原文
现在我完成作业就遇到了gpl16699平台,它并没有bioconductor的包,所以需要下载gpl平台的soft文件,进行注释,官网https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GPL16699 可以看到信息如下:
现在使用r代码完成这个步骤
方法一:使用GEOquery包
rm(list = ls()) ## 魔幻操作,一键清空~
options(stringsAsFactors = F)
library(GEOquery)
gpl=getGEO("GPL16699",destdir = '.')
names(Meta(gpl))
Table(gpl)[1:10,1:2]
ids=Table(gpl)[,c("NAME","GENE_SYMBOL")]
head(ids)
colnames(ids)=c('probe_id','symbol')
head(ids)
这一个方法需要网速保障,并且确保下载的GPL完整,不然会影响后面的操作。
方法二:下载txt文件
在官网https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GPL16699 下载txt文件
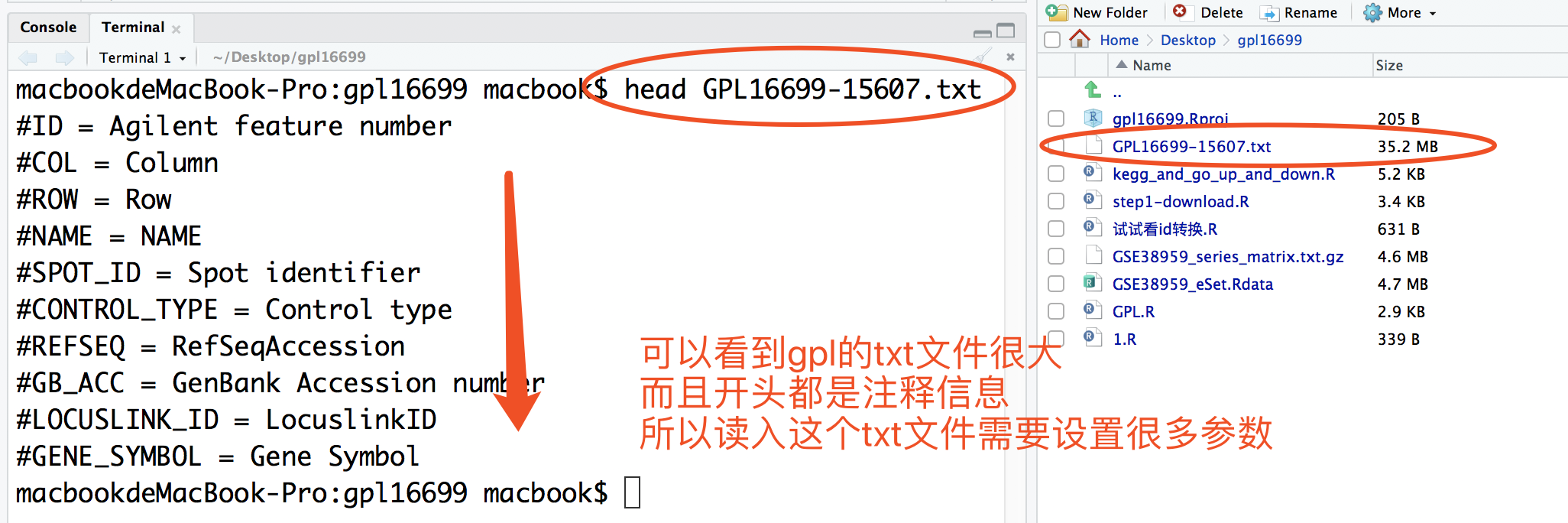
读取它的代码如下:
rm(list = ls()) ## 魔幻操作,一键清空~
options(stringsAsFactors = F)
gpl=read.table("GPL16699-15607.txt",
header = TRUE,fill = T,sep = "\t",
comment.char = "#",
stringsAsFactors = FALSE,
quote = "")
head(gpl)
colnames(gpl)
ids=gpl[,c("NAME","GENE_SYMBOL")]
head(ids)
colnames(ids)=c('probe_id','symbol')
head(ids)
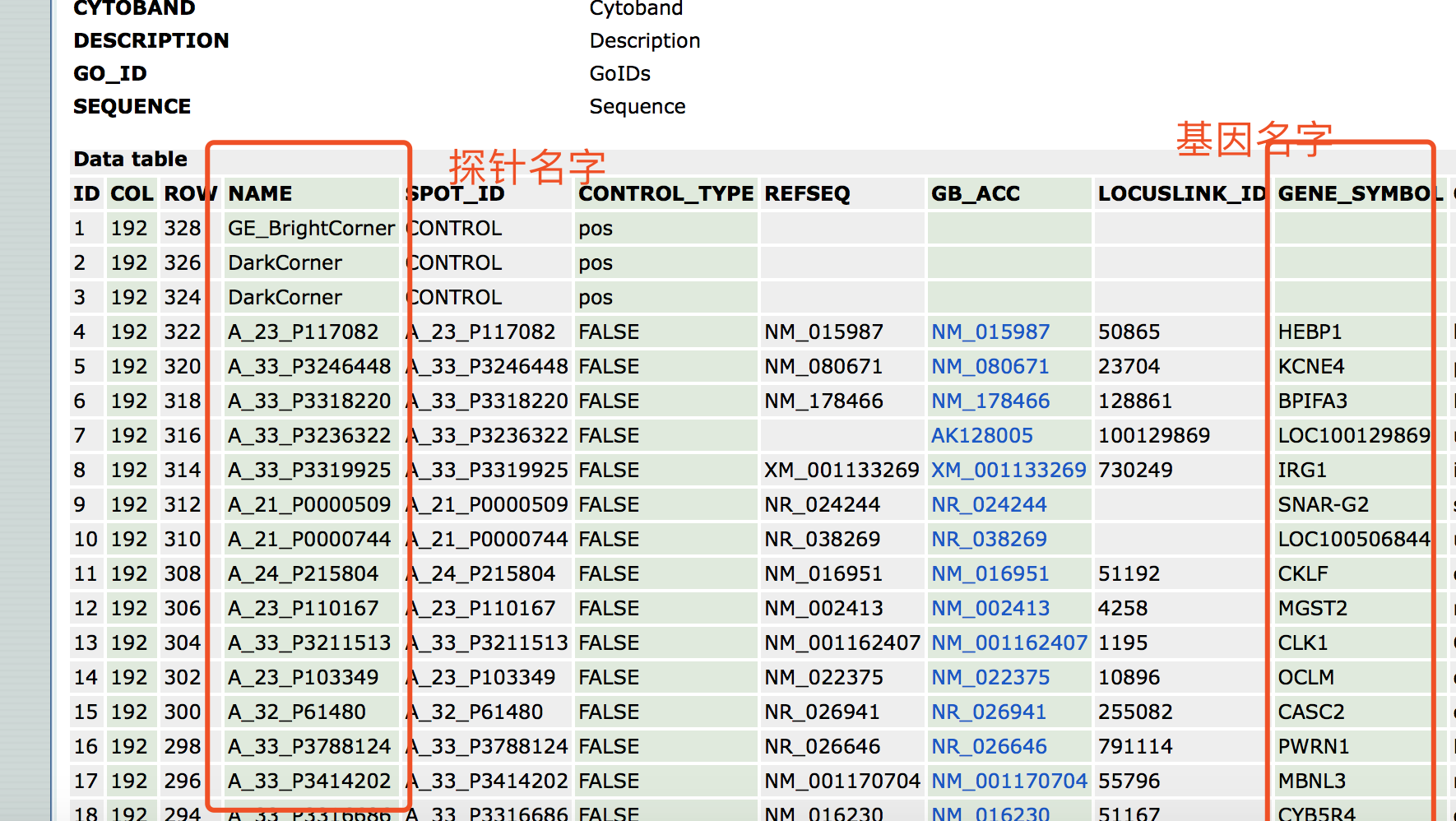
这个就是gpl16699的探针ID与基因名
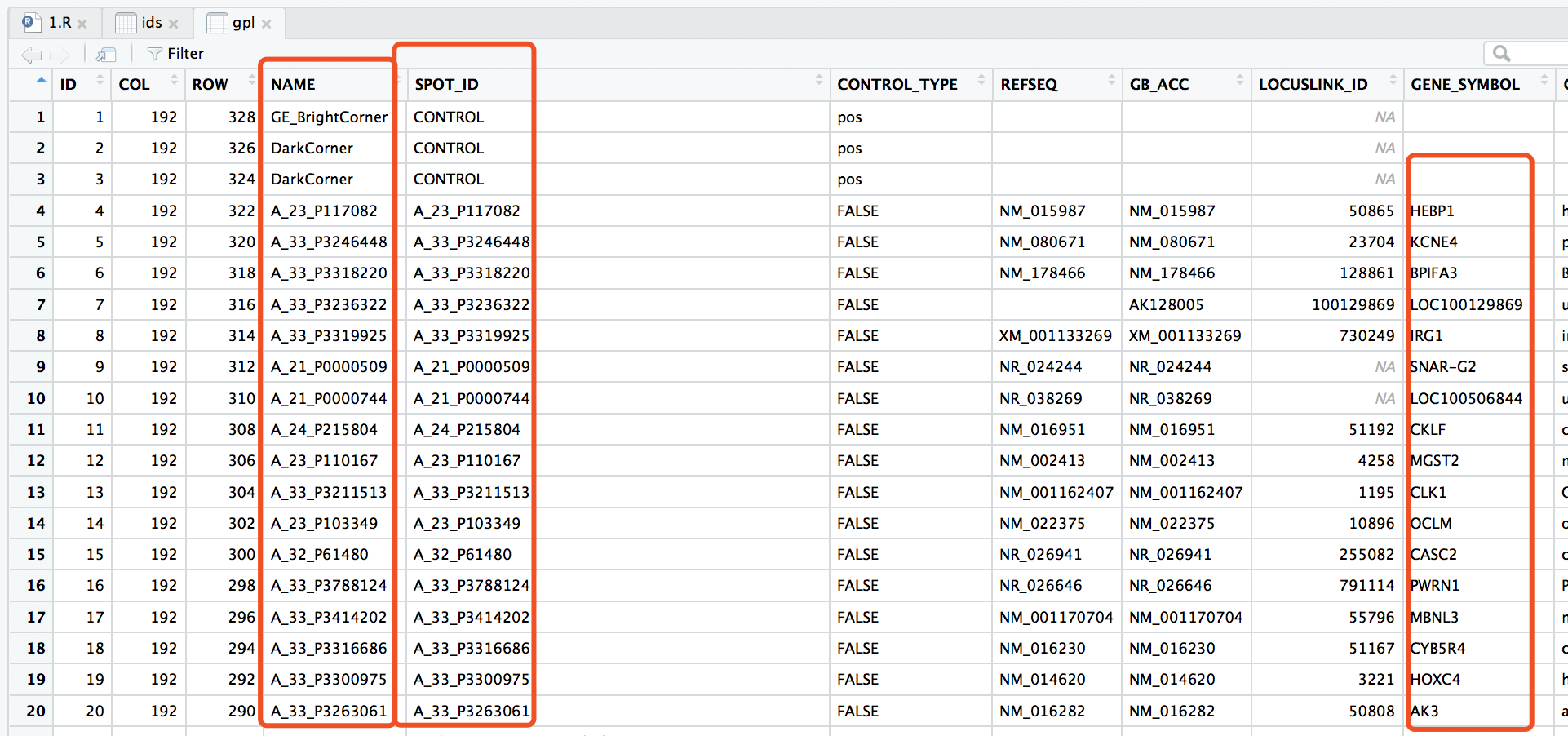
对比一下,方法一里面的getGEO函数下载后使用Table提取到的内容,就是方法二的txt文件,后续处理一致。
大功告成!
后续挖掘见生信技能树B站系列教学视频
需要细读表达芯片的公共数据库挖掘系列推文 ;
- 解读GEO数据存放规律及下载,一文就够
- 解读SRA数据库规律一文就够
- 从GEO数据库下载得到表达矩阵 一文就够
- GSEA分析一文就够(单机版+R语言版)
- 根据分组信息做差异分析- 这个一文不够的
- 差异分析得到的结果注释一文就够
然后看B站的GEO数据挖掘技巧,基本上该分享的都在B站和GitHub了,目录如下:
- 第一讲:GEO,表达芯片与R
- 第二讲:从GEO下载数据得到表达量矩阵
- 第三讲:对表达量矩阵用GSEA软件做分析
- 第四讲:根据分组信息做差异分析
- 第五讲:对差异基因结果做GO/KEGG超几何分布检验富集分析
- 第六讲:指定基因分组boxplot指定基因list画热图
- 第七讲:根据差异基因list获取string数据库的PPI网络数据
- 第八讲:PPI网络数据用R或者cytoscape画网络图
- 第九讲:网络图的子网络获取
- 第十讲:hug genes如何找