最近刷文献,发现一个很有趣的火山图,文献是发表在 October 7, 2019的PNAS:Immune effector monocyte–neutrophil cooperation induced by the primary tumor prevents metastatic progression of breast cancer
可以展现指定基因集的火山图
这篇文章的测序数据是公布的:https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE137300 表达矩阵及差异分析结果,都可以直接下载。
可以看到测序策略是:
GSM4074939 mRNA_Neutrophils_TN1 [tag_191]
GSM4074940 mRNA_Neutrophils_TN1 [tag_482]
GSM4074941 mRNA_Neutrophils_TN2 [tag_367]
GSM4074942 mRNA_Neutrophils_TN2 [tag_706]
GSM4074943 mRNA_Neutrophils_TN2 [tag_1341]
GSM4074944 mRNA_Monocytes_CCR2+_TN1 [tag_191]
GSM4074945 mRNA_Monocytes_CCR2+_TN1 [tag_482]
GSM4074946 mRNA_Monocytes_CCR2+_TN2 [tag_367]
GSM4074947 mRNA_Monocytes_CCR2+_TN2 [tag_706_]
GSM4074948 mRNA_Monocytes_CCR2+_TN2 [tag_1341]
其中:
- the highly metastatic tumors HCl-001 (TN1)
- low metastatic tumors HCl-002 (TN2).
也就是说两种tumor,然后两种细胞,这样的4个分组,但是里面的火山图(不要问我是如何看出来这是一个火山图的)如下:
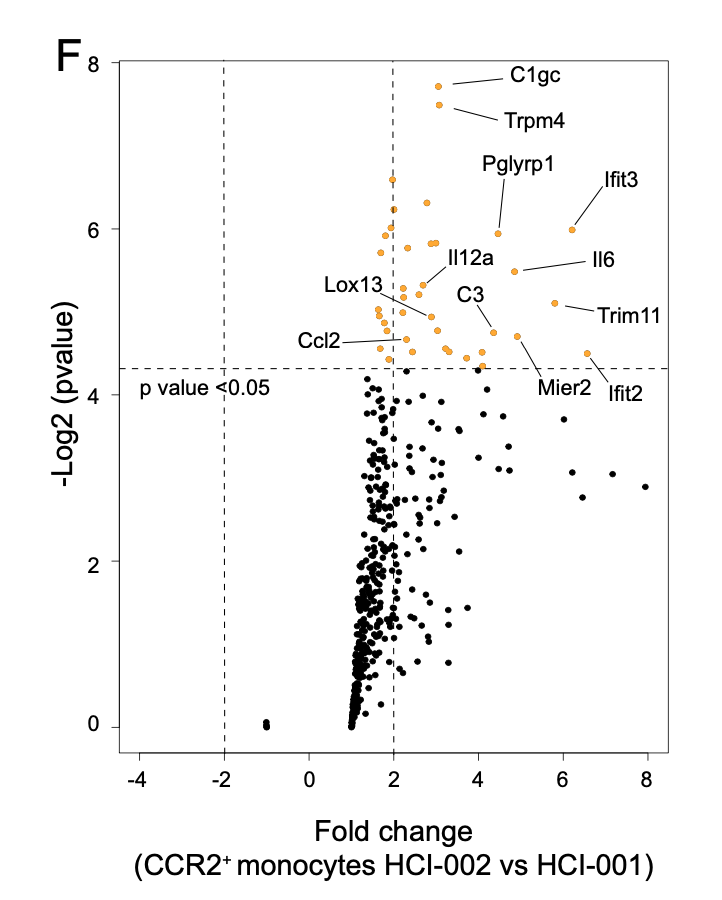
可以看到,是缺半边的,如果你仔细看文章,其实是因为作者使用这样的图是为了选择性的展示数据来说明他自己的生物学故事,他们这个图展现的仅仅是 immune effector genes (GO:0002252) 的基因。
并不是全部的基因。
通常我们的火山图左右两边的点数量差不多,就是说上下调基因数量。
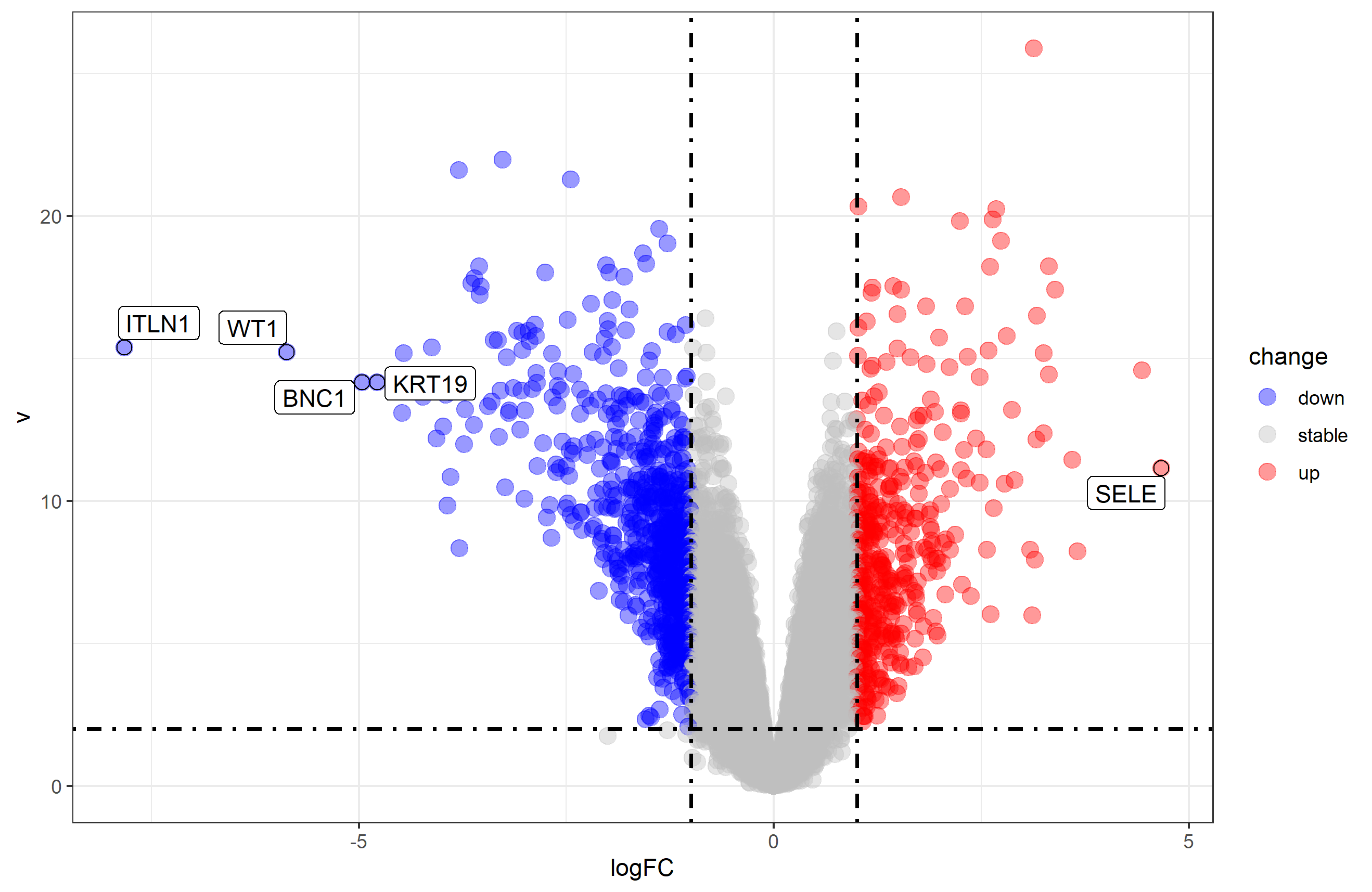
看起来比较正常的火山图是
随便搜索一下很多关于火山图的介绍,我这里就不赘述了。
也有一些时候全部基因的火山图也不正常
如下,我在安排学徒完成11个GSE数据集分析的时候,发现其中一个(GSE21785)的火山图看起来略微有点诡异!
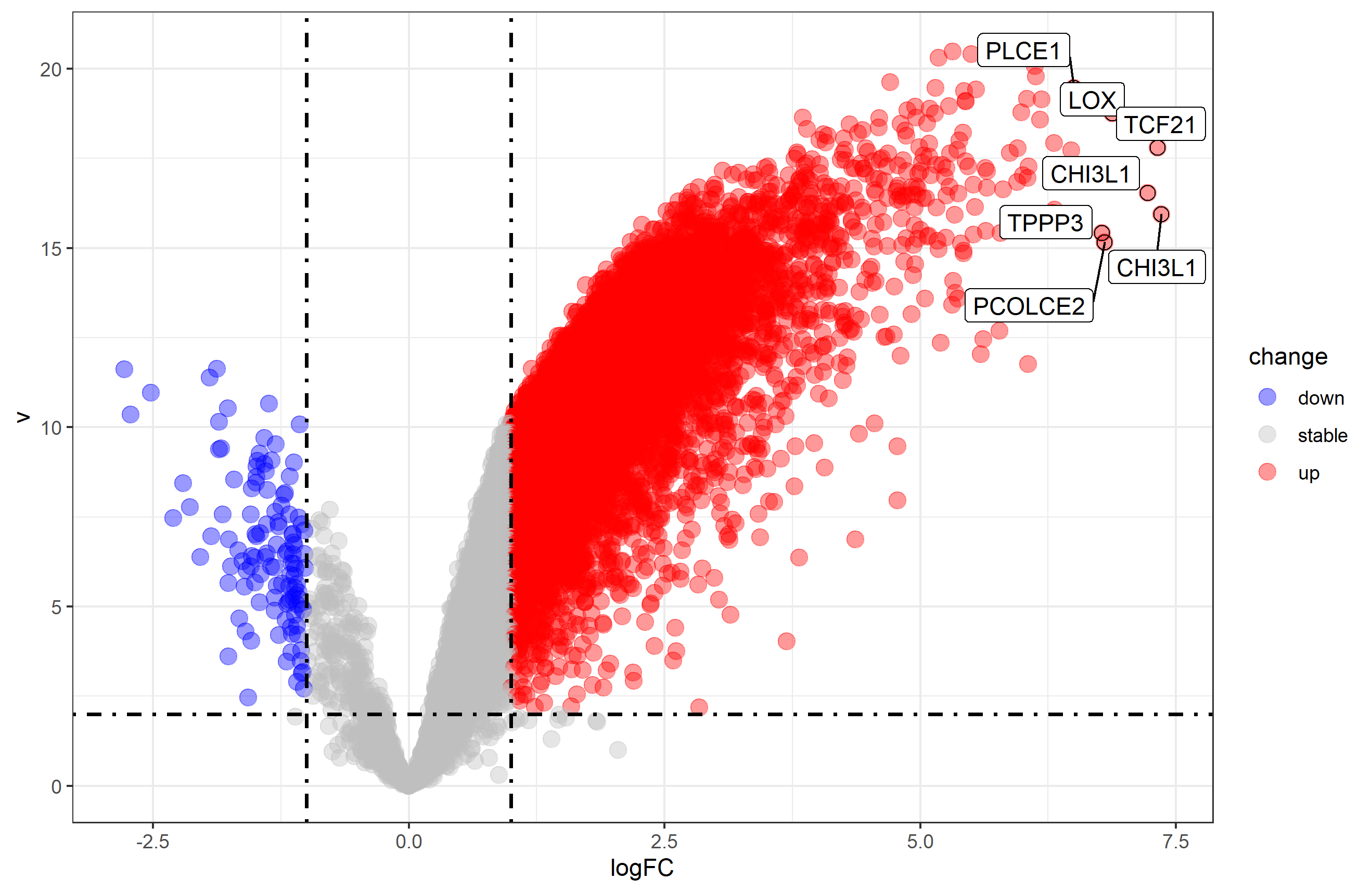
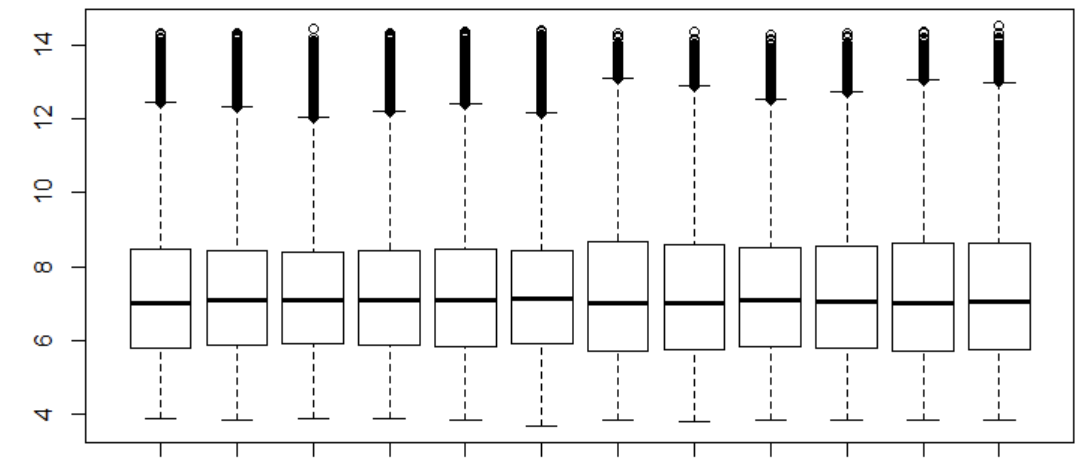
检查代码的时候发现,其表达量分布具有分组差异。
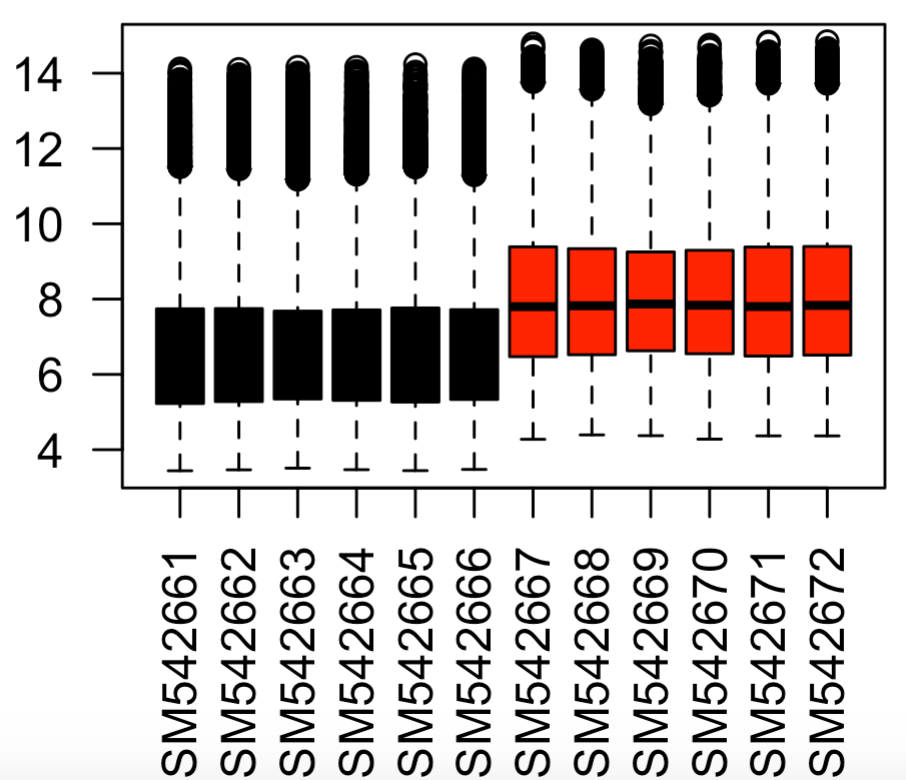
但是如果你从作者上传的芯片原始数据(GSE21785)开始,就会发现,是下面的分布:
原始数据的处理,大家参考:你要挖的公共数据集作者上传了错误的表达矩阵肿么办(如何让高手心甘情愿的帮你呢?) 里面的代码:
# BiocManager::install(c( 'oligo' ),ask = F,update = F)
library(oligo)
# BiocManager::install(c( 'pd.hg.u133.plus.2' ),ask = F,update = F)
library(pd.hg.u133.plus.2)
dir='~/Downloads/GSE84571_RAW/'
od=getwd()
setwd(dir)
celFiles <- list.celfiles(listGzipped = T)
celFiles
affyRaw <- read.celfiles( celFiles )
setwd(od)
eset <- rma(affyRaw)
eset
# http://math.usu.edu/jrstevens/stat5570/1.4.Preprocess_4up.pdf
save(eset,celFiles,file = f)
# write.exprs(eset,file="data.txt")
得到的eset这个对象,与我们之前一直讲解的GEOquery包下载是一样的, 所以后续代码不需要变化。
这些数据集你也尝试一下吧
我觉得蛮有意义的。
GSE1462
GSE18732
GSE20950
GSE21785
GSE26526
GSE32575
GSE43837
GSE474
GSE58979
GSE60291
GSE62832
GSE70529
GSE72158
可以使用我们的4大R包啦
很方便的下载数据
rm(list = ls())
options(stringsAsFactors = F)
library(GEOmirror)
geoChina('GSE21785')
load('GSE21785_eSet.Rdata')
exp <- exprs(gset[[1]])
exp[1:4,1:4]
pd <- pData(gset[[1]])
anno = gset[[1]]@annotation
group_list =c(rep("Tubulus",6),rep("Glomerulus",6))
group_list=factor(group_list,levels = c("Tubulus","Glomerulus"))
boxplot(exp,las=2,col=group_list)
差异分析,火山图,热图,基因注释看以前的教程吧。