生物信息学数据库种类繁多,其中基因ID是很多人比较困惑的,尤其是很多产品居然还不是基因ID的问题,比如表达芯片是探针,所以我策划了一系列ID转换教程,见文末!我的包里面有一个函数大家比较感兴趣,就是为什么可以根据基因ID拿到其染色体坐标呢?而且还可以得到其基因类型。
如下所示:
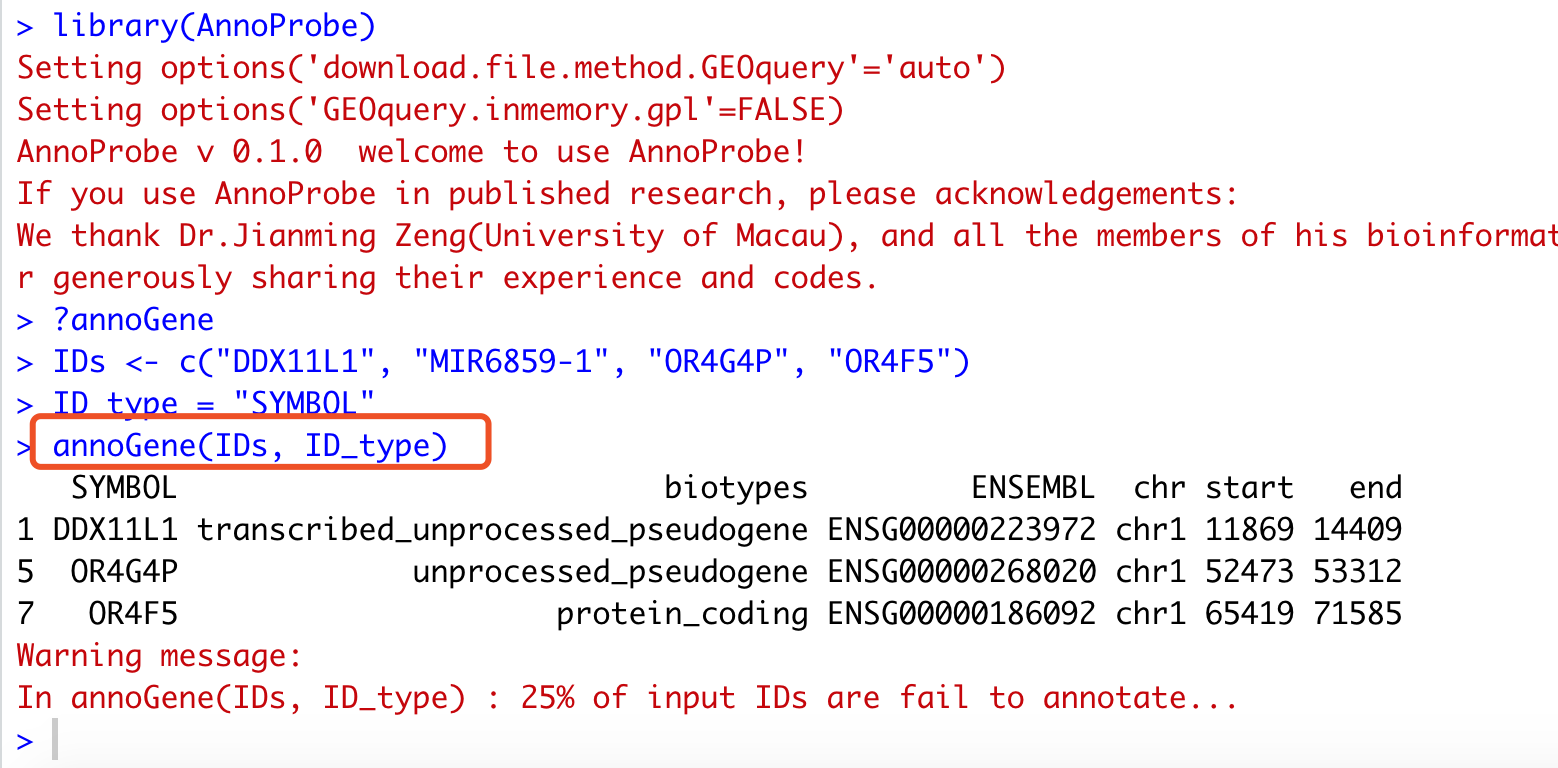
library(AnnoProbe)
IDs <- c("DDX11L1", "MIR6859-1", "OR4G4P", "OR4F5")
ID_type = "SYMBOL"
annoGene(IDs, ID_type)
annoGene(IDs, ID_type,out_file ='tmp.html')
annoGene(IDs, ID_type,out_file ='tmp.csv')
你可以指定ID_type,目前只能是选择 “ENSEMBL” or “SYMBOL”,然后这个函数就会为你进行ID转换及坐标,还有基因类型的注释。
需要使用下面的代码自行下载安装我们的AnnoProbe包
library(devtools)
install_github("jmzeng1314/AnnoProbe")
library(AnnoProbe)
因为这个包里面并没有加入很多数据,所以理论上会比较容易安装,当然,不排除中国大陆少部分地方基本上连GitHub都无法访问。
我做了哪些事情
其实就是下载GENCODE数据库文件,然后格式化即可。我这里使用的是perl语言,实际上可能是使用R更好,保证这个开发流程的统一。
# https://www.gencodegenes.org/human/
nohup wget ftp://ftp.ebi.ac.uk/pub/databases/gencode/Gencode_human/release_32/gencode.v32.annotation.gtf.gz &
# https://www.gencodegenes.org/mouse/
nohup wget ftp://ftp.ebi.ac.uk/pub/databases/gencode/Gencode_mouse/release_M23/gencode.vM23.annotation.gtf.gz &
# https://www.ensembl.org/Rattus_norvegicus/Info/Index
nohup wget ftp://ftp.ensembl.org/pub/release-98/gtf/rattus_norvegicus/Rattus_norvegicus.Rnor_6.0.98.chr.gtf.gz &
zcat gencode.v32.annotation.gtf.gz |perl -alne '{next unless $F[1] eq "HAVANA";next unless $F[2] eq "gene";/gene_id \"(.*?)\.\d+.*?\"; gene_type \"(.*?)\"; gene_name \"(.*?)\"/;print "$3\t$2\t$1\t$F[0]\t$F[3]\t$F[4]"}' > human_gencode.v32.txt
zcat gencode.vM23.annotation.gtf.gz |perl -alne '{next unless $F[1] eq "HAVANA";next unless $F[2] eq "gene";/gene_id \"(.*?)\.\d+.*?\"; gene_type \"(.*?)\"; gene_name \"(.*?)\"/;print "$3\t$2\t$1\t$F[0]\t$F[3]\t$F[4]"}' > mouse_gencode.vM23.txt
zcat Rattus_norvegicus.Rnor_6.0.98.chr.gtf.gz |perl -alne '{next unless $F[2] eq "gene";/gene_id \"(.*?)\";.*?gene_name \"(.*?)\";.*?gene_biotype \"(.*?)\";/;print "$2\t$3\t$1\t$F[0]\t$F[3]\t$F[4]"}' > rat_ensembl_6.0.98.txt
得到的文件如下:
53004 human_gencode.v32.txt
48961 mouse_gencode.vM23.txt
32623 rat_ensembl_6.0.98.txt
这些就加载到R包里面,有了这些数据源,就可以很轻松的进行基因ID转换或者注释了。
表达芯片探针ID转换大全
在2019年的尾巴,我推出3个R包,
- 第一个是整合全部的bioconductor里面的芯片探针注释包。
- 第二个是整合全部GPL的soft文件里面的芯片探针注释包。
- 第三个是下载全部的GPL的soft文件里面的探针碱基序列比对后注释包。
配合着详细的介绍:
因为这些包暂时托管在GitHub平台,但是非常多的朋友访问GitHub困难,尤其是我打包了好几百个GPL平台的注释信息后, 我的GitHub包变得非常臃肿,大家下载安装困难,所以我重新写一个精简包,也在:芯片探针ID的基因注释以前很麻烦 和 :芯片探针序列的基因注释已经无需你自己亲自做了, 里面详细介绍了。最重要的是idmap函数,安装方法说到过:芯片探针序列的基因注释已经无需你自己亲自做了, 使用起来也非常简单:
library(AnnoProbe)
ids=idmap('GPL570',type = 'soft')
head(ids)
仅仅是一句话,就拿到了这个平台的探针的注释信息。需要注意的是,这个函数的type参数,其实是有3个选择,这里我演示的是选择soft这个来源的基因注释信息。
并不是所有的平台都是有soft注释,也不是所有的平台都被我的这个工具囊括哦。