前面我们介绍该软件用法很多了,而且也对基于bam文件做可变剪切的软件leafcutter和rMATS的比较
现在我们来根据MFF基因来深度理解可变剪切的软件leafcutter的结果!
首先是差异分析结果
$grep MFF leafcutter_ds_cluster_significance.txt
chr2:clu_4932_NA Success 4.6012973874349 7 0.238436066745941 0.634806885002018 MFF
chr2:clu_4933_NA Success 81.7113181603927 15 5.08675778183616e-27 1.39918539586235e-23 MFF
cluster status loglr df p p.adjust genes
需要大家仔细理解的两列就是loglr和df
其中df就是自由度,就是该转录本的外显子的数量减去1,其实就是内含子的个数啦!
各个样本的表达
单独查看每个转录本在每个样本的内含子的表达量如下:
首先看看control组的一个样本
$zcat SRR2016934.junc.tumor.sorted.gz |grep clu_4932_NA
chr2:227325427:227328678:clu_4932_NA 32/320
chr2:227325427:227330626:clu_4932_NA 13/320
chr2:227325427:227332419:clu_4932_NA 9/320
chr2:227325542:227328678:clu_4932_NA 1/320
chr2:227328789:227329606:clu_4932_NA 12/320
chr2:227328789:227330626:clu_4932_NA 106/320
chr2:227329783:227330626:clu_4932_NA 12/320
chr2:227330846:227332419:clu_4932_NA 135/320
再看看tumor组的一个样本:
$zcat SRR2016947.junc.tumor.sorted.gz |grep clu_4932_NA
chr2:227325427:227328678:clu_4932_NA 42/400
chr2:227325427:227330626:clu_4932_NA 2/400
chr2:227325427:227332419:clu_4932_NA 8/400
chr2:227325542:227328678:clu_4932_NA 2/400
chr2:227328789:227329606:clu_4932_NA 13/400
chr2:227328789:227330626:clu_4932_NA 112/400
chr2:227329783:227330626:clu_4932_NA 19/400
chr2:227330846:227332419:clu_4932_NA 202/400
综合的表达量矩阵
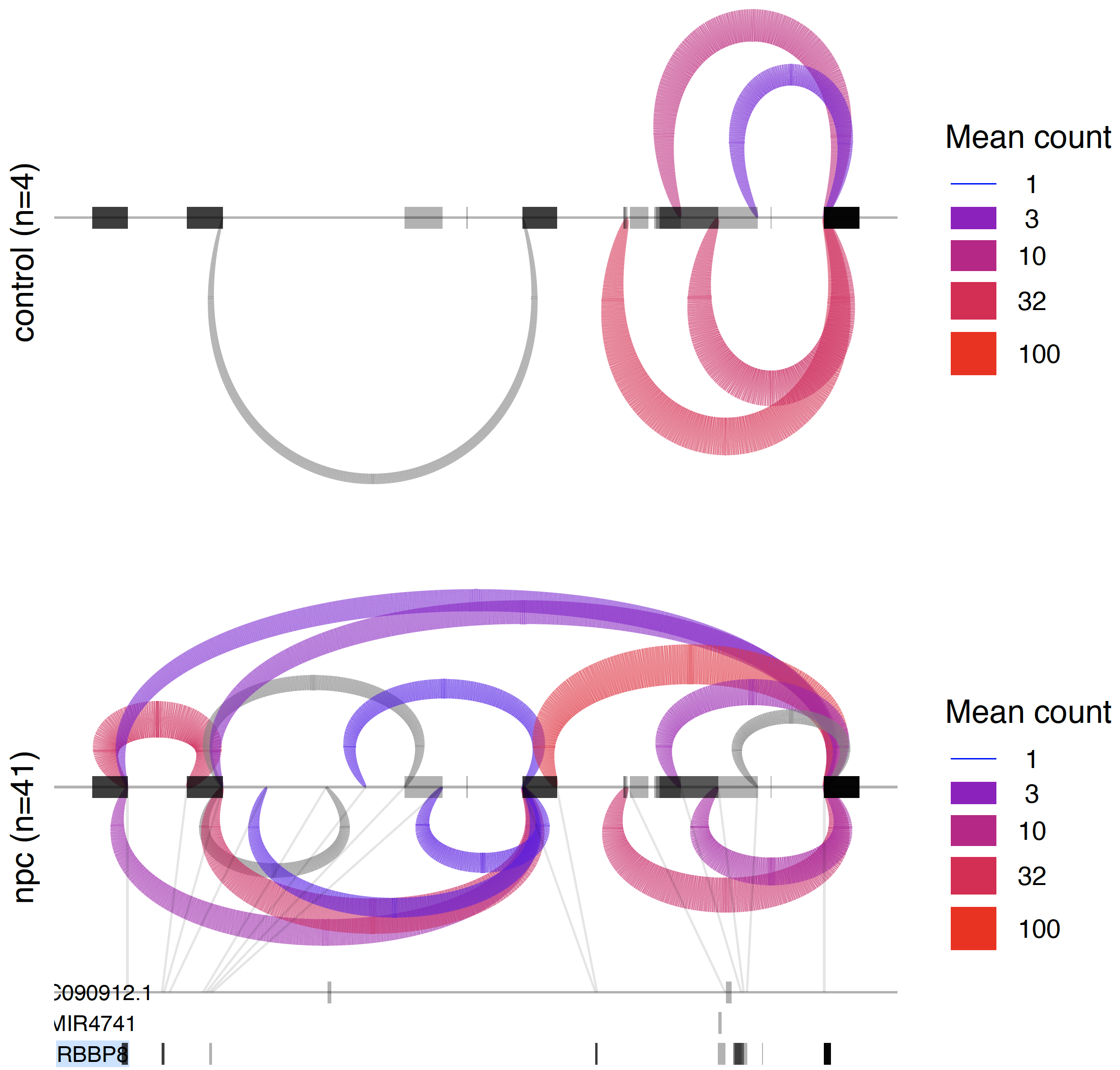
再看看合并的表达矩阵的热图,我选择的这个例子并不是很好,因为两个分组的样本数量严重失衡,但是根据这样的分组,也是可以很明显看到一个可变剪切示例:
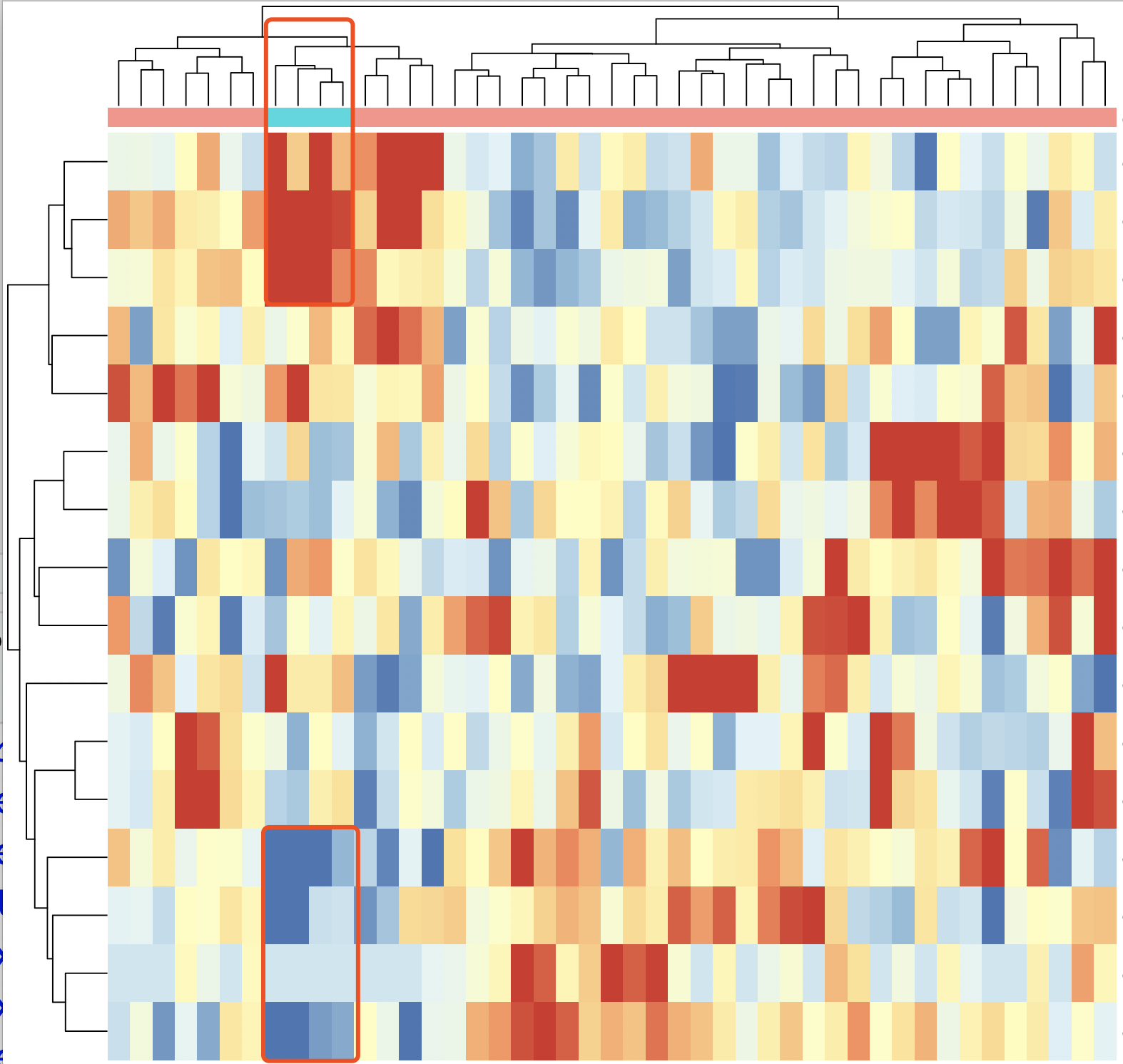
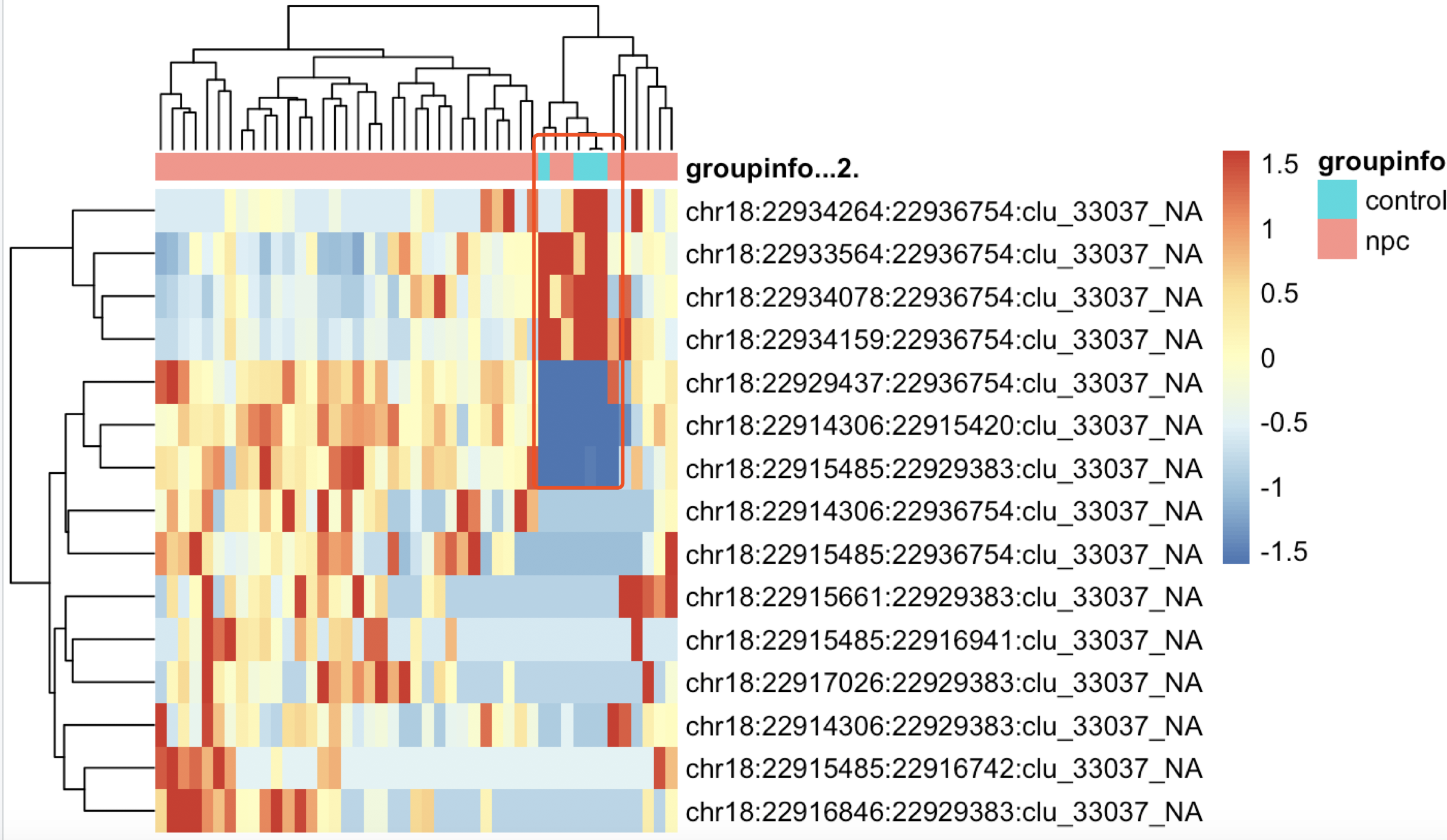
当然, 我们也可以选择另外一个更显著的基因,即loglr更大的,比如RBBP8,如下:
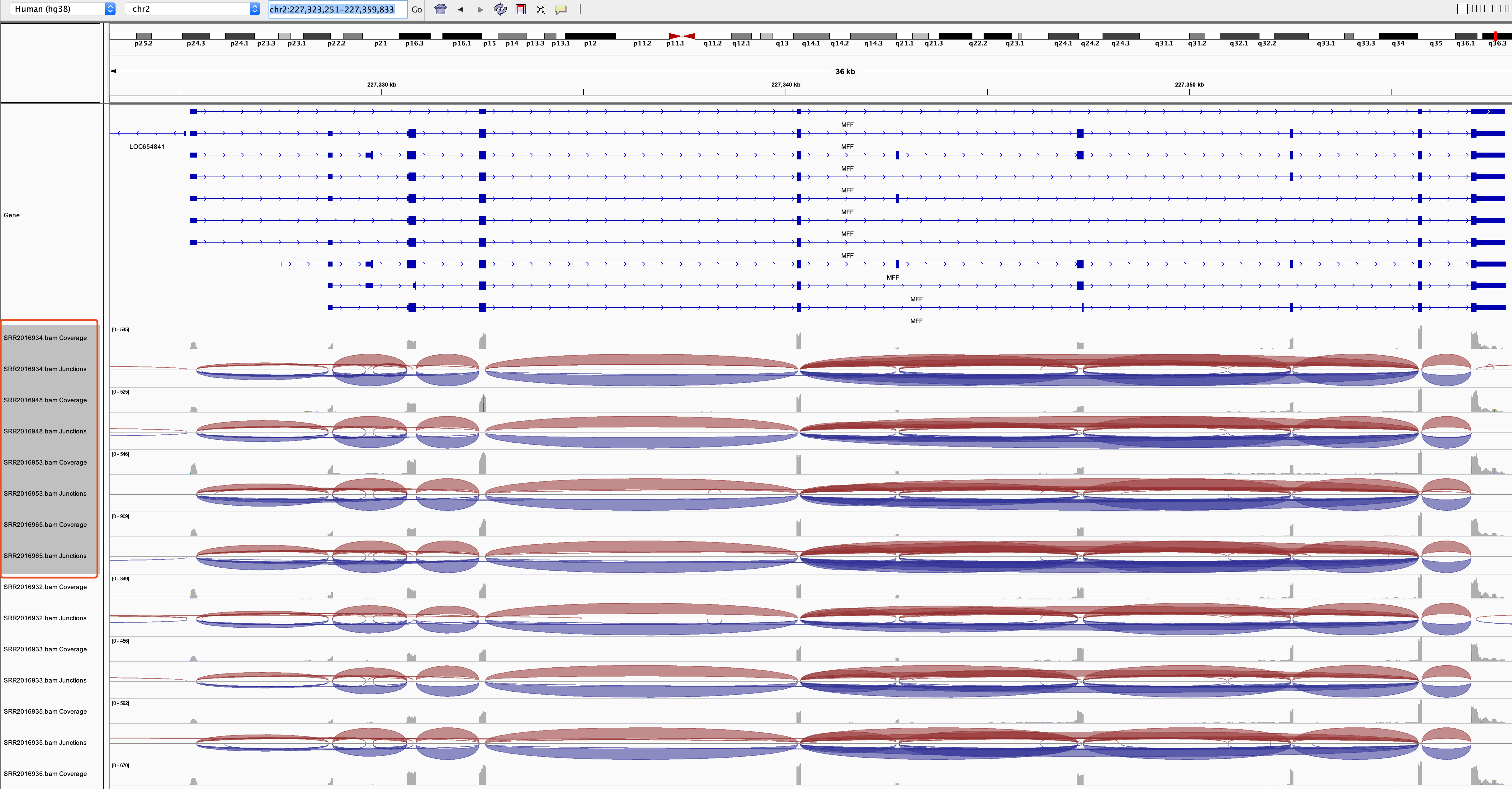
IGV查看
首先我们看看MFF基因,不是很好看出来这个差异。
再看看RBBP8基因,实际上也是有点难看出来差异:
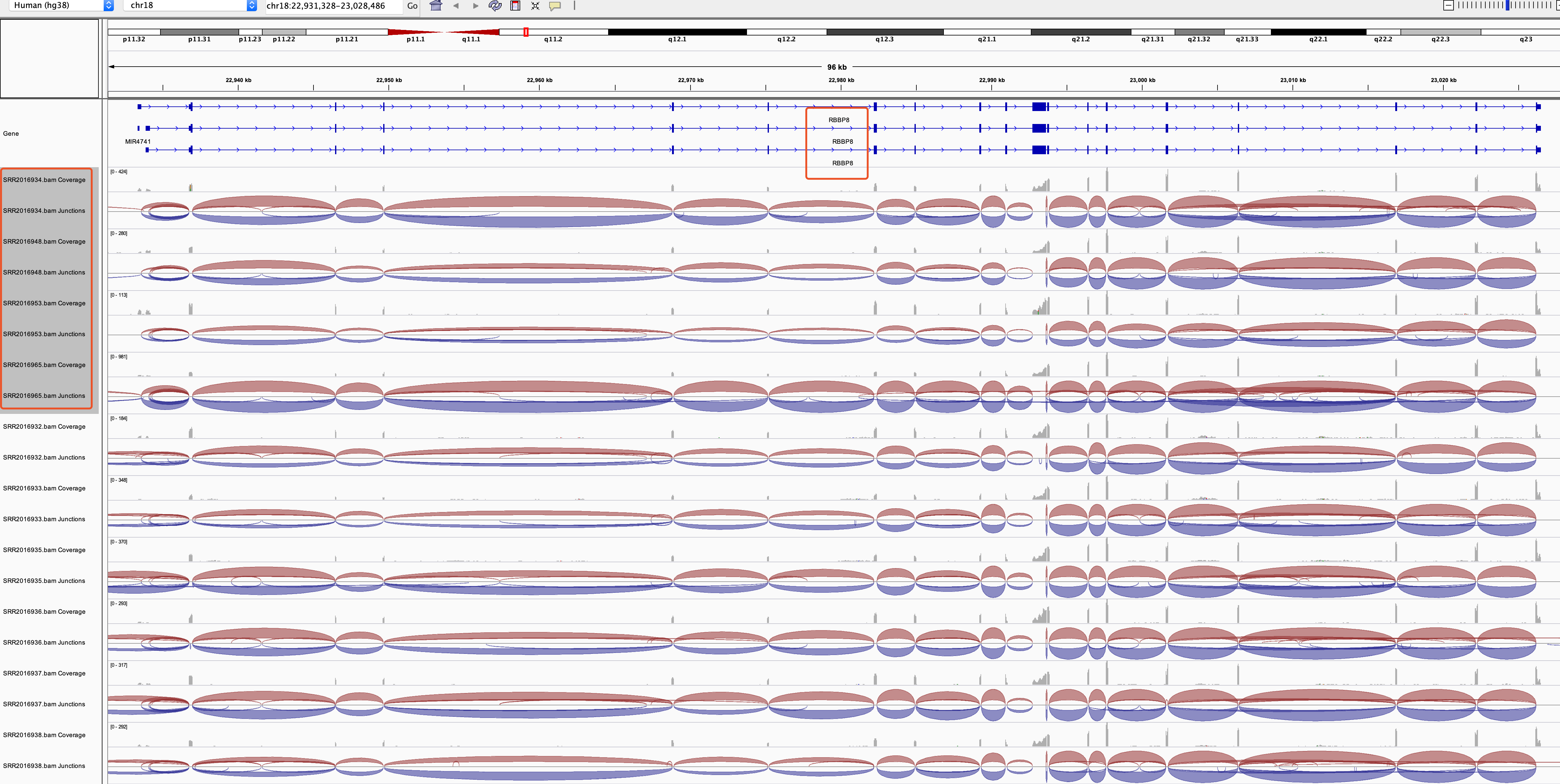
但是可变剪切的软件leafcutter本身的可视化就很明显: