在整个生信技能树的历史上,就分享过两次价值一千元的:
- 第一次是:TCGA的28篇教程-风险因子关联图-一个价值1000但是迟到的答案
- 第二次是:(重磅!价值一千元的R代码送给你)芯片探针序列的基因组注释
其中第二个教程是纯粹的R代码技巧,怕粉丝看不懂,我还刻意花了一个星期做铺垫: - 1 把fasta序列读入到R里面去
- 2 使用refGenome加上dplyr玩转gtf文件
- 3 把bam文件读入R,并且转为grange对象
- 4 在R里面使用Rsubread完成组学分析全套流程
- 5 在R里面对坐标进行映射
- 6 下载所有芯片探针序列并且写成fasta文件
有两个弊端
根据粉丝的反馈,是有两个问题的,首先是该R包在Windows平台是无法使用的,然后是大家下载参考基因组总是搞错!
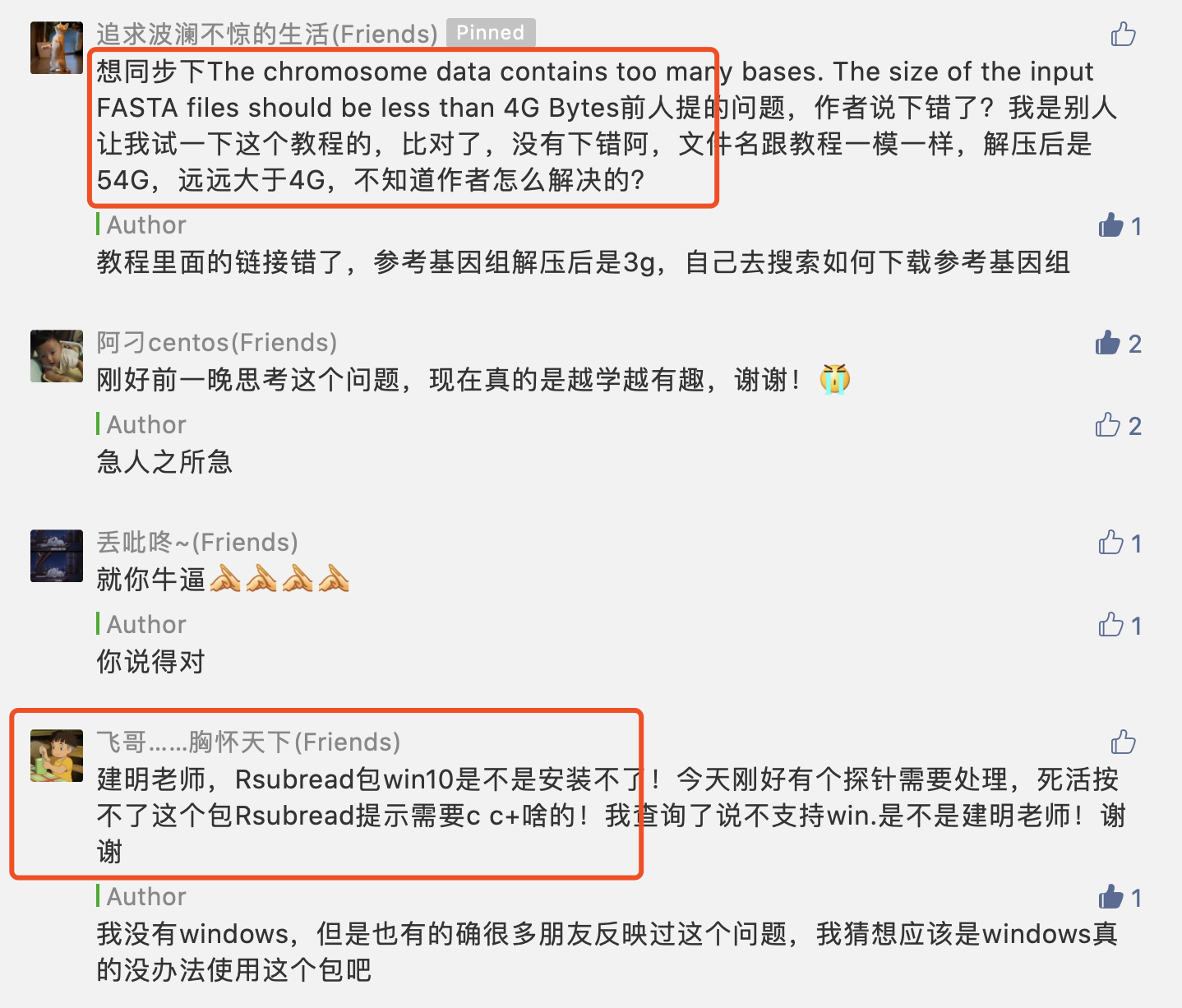
还专门有粉丝发邮件求助,问我为什么,他跟着我的教程:(重磅!价值一千元的R代码送给你)芯片探针序列的基因组注释 报错:ERROR: the provided reference sequences include more than 4 billion bases 初步怀疑是电脑配置不够,就升级到了96GB内存,20核心CPU,1.2T硬盘,但是报错依旧!
所以我就让他指明是哪一个步骤代码问题,结果他告诉我下载的1G参考基因组解压后是54G,我的天!
我代码中说的数据库:’Homo_sapiens.GRCh38.dna.toplevel.fa‘ 因为并没有给出下载的链接,所以导致初学者只能是自己折腾碰壁了,但是正常的生物学背景知识朋友都应该是知道人类参考基因组是3G左右啊!如果你下载的是toplevel版本的基因组: Homo_sapiens.GRCh38.dna.toplevel.fa.gz,文件大小1G,解压后54G!!!实际上用它对应的primary版本就够了:Homo_sapiens.GRCh38.dna.primary_assembly.fa.gzprimary的版本中是不包括haplotype info的,而top level中会包含大量的变异信息,而这部分是很冗余并且一般也用不太到。其实你可以使用我们的AnnoProbe包
目前仍然是 host 在GitHub上面:https://github.com/jmzeng1314/annoprobe
如果大家觉得有帮助,后续我会考虑抽时间去发布在bioconductor里面,甚至写成SCI文章供大家引用。
以前大家是需要自己下载探针序列进行参考基因组比对后注释,比如我在 (重磅!价值一千元的R代码送给你)芯片探针序列的基因组注释 提到的例子;关于Human LncRNA Expression Array V4.0 AS-LNC-H-V4.0 20,730 mRNAs and 40,173 LncRNAs 8*60K这个芯片探针的重新注释,一般文献里面的描述是:
- probe sequences 探针序列下载
- uniquely mapped to the human genome (hg19) by Bowtie without mismatch. 参考基因组下载及比对
- chromosomal position of lncRNA genes based on annotations from GENCODE (Release 23)坐标提取,最后使用bedtools进行坐标映射
但是大部分人是没有linux操作能力,无法完成这个流程,使用我们的包可以轻轻松松达到探针注释的目的!首先下载安装我们的AnnoProbe包
library(devtools) install_github("jmzeng1314/AnnoProbe") library(AnnoProbe)因为这个包里面并没有加入很多数据,所以理论上会比较容易安装,当然,不排除中国大陆少部分地方基本上连GitHub都无法访问。
然后使用AnnoProbe包获取探针注释信息
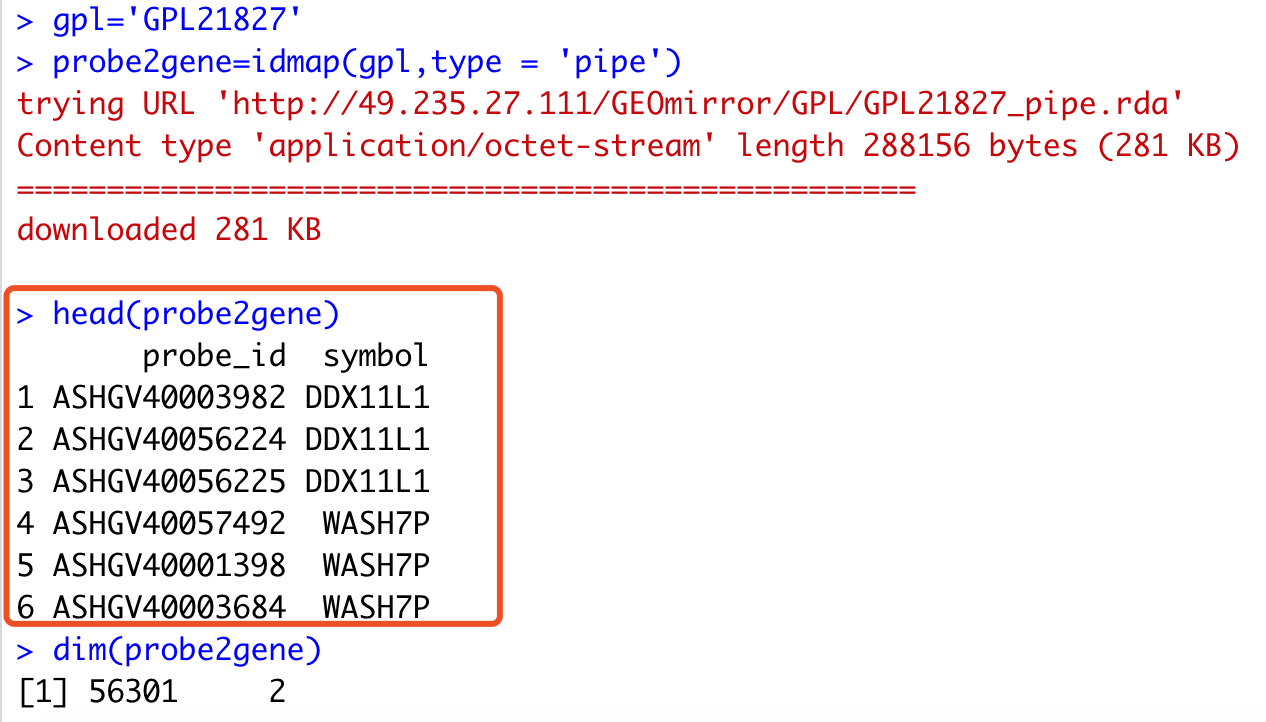
# GPL21827[Accession] - GEO DataSets Result - NCBI - NIH # https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GPL21827 gpl='GPL21827' probe2gene=idmap(gpl,type = 'pipe') head(probe2gene)轻轻松松的几行代码,就拿到了探针的注释信息哦
是不是很激动?