外显子技术是仅次于转录组的热门 NGS 应用,尤其是在肿瘤研究方向,大量的癌症多组学队列其实就是转录组加上外显子而已。实际上并没有专门的肿瘤转录组教程系列,但是肿瘤外显子却不然,如果大家三年前追过我的直播基因组活动,就应该知道同样是DNA层面的测序,全基因组,外显子组还有捕获基因靶向测序,在肿瘤研究里面不仅仅是找跟参考基因组不一样的位点,就是所谓的变异位点而已。
肿瘤外显子实验设计里面通常是对一个病人既测序其肿瘤组织又测序其正常组织(癌旁或者血液),这样的话,分析流程里面就需要分别独立比对到参考基因组,然后筛选出那些出现在肿瘤组织里面却并没有出现在同一个病人组织里面的那些突变,就是我们所说的体细胞突变。虽然,目前我的B站74小时视频并没有肿瘤外显子视频教学课程,但是已有的WES视频教学教程,加上我这几年在生信技能树陆陆续续写的肿瘤相关教程,还有菜鸟团的肿瘤外显子数据处理系列教程,目前整理到了https://www.yuque.com/biotrainee/wes 知识库,已经足够大家学会啦。为此,我奉上习题一套,大家如果做完这个小作业没有问题就说明大家掌握了基础的肿瘤外显子分析流程了哦。
step1:读文献
文章:A Targetable EGFR-Dependent Tumor-Initiating Program in Breast Cancer
自行搜索了解一些背景知识:
- epidermalgrowth factor receptor (EGFR)
- EGFR inhibition by gefitinib
- triple-negative breast cancer (TNBC)
- patient-derived xenografts (PDXs)
- Deep single-cell RNAsequencing of 3,500 cells
主要是关注实验设计
作者制作了一批TNBCs (成功率15/18)的PDX模型,然后用这些模型来测试其对 EGFR inhibitor gefitinib 敏感情况。前人报道该药物在TNBC病人里面有效率是38.7%,与他们的实验想符合(6/18), 但是其中有一个人的反映比较特殊,就是 GCRC1735, 一个70岁的老奶奶,该药物治疗效果出奇的好。 所以就对这个老奶奶的肿瘤组织进行一系列的NGS探索。基因检测表明该老奶奶有一个 pathogenic BRCA1 mutation (p.C1225Sfs) 和a somatic TP53 alteration (p.R249T) ,而EGFR基因上面既没有突变也没有拷贝数变异,EGFR 这个通路相关的基因也没有太大的异常。step2:查看测序数据
数据都在SRA数据库里面, https://trace.ncbi.nlm.nih.gov/Traces/study/?acc=SRP100090 (bulk的外显子和转录组测序),我们这个题目仅仅是关心肿瘤外显子数据,如下:
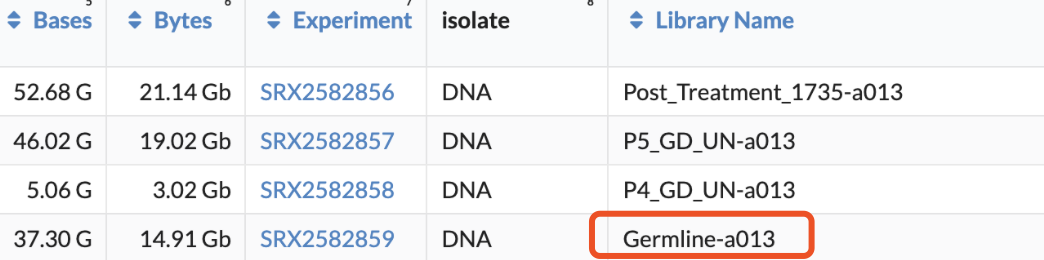
在SRA数据库下载比较慢,建议直接去EBI数据库搜索并且下载即可。step3:构建肿瘤外显子流程环境
主要是相关软件和数据库,其中软件可以conda进行管理,GATK建议最新版,数据库的话,下载会非常耗费时间。建议大家自行看视频,慢慢学咯: https://space.bilibili.com/338686099/channel/detail?cid=94251
step4:跑SNV和CNV流程
基本上参考我们菜鸟团的肿瘤外显子数据处理系列教程即可,可以在https://www.yuque.com/biotrainee/wes 知识库查看,或者看下面的目录:
- 肿瘤外显子数据处理系列教程(一)读文献并且下载测序数据
- 肿瘤外显子数据处理系列教程(二)质控与去接头
- 肿瘤外显子数据处理系列教程(三)比对
- 肿瘤外显子数据处理系列教程(四)比对结果的质控
- 肿瘤外显子数据处理系列教程(番外篇)bam文件载入igv可视化
- 肿瘤外显子数据处理系列教程(五)GATK的最佳实践
- 肿瘤外显子数据处理系列教程(六)vcf文件的注释及ANNOVAR的使用
- 肿瘤外显子数据处理系列教程(七)maftools可视化
- 肿瘤外显子数据处理系列教程(八)不同注释软件的比较(上):安装及使用
- 肿瘤外显子数据处理系列教程(八)不同注释软件的比较(中):注释后转成maf文件
- 肿瘤外显子数据处理系列教程(八)不同注释软件的比较(下):可视化比较maf文件
- 肿瘤外显子数据处理系列教程(九)拷贝数变异分析(主要是GATK)
- 肿瘤外显子数据处理系列教程(九)拷贝数变异分析(不同软件的比较)
step5:重复出来SNV表格和IGV截图
文章附件有SNV列表,如下:
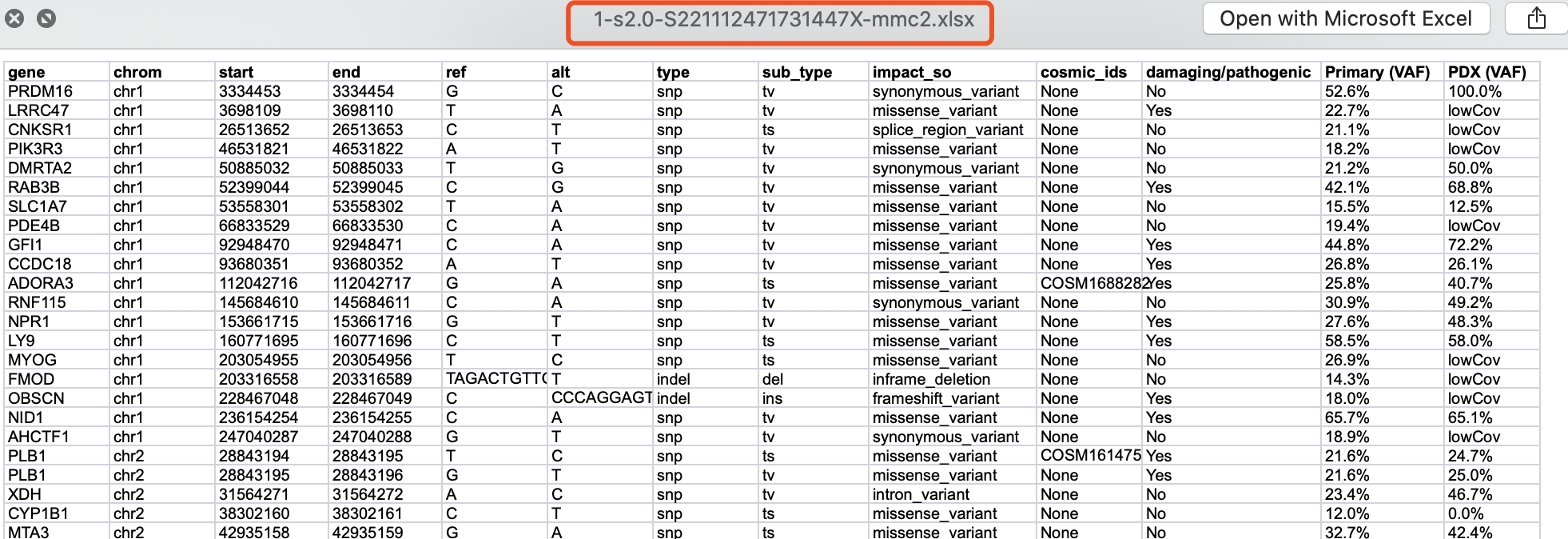
然后其中一个IGV截图需要大家复现:
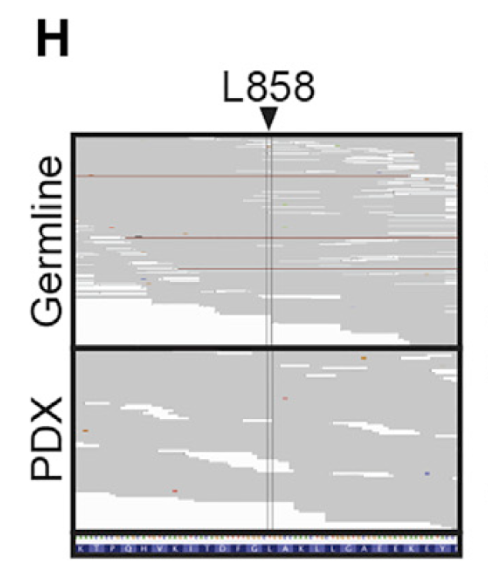
step6:看具体区域的CNV情况
同样的,查看指定区域的CNV情况,如下:
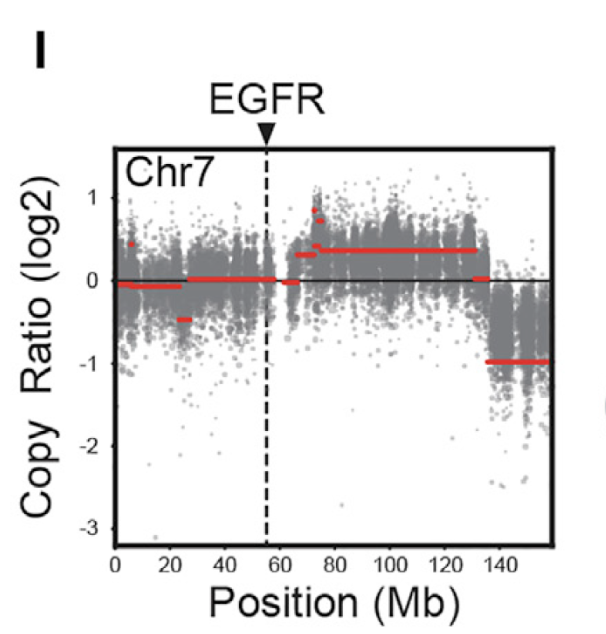
step7:SNV和CNV的基因列表和EGFR通路基因取交集
如下所示:
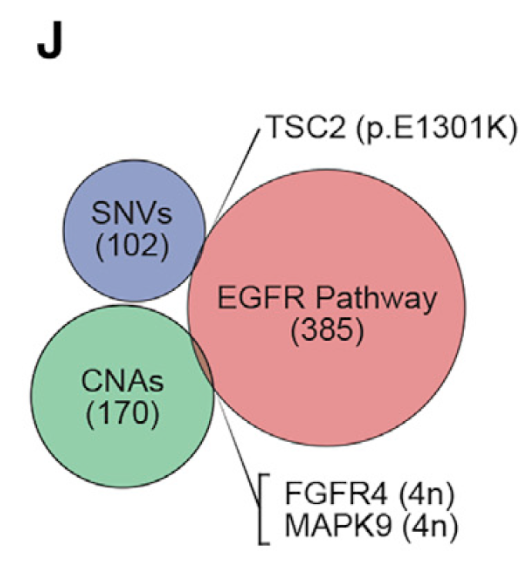
step8:肿瘤组织的SNV和PDX模型的SNV的VAF散点图
在文章附件里面有SNV列表,所以可以直接导入R里面自行绘制散点图,如下:
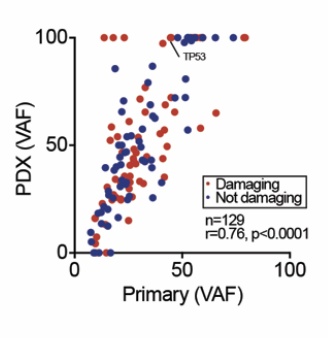
当然了,我们的作业是要求大家自行下载fq测序数据后走肿瘤外显子流程,然后对自己得到的SNV列表进行绘制上面的肿瘤组织的SNV和PDX模型的SNV的VAF散点图!你会发现这个作业跟你从公司拿到的肿瘤外显子报告差异很大
一、质控(fastqc +tookit)
1数据质量:
- 1)碱基质量分布
- 2)reads质量分布
- 3)reads长度分布
- 4)GC含量
2数据过滤 - 1)原始reads数
- 2)平均质量值>Q20 reads数目和比例
- 3)平均质量值>Q30 reads数目和比例
- 4)过滤掉reads中碱基质量<Q20的碱基占比超过5%的reads。统计clean data的reads和比例。
二、比对(bwa)
1)比对上基因组的reads数及占总数的比例
2)完全匹配的reads数
3)匹配上各个染色体的reads数
4)染色体上的覆盖深度
5)落在目标区域(exon)的reads数
6)落在目标区域+-100的reads数
7)目标区域碱基覆盖深度
8)目标区域碱基被覆盖比例
9)目标区域碱基被覆盖(50X,100X,150X,200X。。。)的比例三、find SNV(samtools +picard+gatk+varscan)
1)picard :sam >sort.bam
2)gatk :sort.bam >sort.dedup.bam (去重复)
3)gatk :sort.dedup.bam > realign.bam (重新比对,indel和snp校正)
4)Gatk :碱基质量重打分。(未进行)
5)Varscan :call SNV四、突变注释
1)annovar注释。
2)注释结果统计(同义,非同义突变,基因上下游,内含子,外显子上等)
3)dbsnp 注释(找到的snp是否在dbsnp数据库上)
4) cosmic63 :癌症相关突变五、突变分析
1)snv在个染色体上的分布
2)各基因上snv的分布
3)Snv位点较多的基因进行功能分析(pathway,kegg的通路分析和Go功能富集)