也许是一种缘分吧,现在我的视频教程里面,居然是表观调控相关内容最多,本来就有ChIP-seq数据处理和ATAC了,而且前两天还给大家发了表观调控整合教学视频:
- 视频首先可以在B站看到:https://www.bilibili.com/video/av77098514 (失效日期随时),请尽快学习
- 如果你需要离线查看也可以选择微云下载:https://share.weiyun.com/5sEh0m2(失效日期随时),请尽快保存
- 视频课程配套课件(幕布思维导图):https://mubu.com/doc/3Bd4aieYug
- 配套代码目前也是微云下载:https://share.weiyun.com/5qmxu7Z(失效日期随时),请尽快保存
基本上每个过来我这边学习一个月以上的学徒我都会让他们学习多种组学(围绕着中心法则),而且有了Linux基础和R语言能力后, 跟着我们的视频教程很容易就学会基础流程,毫无压力。 - 给学徒ChIP-seq数据处理流程(附赠长达5小时的视频指导)
- 九月学徒ChIP-seq学习成果展(6万字总结)(上篇)
- 九月学徒ChIP-seq学习成果展(6万字总结)(下篇)
step1:读文献
文章是:Enhancer invasion shapes MYCN-dependent transcriptional amplification in neuroblastoma 发表于2018年01月的NG杂志。需要自行搜索学习一些背景知识:
-
增强子:是DNA上一小段可与蛋白质(反式作用因子:trans-acting factor)结合的区域,与蛋白质结合之后,基因的转录作用将会加强。
-
启动子: RNA 聚合酶识别、结合和开始转录的一段 DNA 序列,它含有 RNA 聚合酶特异性结合和转录起始所需的保守序列,一般位于转录起始位点的上游。
-
神经母细胞瘤:儿童最常见的颅外实体肿瘤,主要起源于交感神经系统,发生在身体各个部位。
-
编码致癌转录因子 MYCN 的基因座的扩增是高危神经母细胞瘤的定义特征。
MYC 转录因子家族,包括 MYC,MYCN 和 MYCL,包括癌症中最常改变的癌基因,这些蛋白质通常充当细胞生长信号的整合者并介导涉及多种过程的转录应答,这些过程包括增殖,细胞生长,分化,存活和多能性 (proliferation, cell growth, differentiation, survival and pluripotency)。step2:查看测序数据
这篇文章使用的NGS技术和数据集非常之多,作为ChIPseq视频课程小作业,我们无需完全弄懂它,只需要处理简单的几个数据,然后绘制几个图表即可。
-
- GSE80149 , [ARRAY]
- GSE80150 , [ChIP_RX]
- GSE80151 , [ChIP-seq]
- GSE80152 , [ATAC-seq]
- GSE80153 , [RNA-seq]
- GSE100538 , [ChIP-seq Th-MYCN]
我这里布置的作业仅仅是两个IP和一个INPUT,就3个fastq测序数据而已: - GSM2113520 BE2C_INPUT
- GSM2113521 BE2C_MYCN
- GSM2113523 BE2C_TWIST
大家需要搜索拿到其EBI数据库的fastq文件地址,比如首先 https://www.ebi.ac.uk/ena,比如搜索 SRR3356385
就可以得到:https://www.ebi.ac.uk/ena/data/view/SRR3356385
里面就有下载地址:ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR335/005/SRR3356385/SRR3356385.fastq.gzstep3:构建ChIPseq数据分析流程
看视频课程 https://www.bilibili.com/video/av29255401 ,或者我们生信技能树的公众号推文均可:
- 给学徒ChIP-seq数据处理流程(附赠长达5小时的视频指导)
- 九月学徒ChIP-seq学习成果展(6万字总结)(上篇)
- 九月学徒ChIP-seq学习成果展(6万字总结)(下篇)
需要使用Linux环境,用conda配置好软件环境,走数据分析基本流程。step4:两个ChIPseq数据取交集
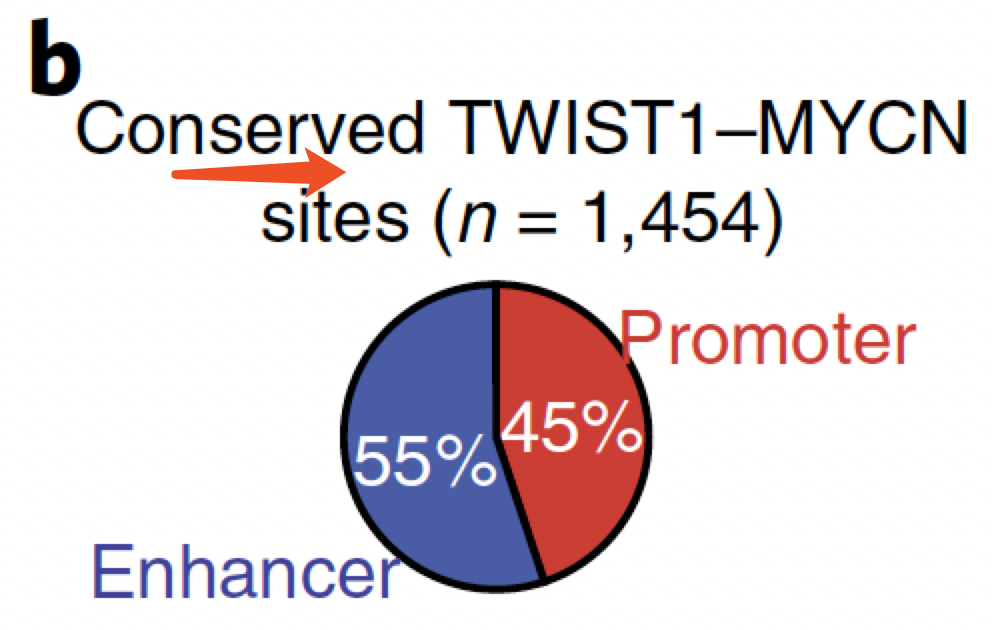
前面我们下载了3个数据,是两个IP和一个INPUT,所以两个IP都是会有bed格式的peaks文件,简单的取交集就是图b, Pie chart showing the genomic distribution of overlapping TWIST1- and MYCN-bound sites.
其中把TWIST1- and MYCN共有的peaks分类成为 enhancer和promoter 第五步的事情啦。
step5:定义enhancer区域
把TWIST1- and MYCN共有的peaks分类成为 enhancer和promoter,这个需要看文献,其中promoter很简单,就是看peaks是否坐落于基因的转录起始位点上下游的2kb区域内。
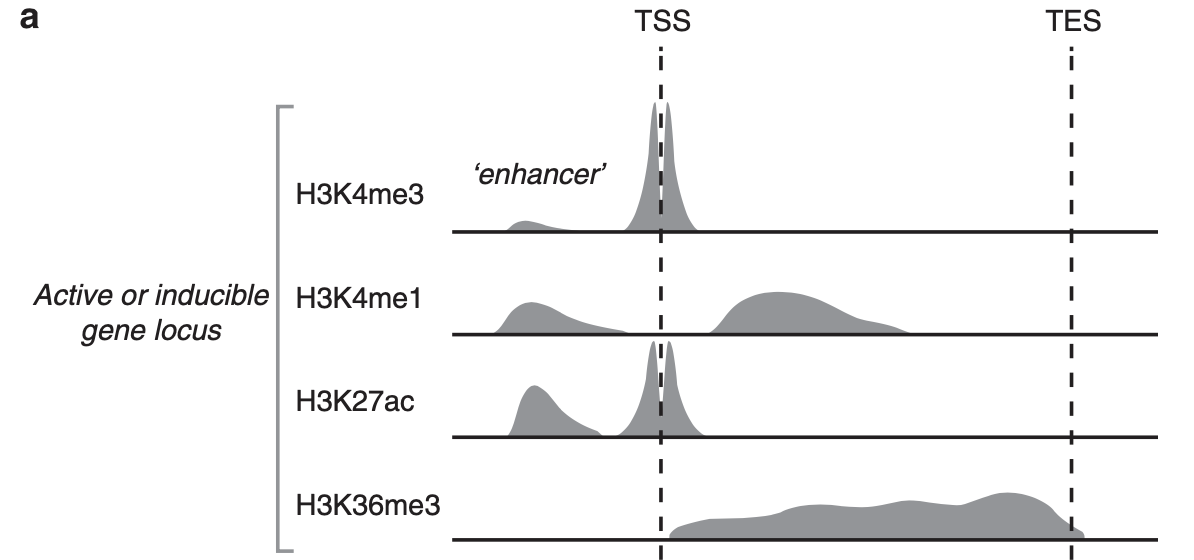
但是enhancer需要借助这篇文章的其它数据来辅助定义了。GSM2113517 BE2C_BRD4 GSM2113518 BE2C_H3K27AC GSM2113519 BE2C_H3K4ME3 GSM2113520 BE2C_INPUT GSM2113521 BE2C_MYCN GSM2113522 BE2C_RNA_POL2 GSM2113523 BE2C_TWIST就是H3K27AC和H3K4ME3的数据也是需要下载,走流程拿到peaks的。
step6:对peaks区域找motif
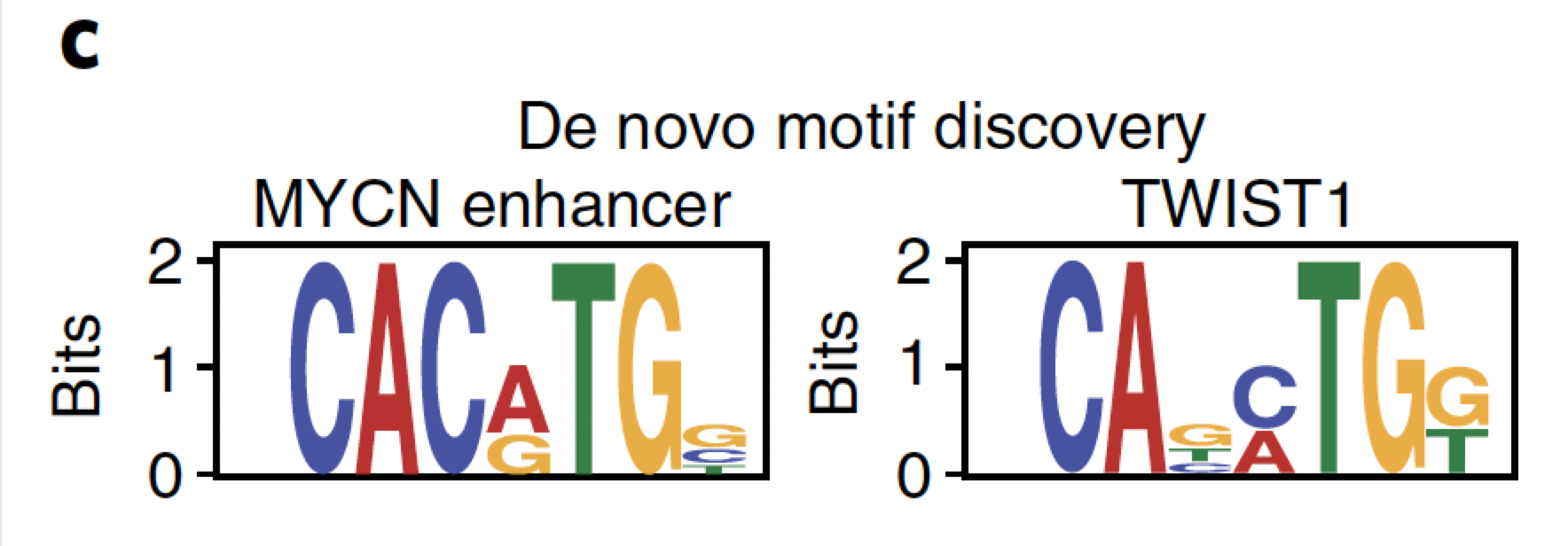
需要查看对peaks区域找motif的教程 ,对上面分成为 enhancer和promoter的peaks分别去寻找motif,绘制图c, De novo motif analysis of enhancer regions for MYCN- and TWIST1-binding sites
当然了,大概率上你运行我们的视频课程的软件代码得到的图表都很初步,如果需要让它变成发表级别质量,需要学一学AI等科研绘图技术,我们明天周一会推出一个这样的科研绘图精品课程!