虽然我一直讲解的GEO数据挖掘,都是基于mRNA这样的表达芯片,但实际上miRNA,lncRNA,甚至circRNA芯片也是大同小异的分析流程。
所以,如果你确实是第一次接触circRNA芯片数据,完全不用担心, 你只需要把它看作是你不知道基因名字的mRNA芯片,最后得到的各种各样的分析结果,只不过是以circRNA的ID来表示而已。比如:
- 中南大学湘雅二医院陈林老师实验室通过Arraystar CircRNA芯片发现一种调控口腔鳞状细胞癌发生发展的circRNA分子—circRNA_100290。
- 就是分析OSCC及NCMT样本,分析circRNA表达谱,发现共有280个circRNA表达发生显著表达,其中139个circRNA上调,141个circRNA下调,从上调最显著的10个circRNA中选出circRNA_100290, q-PCR验证circRNA_100290确实在OSCC组织中表达显著上调。
- 曹雪涛院士课题组用Arraystar CircRNA芯片研究发现circMTO1可以作为microRNA的吸附海绵结合癌基因miR-9从而上调p21的表达从而抑制肝细胞癌(hepatocellular carcinoma, HCC)的生长
- 在7例肝癌组织和7例正常肝组织中circRNA的差异表达,筛选出肝细胞癌中20个差异显著的CircRNA做聚类分析(其中10个circRNA上调,10个circRNA下调),qPCR验证了这20个circRNA与芯片结果一致。
是不是很眼熟啦!不会害怕了吧!
下面我们看看一篇真实的circRNA芯片文献吧,发表在Cancer Cell International,时间是 November 2019 ,标题是 A 3-circular RNA signature as a noninvasive biomarker for diagnosis of colorectal cancer,值得一提的是,作者并没有上传其芯片数据哦。临床课题设计的一般原则
毕竟高通量技术成本不菲,测序也好,芯片也罢,一下子对成百上千病人队列都做经济上有压力,所以通常是少数几个病人得到结果后,进一步筛选10个左右的靶基因进行后续大规模人群实验验证说明我们高通量数据分析结果的可靠性。
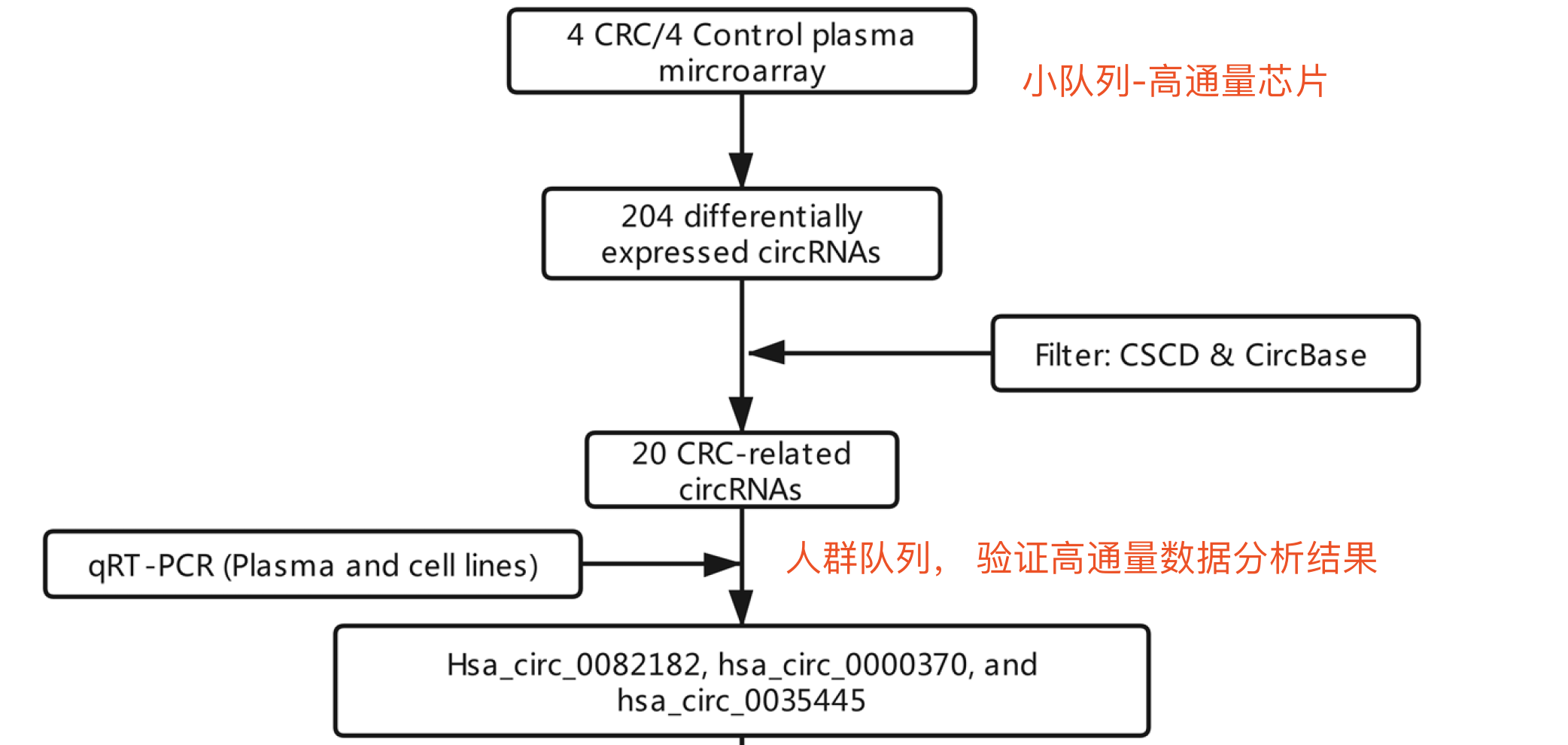
表达芯片的标准差异分析
表达芯片是目前应用最广泛的高通量技术啦,虽然大家接触的都是mRNA这样的表达芯片,但实际上miRNA,lncRNA,甚至circRNA芯片也接连成为科研热点,但是数据分析流程是没有区别的。都是走标准分析流程,火山图,热图,GO/KEGG数据库注释等等。这些流程的视频教程都在B站和GitHub了,目录如下:
- 在7例肝癌组织和7例正常肝组织中circRNA的差异表达,筛选出肝细胞癌中20个差异显著的CircRNA做聚类分析(其中10个circRNA上调,10个circRNA下调),qPCR验证了这20个circRNA与芯片结果一致。
- 第一讲:GEO,表达芯片与R
- 第二讲:从GEO下载数据得到表达量矩阵
- 第三讲:对表达量矩阵用GSEA软件做分析
- 第四讲:根据分组信息做差异分析
- 第五讲:对差异基因结果做GO/KEGG超几何分布检验富集分析
- 第六讲:指定基因分组boxplot指定基因list画热图
仅仅是最后得到的差异分子,并不是以前的mRNA后面的基因名,而是miRNA,lncRNA,甚至circRNA的ID,看起来很陌生罢了。感兴趣可以细读表达芯片的公共数据库挖掘系列推文 ; - 解读GEO数据存放规律及下载,一文就够
- 解读SRA数据库规律一文就够
- 从GEO数据库下载得到表达矩阵 一文就够
- GSEA分析一文就够(单机版+R语言版)
- 根据分组信息做差异分析- 这个一文不够的
- 差异分析得到的结果注释一文就够
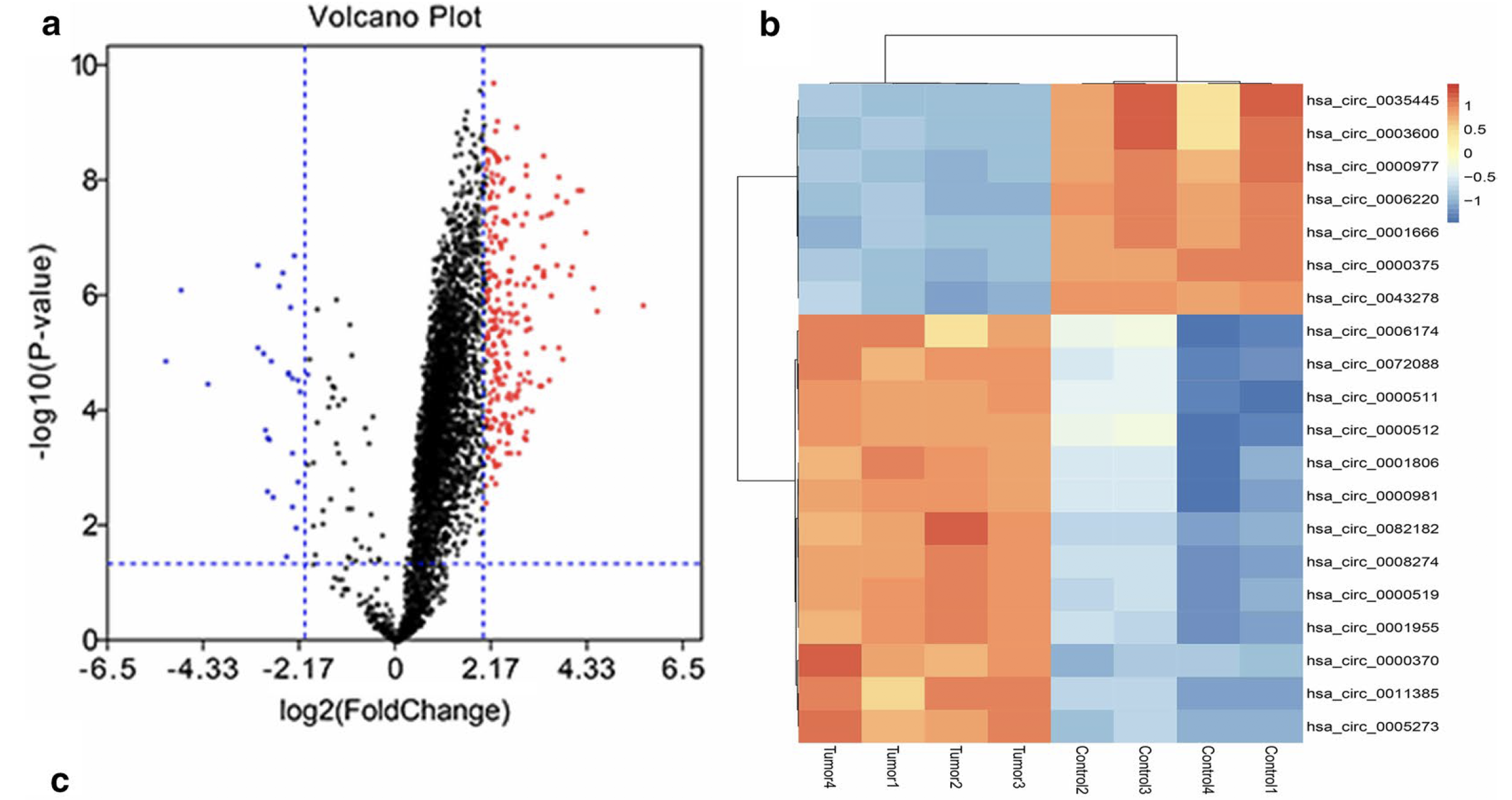
本文对于cirRNA芯片的表达矩阵分析也不例外,就是差异分析后的火山图及热图如下:
生物信息学数据分析流程
通常是描述一下所采用的芯片,然后分析方法,这个文章里面的并不是最佳分析方法。其实你看上面的火山图就看得出来,典型的半边天火山图呀,详见:惊!画出来的火山图居然缺半边?

里面提到的两个数据库非常值得大家学习。(PS:如果你有这两个数据库的介绍心得需要分享,欢迎投稿到我们生信技能树哈)我这里摘抄 doi:10.3969/j.issn.1004-5619.2018.01.014 文献里面的关于circRNA 常用数据库主要介绍 - (1)circBase[20](http://www.circbase.org),由GLAZAR等开发的一个数据库,收集并合并了大量包括人、小鼠、腔棘鱼、线虫、果蝇等成套的circRNA相关数据资料,能够浏览访问和下载相关数据资料。该数据库能够鉴定RNA测序结果中目前已知的circRNA,可以通过互联网免费访问网站数据库,但该数据库更新不够及时,所收录的基因种属种类不够多、组织来源不够全面。
- (2)Circ2Traits[21](http://gyanxet-beta.com/circdb),是一个收集与疾病或性状相关的circRNA数据库。该数据库识别与miRNA相关的疾病和circRNA的相互作用,然后计算circRNA与疾病相关的可能性。针对特定疾病,该数据库可以构建预测的miRNA与蛋白编码基因、非编码基因以及circRNA基因的相互作用的网络图。此外,数据库还能比对出疾病相关的单核苷酸多态性(single nucleotide polymorphism,SNP)在 circRNA 上的位置,然后通过Ago分析两者之间的相互作用。
- (3)deepBase[22](http://rna.sysu.edu.cn/deepBase/),是一个研究非编码RNA的综合数据库,同时收录小RNA、长非编码RNA以及circRNA等非编码RNA的数据,旨在研究深度测序发现的各种非编码RNA的表达形式与功能,包含了大约15万的circRNA基因(人、鼠、果蝇、线虫等),构建了最全面的circRNA表达图谱。
比较特殊的就是构建CeRNA network
这个步骤本质上也是数据库查询,而且已经被广泛咀嚼过的TCGA数据库挖掘套路,这里就不展开讨论了。
cirRNA背景知识
资料来源于 Arraystar中国区唯一代理服务商 – 康成生物的介绍
环状RNA(circRNA)是一类由特殊剪接机制形成的、具有闭合环状结构、大量存在于真核转录组中的非编码RNA;也是目前生命科学和医学领域的研究热点分子。circRNA分子的组织特异性、疾病特异性、时序特异性及高稳定性等特征,使得circRNA作为临床疾病的biomarker具有明显的优势。近来研究显示,环状RNA在不同物种中起到miRNA海绵的作用,称之为竞争性内源RNA(ceRNA),即能竞争性结合miRNA。而与疾病关联性miRNA的相互作用说明环状RNA对疾病的调控起着非常重要的作用。此外,一些内含子类型的环状RNA(ciRNA)会促进宿主基因的转录。
实际上,你把上面的circRNA换成其它类型的RNA,就是一样的Arraystar开发世界首款商业化circRNA芯片(2014)
1. 第一款用于circRNA检测的商业化芯片
针对circRNA设计,所有circRNA来源于该领域的标志性研究文献,所有circRNA都经过了严谨的实验验证。
2. 剪接位点特异性的探针
能准确、可靠地检测circRNA,即便在相应线型RNA存在的情况下也能特异性检测circRNA。
3. 详细的circRNA注释
所有circRNA均经过生物信息学分析,预测其结合的microRNAs,并在实验报告中明确这些注释信息(如下图所示)。根据这些信息,客户可以很方便的从miRNA海绵的角度研究circRNA的作用机制和生物功能。
4. 性能保证
灵敏度高,重复性佳,线性检测范围达5个数量级。并设计有各类严谨的质控探针,确保芯片实验质量。 - Human 6×7K 5396 Human circular RNAs
- Mouse 6×7K 1797 Mouse circular RNAs
升级circRNA芯片
Arraystar公司在全球首款circRNA芯片基础上迅即升级版本为V2.0。其circRNA来源融合了环状RNA研究的最新顶尖文献,所有cicrRNA都经过了严谨的实验验证,以便于对不同生理及病理条件下的circRNA进行系统的研究。同时我们对所有差异表达的circRNA用高匹配值的miRNA靶标位点进行了标注,这将有利于对circRNA作为天然miRNA海绵功能的进行研究。
Arraystar公司circRNA芯片产品列表
| 服务 | 芯片 | 规格 | 描述 |
| :—————————————————-: | :—————————————-: | :—-: | :————————————: |
| Human circular RNA Microarray Service | Human circular RNA Array V2.0 | 8x15K | 13,617 human circular RNAs |
| Mouse circular RNA Microarray Service | Mouse circular RNA Array V2.0 | 8×15K | 14,236 Mouse circular RNAs |
| Rat circular RNA Microarray Service | Rat circular RNA Array V2.0 | 8×15K | 14,145 Rat circular RNAs |