我前面写过 单基因GSEA分析策略(数据分析免费做活动继续) ,然后马上就碰到了一个求助,复现下面的图表!
发表在Cancer Management and Research的简单数据挖掘杂志:Apolipoprotein C1 (APOC1) promotes tumor progression via MAPK signaling pathways in colorectal cancer,仔细下载文献学习。
数据下载
关于TCGA数据下载,我挑选了部分,写了6个数据下载系列教程:
- TCGA的28篇教程- 使用R语言的cgdsr包获取TCGA数据(cBioPortal)
- TCGA的28篇教程- 使用R语言的RTCGA包获取TCGA数据 (离线打包版本)
- TCGA的28篇教程-使用R语言的RTCGAToolbox包获取TCGA数据(FireBrowse portal)
- TCGA的28篇教程- 批量下载TCGA所有数据 ( UCSC的 XENA)
- TCGA的28篇教程-数据下载就到此为止吧
- TCGA的28篇教程-整理GDC下载的xml格式的临床资料
但是,建议你选择UCSC的xena数据库下载方式。

首先看表达差异
(A) APOC1 was highly expressed in CRC (n=380) samples compared to adjacent normal (n=50) samples based on The Cancer Genome Atlas (TCGA) database (unpaired t-test, P=0.012). (B) APOC1 was highly expressed in colorectal cancer samples compared to the adjacent normal samples of a matched paired group (n=25) based on The Cancer Genome Atlas (TCGA) database (paired t-test, P=0.002).
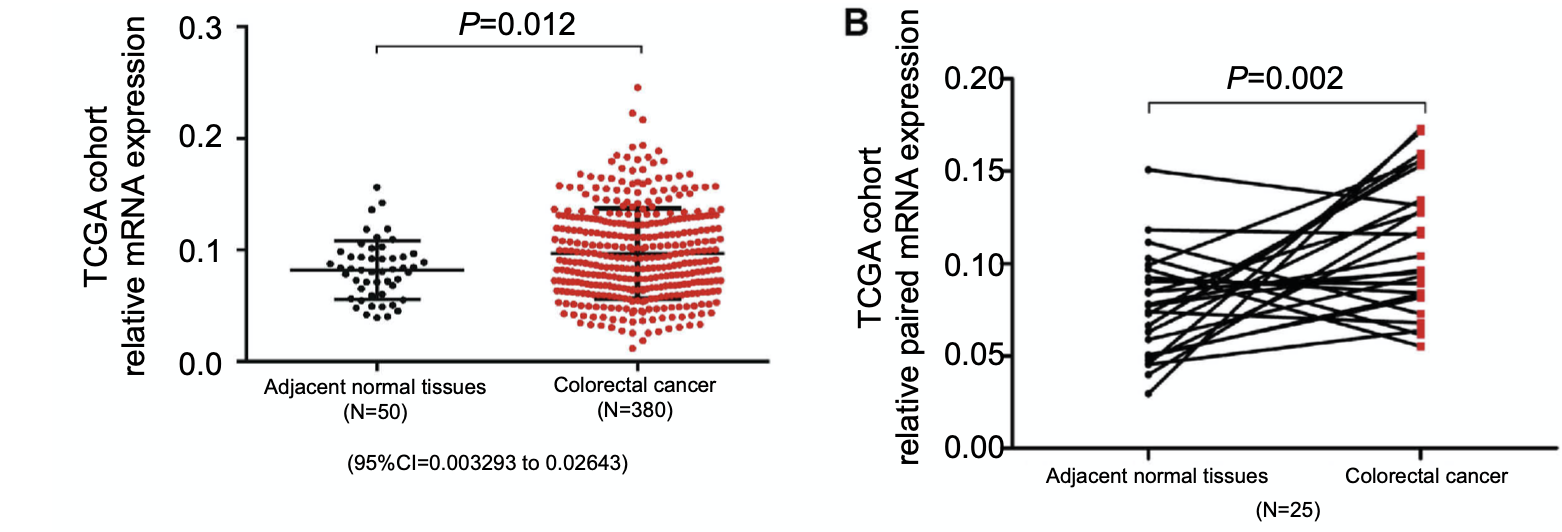
然后看生存效果
我已经在生信技能树多次介绍过生存分析:
- 集思广益-生存分析可以随心所欲根据表达量分组吗
- 生存分析时间点问题
- 寻找生存分析的最佳基因表达分组阈值
- apply家族函数和for循环还是有区别的(批量生存分析出图bug)
- TCGA数据库生存分析的网页工具哪家强
而且使用TCGA数据库来看感兴趣基因的生存情况非常简单,一个网页工具即可,都无需R语言了
(F) and (G) Kaplan– Meier survival analysis according to APOC1 expression in 140 patients with CRC. The overall survival (OS) and disease-free survival (DFS) for patients with high versus low APOC1 expression. The difference is statistically significant based on the log-rank test (both P<0.001).
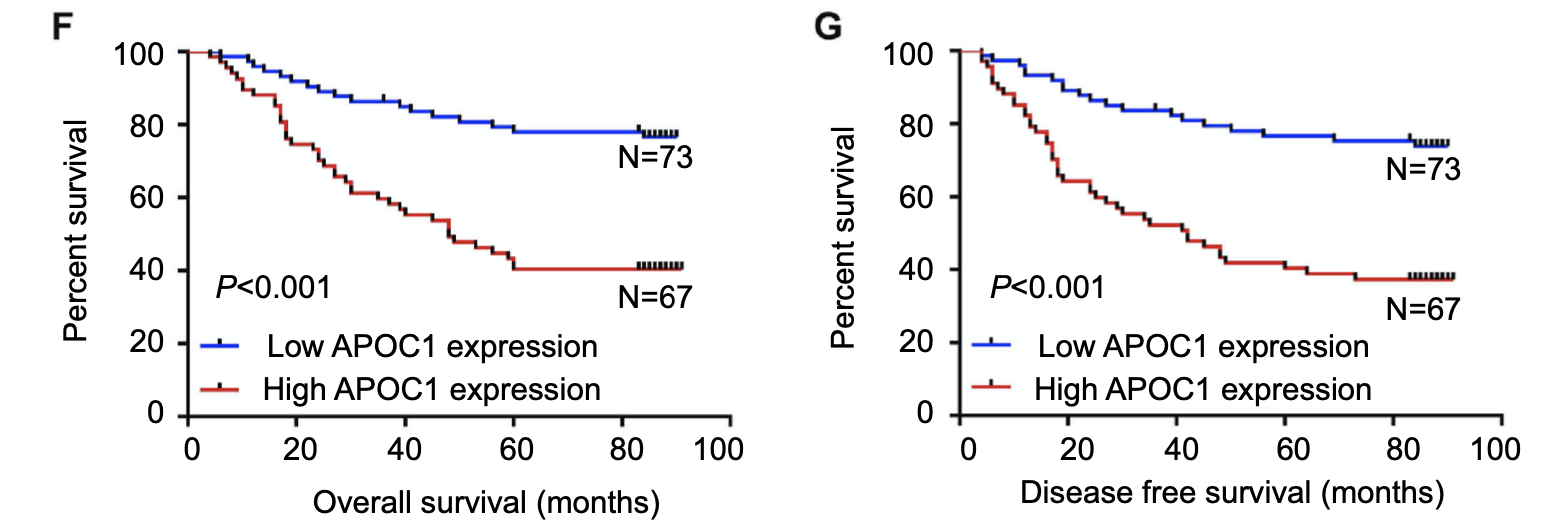
单基因的GSEA
首先需要根据感兴趣的基因表达量高低,对病人进行分组。
(A) GSEA-generated heatmap for highly enriched genes in the MAPK signaling pathway in the APOC1-higher expression group compared to the APOC1-lower expression group from the TCGA COAD-READ dataset.

运行GSEA,需要指定感兴趣的通路进行可视化
(B) GSEA on the TCGA COAD-READ dataset identified MAPK signaling pathways as a regulatory target of APOC1. The GSEA enrichment plot shows values for normalized enrichment score (NES) =1.87 and nominal P-value =0.004.
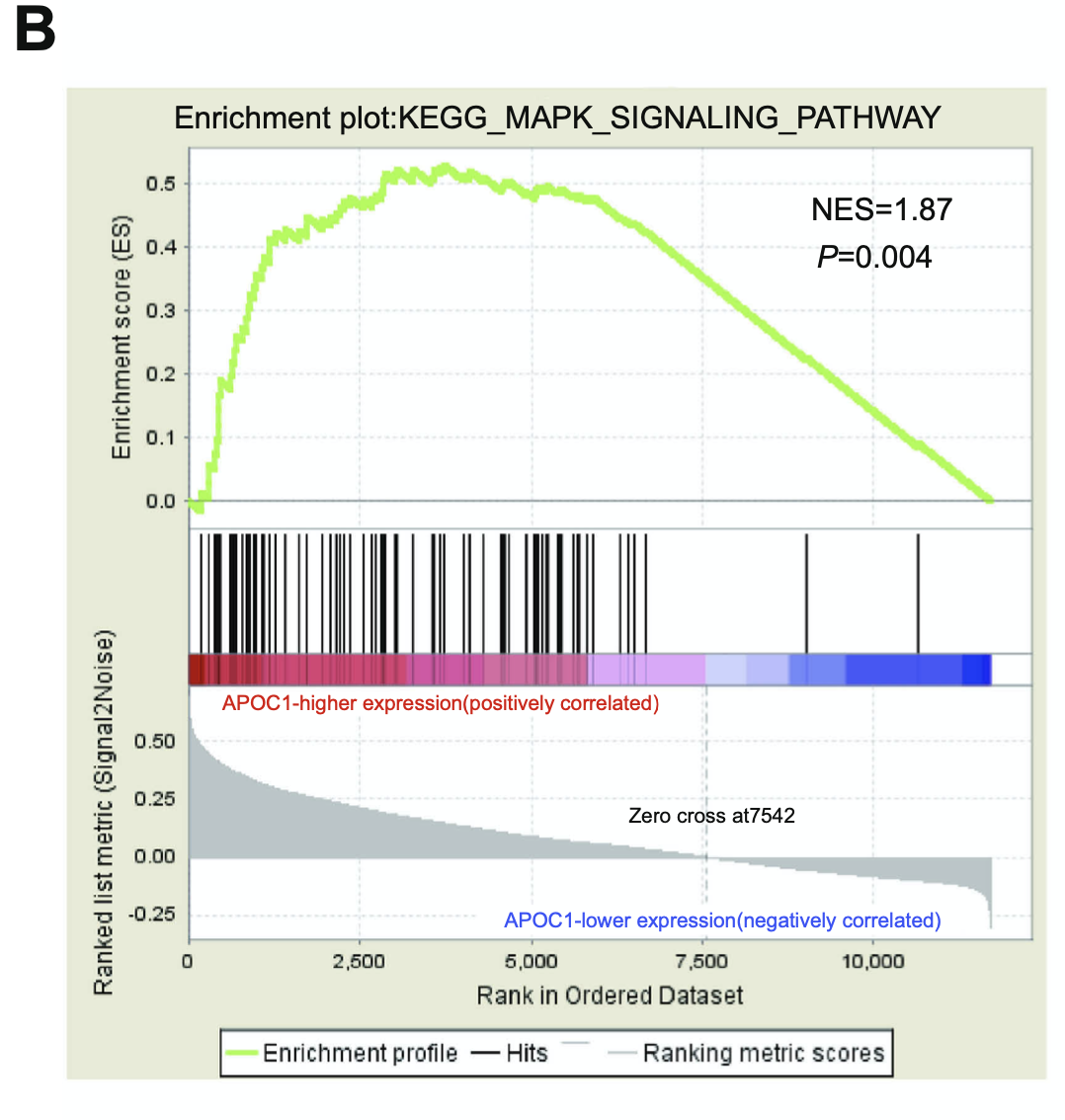
如果大家感兴趣GSEA分析原理和用法,看合辑 - GSEA的统计学原理试讲
- 用GSEA来做基因集富集分析
- 批量运行GSEA,命令行版本
- 费九牛二虎之力也无法重现的GSEA图
- GSEA分析合理性讨论
- 做GSEA分析你的基因到底该如何排序
- 重复不出来我费九牛二虎之力重复不出来的GSEA文章?
- 数据挖掘任务-根据前面教程复现ssGSEA热图