你只需要去查询gencode数据库,就可以看到LINC01666的的坐标是已知的, ENSG00000279579 chr21 8759077 8761335,长度也就是2kb多一点而已,所以在上面设计探针的话应该是不会太多的。请大家记住这个坐标:chr21:8759077-8761335
最近有粉丝反映在挖掘一个公共数据的时候,发现这个芯片平台的LINC01666的探针注释可能是错的,平台如下所示:
- Agilent-028004 SurePrint G3 Human GE 8x60K Microarray (Feature Number version)
- https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GPL13607
使用annoprobe包拿到该平台的注释信息
为了方便大家对表达芯片的探针ID进行注释,我写一个精简包。也在:芯片探针ID的基因注释以前很麻烦 和 :芯片探针序列的基因注释已经无需你自己亲自做了, 里面详细介绍了。如果你想使用的话,最好是先看看哈!
其实最重要的是idmap函数,安装方法说到过:芯片探针序列的基因注释已经无需你自己亲自做了, 使用起来也非常简单:
library(AnnoProbe)
ids=idmap('GPL570',type = 'soft')
head(ids)
仅仅是一句话,就拿到了这个平台的探针的注释信息。需要注意的是,这个函数的type参数,其实是有3个选择,这里我演示的是选择soft这个来源的基因注释信息。
并不是所有的平台都是有soft注释,也不是所有的平台都被我的这个工具囊括哦。
# Agilent-028004 SurePrint G3 Human GE 8x60K Microarray (Feature Number version)
# https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GPL13607
rm(list = ls())
library(AnnoProbe)
# 如果GPL平台提供注释,就使用这个方法
library(GEOquery)
i='GPL13607'
gpl <- getGEO( i , destdir=".")
save(gpl,file = 'GPL13607.Rdata' )
load('GPL13607.Rdata')
library(GEOquery)
colnames(Table(gpl))
head(Table(gpl))
tmp=Table(gpl)
write.table(tmp,file = 'GPL13607.txt',sep = '\t',quote=F,row.names = F)
ids=Table(gpl)[,c(1,6)]
ids2=ids
head(ids2)
## 如果GPL平台没有提供,就使用我的注释
ids3=idmap(gpl='GPL13607',type = 'pipe')
head(ids3)
length(unique(ids2[,1]))
length(unique(ids3[,1]))
检查发现LINC01666的探针的确是冲突的
比如我注释A_19_P00803024代表的序列 AGTTCTTCCCTGGAAACTAGGTCTCGTATAAACTTCTGTAAATGCGACCCAGGAGGACTA 按照道理应该是可以注释到LINC01666这个基因的。但是GPL里面的信息如下:
7121 A_19_P00803024 0
lincRNA:chr1:142789202-142921002_F
lincRNA:chr1:142789202-142921002 forward strand
hs|chr1:142890398-142890457
AGTTCTTCCCTGGAAACTAGGTCTCGTATAAACTTCTGTAAATGCGACCCAGGAGGACTA
lincRNA
这个就是很诡异了,GPL里面显示的是 lincRNA:chr1:142789202-142921002 ,那么这个探针序列,应该是可以比对到这个坐标吧,可是我blat了一下,分析:
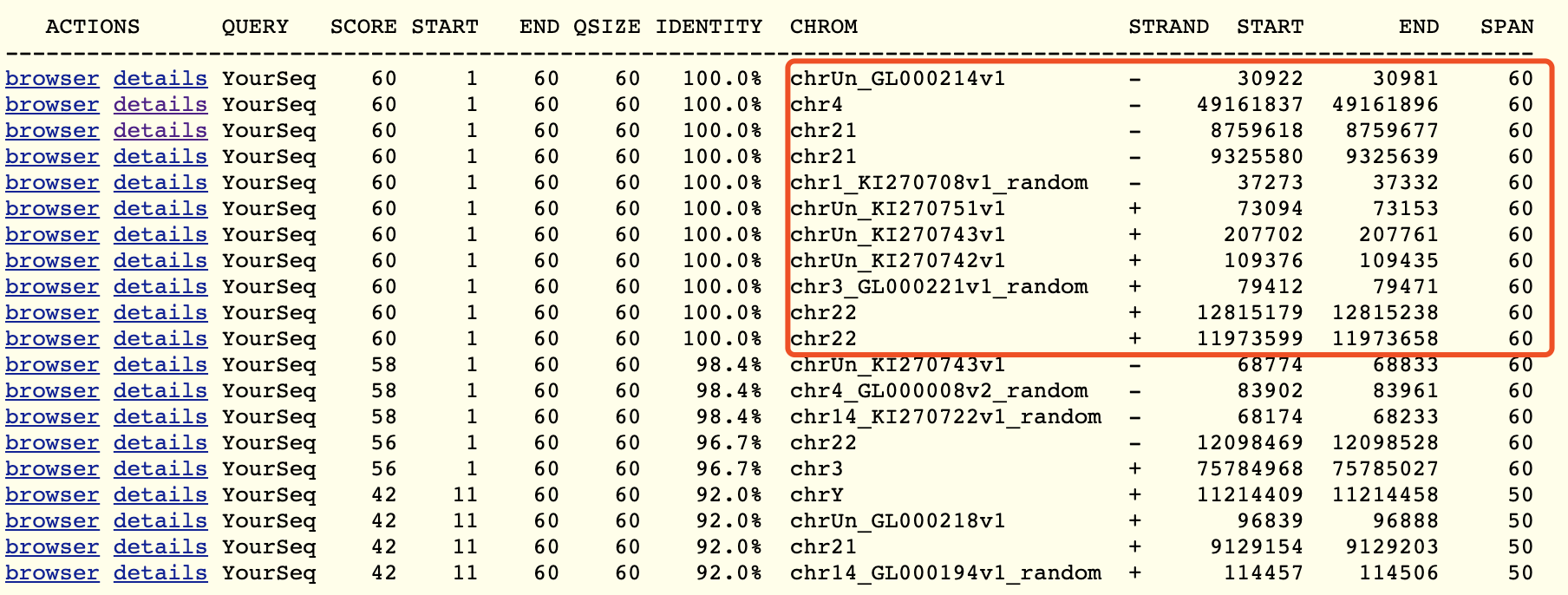
目瞪狗呆!一个探针序列,就60个碱基,比对到了那么多位置,而且都是100%的比对情况,哔了狗了!可以看到gencode数据库的LINC01666的基因坐标 chr21:8759077-8761335 是包含了上面的第3个比对情况的,所以我们把这个探针注释到LINC01666基因是没有错误的!
但是,为什么gpl里面显示它这个探针是 lincRNA:chr1:142789202-142921002 ,几乎可以肯定gpl里面的信息是错的!
探针A_19_P00811275
再看另外一个探针 A_19_P00811275 , 我的注释结果是:AC138776.1 transcribed_unprocessed_pseudogene ENSG00000286406 chr22 11827523 11910358 ,但是GPL里面的信息是:
29278 A_19_P00811275 0
lincRNA:chr1:142789202-142921002_R
lincRNA:chr1:142789202-142921002 reverse strand
hs|chr1:142790275-142790216
TAGAATTCGCCCTACTACTACCCTTACTGTGAGCCCTTCAAACAATCTGATACTAATAAT
lincRNA
同样的道理,我们看看blat比对结果:
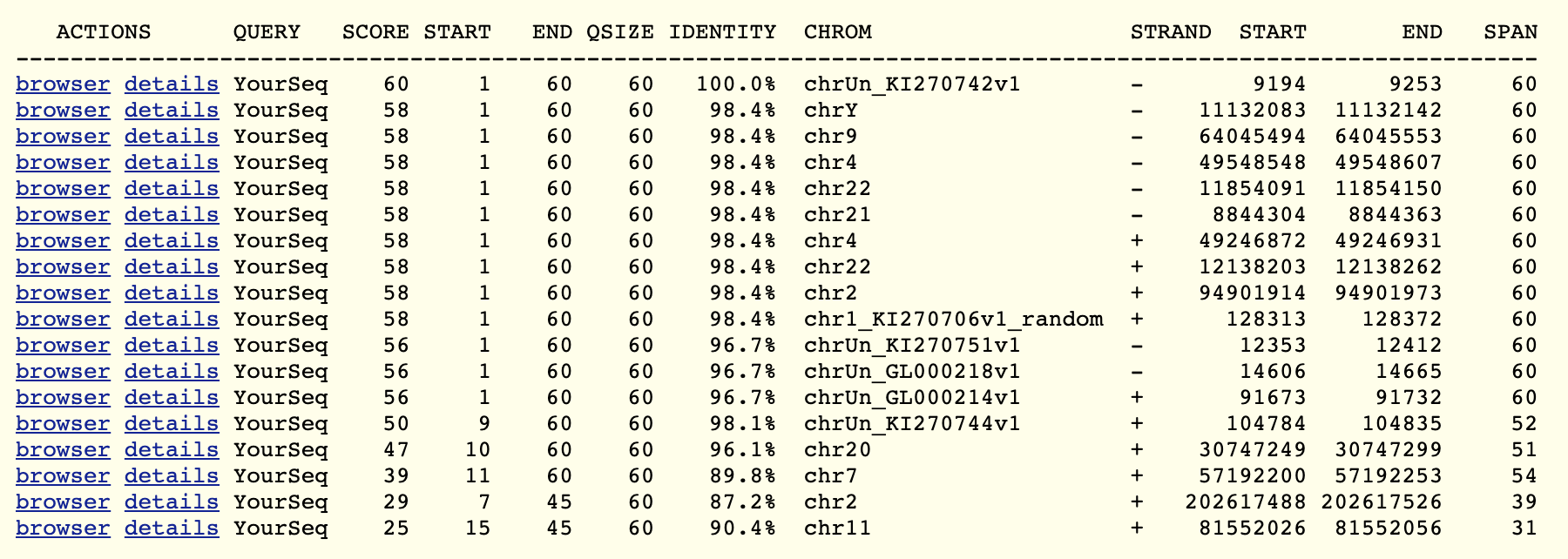
没啥好说的,我仍然是对的!
探针A_33_P3416922
最后看看探针A_33_P3416922,在GPL注释好奇怪,是chrUn开头的,通常这样的话,这个探针就可以剔除了,因为都不在常规染色体上面,到时候即使分析出来,这个探针很重要,后续实验验证,或者生物学解释都是苍白无力的!
55797 A_33_P3416922 0
A_33_P3416922 Unknown
hs|chrUn_gl000240:000037946-000038005 ACACCTTTCCTTCTATCCAAGCCCTCATATTATGCTCTGACAATAAATTGGGCTTTTCCA
A_33_P3416922
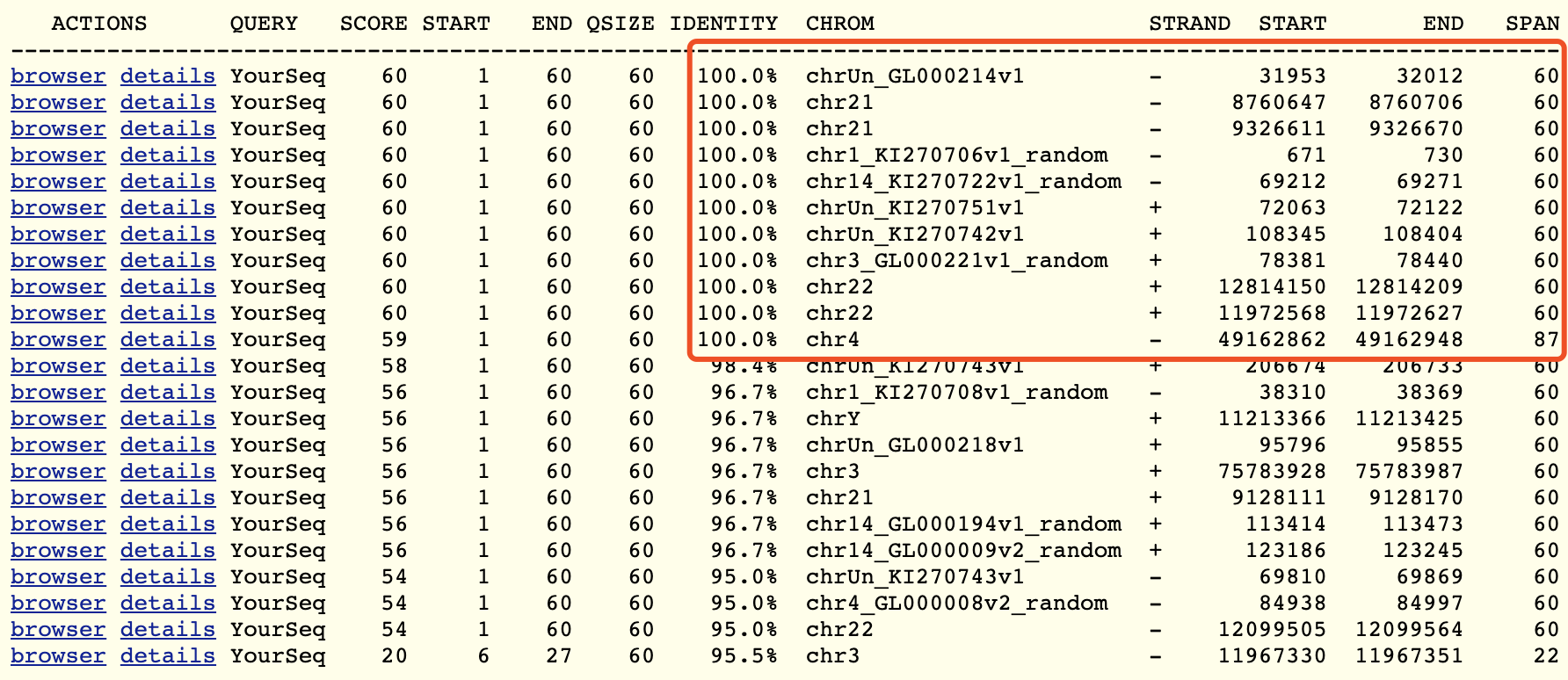
那么,同样的,我给它的注释是LINC01666的基因坐标 chr21:8759077-8761335 ,blat可以看到,也是合理的!
总结一下
从一个lncRNA的角度,人工审核了3个探针,发现GPL注释是有问题的,相比起来,我的R包注释要准确可靠的多!
欢迎大家继续试用体验哈。
为了方便大家对表达芯片的探针ID进行注释,我写一个精简包。也在:芯片探针ID的基因注释以前很麻烦 和 :芯片探针序列的基因注释已经无需你自己亲自做了, 里面详细介绍了。如果你想使用的话,最好是先看看哈!
其实最重要的是idmap函数,安装方法说到过:芯片探针序列的基因注释已经无需你自己亲自做了, 使用起来也非常简单。
芯片探针ID注释免费做
GEO数据库挖掘,我写了大量教程,其实卡脖子的就两个地方,首先是样本进行合理的分组,需要生物学背景和编程技能。另外一个就是探针序列的基因注释啦,这个我可以帮助大家。本次活动我可以帮你免费做一次 表达芯片的探针ID注释
我们推文里面提到的各种各样的数据分析环节都是我非常有经验的,比如我在lncRNA的一些基础知识 ,和lncRNA芯片的一般分析流程 介绍过的那些图表,以及下面的目录的分析内容 对我来说是举手之劳,希望可以帮助到你!
- 转录组数据分析的4个维度认识(数据分析继续免费哦) RNA-seq数据的2个分组差异分析,热图,PCA图,火山图等等
- 根据感兴趣基因看肝癌免疫微环境的T细胞亚群差异 条形图或者箱线图
- 查看感兴趣基因的甲基化水平和RNA表达水平(数据分析免费做)相关性 散点图或者箱线图
- 我不相信kmplot这个网页工具的结果(生存分析免费做)
- 单基因GSEA分析策略(数据分析免费做活动继续)
- 干扰一个基因然后分析全局基因表达其实是无法定位该基因完整功能(春节免费数据分析活动继续)
- log与否会改变rpkm形式表达矩阵top的mad基因列表 WGCNA分析免费做
- 甲基化信号值的差异分析也许不应该是看logFC 甲基化信号矩阵差异分析免费做
- WGCNA得到模块之后如何筛选模块里面的hub基因 WGCNA分析免费做
- 既然可以看感兴趣基因的生存情况,当然就可以批量做完全部基因的生存分析
还是老规矩,发送数据分析要求,以及简短的项目描述到我的邮箱 jmzeng1314@163.com
邮件正文最好是加上你是啥时候认识生信技能树的哦,或者其它一些寒暄的话,自我介绍也行。主要是考虑到可能想免费分析数据的朋友很多,所以会根据你的来信,我主观判定一个优先级哦。目前我有20多个愿意长期在我的指导下进行数据探索的学徒,等我的团队扩大到200人,我们应该是可以做到数据分析全部免费,敬请期待哈!