2013年,“单细胞测序”被nature杂志选为“年度技术”,以突出这项技术可以对单个细胞进行DNA和RNA测序,进而让科学家能够推断不同细胞的生物学差异。从此单细胞测序飞速发展,扩大了我们对细胞异质性及其对细胞功能的影响的认识,我们单细胞天地也是这两年集中火力系统性整理了大量单细胞数据分析经验。
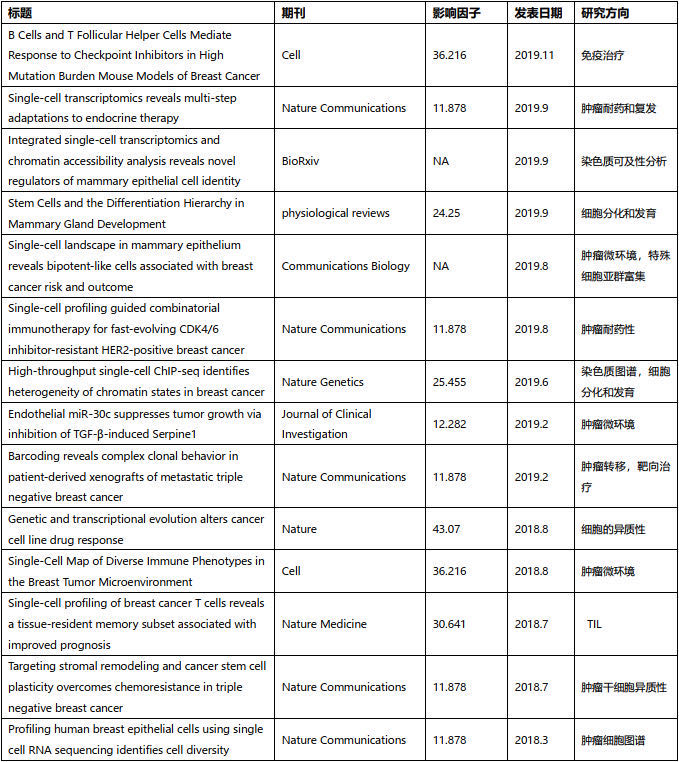
乳腺癌是单细胞领域文章最多的,列表如下:
而“2019年度技术”(Method of the Year 2019)选择了单细胞多组学,可以对单个细胞的基因组、转录组、蛋白质组、代谢组等生物学标志进行多维分析,这个分析难度更高。我们还在研发阶段,不过看到朋友圈有人分享一个单细胞病理水平的研究,发表在 Nature (20 January 2020),文章标题是:The single-cell pathology landscape of breast cancer.
该研究首先利用成像型质谱流式细胞仪同时对35种生物学标志进行量化,从而获得乳腺癌患者肿瘤组织的高维病理学图像,图像识别技术看里面的单细胞各种生物学标志物的测量值。
病人队列及取样
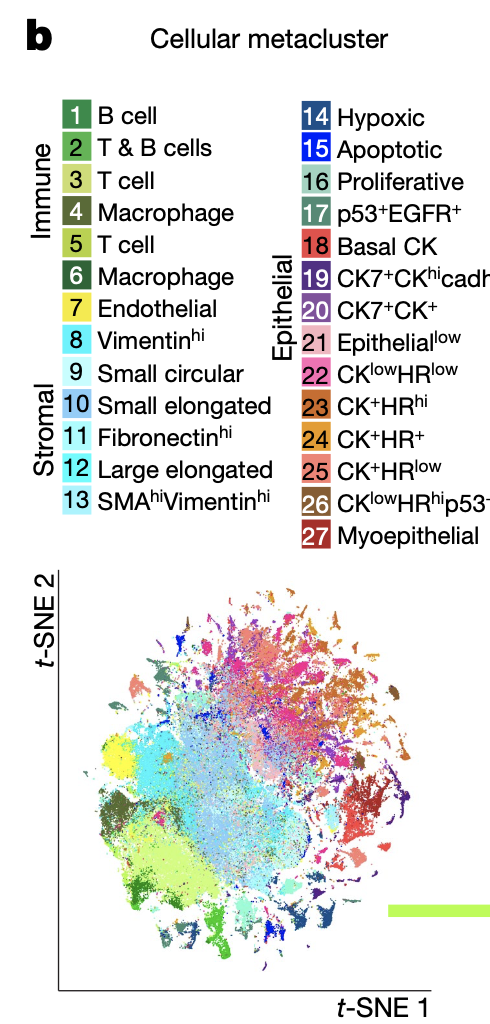
We identified 855,668 cells in 381 images (289 tumour, 87 healthy breast and 5 liver controls), and quantified the expression of both marker genes and the spatial features of each cell.
171,288 subsampled 单细胞tSNE图
拿到矩阵后也是同样的分析,走我们一直讲解的R包及基础流程即可,分别是: scater,monocle,Seurat,scran,M3Drop 需要熟练掌握它们的对象,:一些单细胞转录组R包的对象 流程也大同小异:
- step1: 创建对象
- step2: 质量控制
- step3: 表达量的标准化和归一化
- step4: 去除干扰因素(多个样本整合)
- step5: 判断重要的基因
- step6: 多种降维算法
- step7: 可视化降维结果
- step8: 多种聚类算法
- step9: 聚类后找每个细胞亚群的标志基因
- step10: 继续分类
乳腺癌恶性的上皮细胞和肿瘤微环境分开分析
首先需要有生物学背景,知道细胞可以分成:
- (1) tumour epithelium;
- (2) endothelium;
- (3) immune cells;
- (4) surrounding stroma;
- (5) endothelium and T cells;
- (6) proliferating epithelium surrounding endothelial cells.
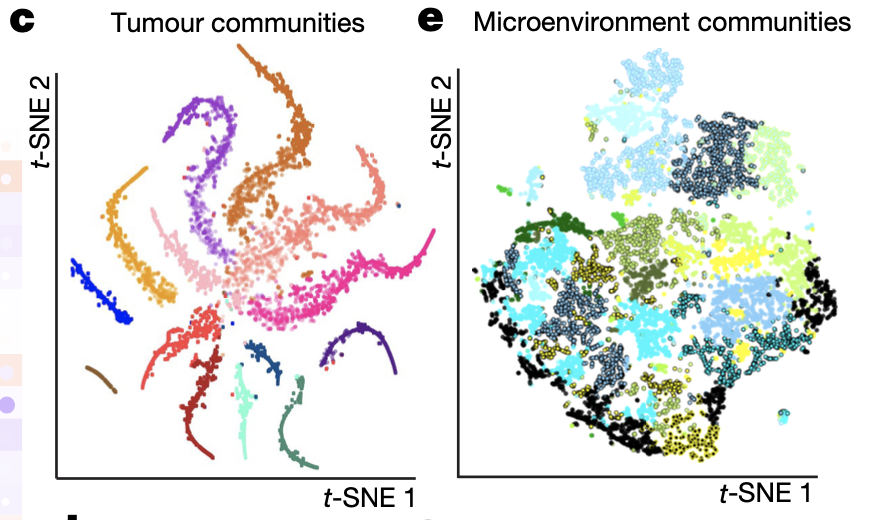
- 图C是 Individually coloured epithelial communities (n = 8,495)
- 图E是 microenvironment communities (n = 12,854)
而且得到了规律之后,又纳入72个病人,采样344 additional images containing a total of 411,410 cells 验证。