我们推文里面提到的各种各样的数据分析环节都是我非常有经验的,比如我在lncRNA的一些基础知识 ,和lncRNA芯片的一般分析流程 介绍过的那些图表,以及下面的目录的分析内容 对我来说是举手之劳,希望可以帮助到你!
- 转录组数据分析的4个维度认识(数据分析继续免费哦) RNA-seq数据的2个分组差异分析,热图,PCA图,火山图等等
- 根据感兴趣基因看肝癌免疫微环境的T细胞亚群差异 条形图或者箱线图
- 查看感兴趣基因的甲基化水平和RNA表达水平(数据分析免费做)相关性 散点图或者箱线图
- 我不相信kmplot这个网页工具的结果(生存分析免费做)
- 单基因GSEA分析策略(数据分析免费做活动继续)
你只需要发送数据分析要求,以及简短的项目描述到我的邮箱 jmzeng1314@163.com (活动时间仅限于春节前后一个月哈)
邮件正文最好是加上你是啥时候认识生信技能树的哦,或者其它一些寒暄的话,自我介绍也行。主要是考虑到可能想免费分析数据的朋友很多,所以会根据你的来信,我主观判定一个优先级哦。目前我有20多个愿意长期在我的指导下进行数据探索的学徒,等我的团队扩大到200人,我们应该是可以做到数据分析全部免费,敬请期待哈!
为什么分析是免费的呢?
因为你给我的需求,只要是常规的,对我来说,跑一个代码真的是举手之劳,会自动化输出一大堆的图表,我全部打包给你即可。但是因为是免费的,我不会给你如何解释,只会告诉你R包版本,数据库版本,软件阈值参数,如果你使用这些图表发表的话,就可以用上了。
但是代码并没有给你,因为如果你找我帮忙分析数据,大概率上是因为你看不懂代码,给你也是浪费。有趣的是很多朋友留言说需要我的代码去学习一下,想知道如何批量做全部的分析,自动化输出一堆图表,如下:
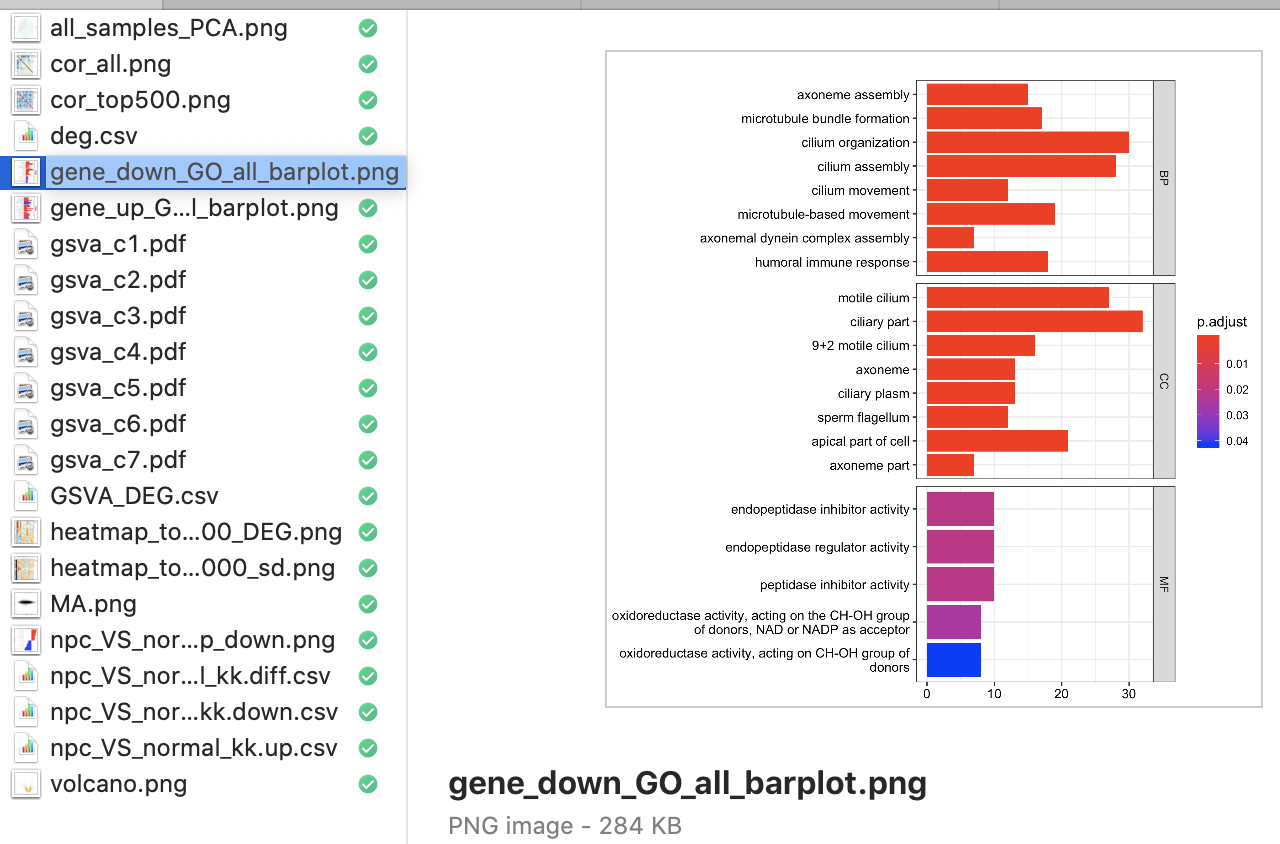
就是走标准分析流程,火山图,热图,GO/KEGG数据库注释等等。这些流程的视频教程都在B站和GitHub了,目录如下:
- 第一讲:GEO,表达芯片与R
- 第二讲:从GEO下载数据得到表达量矩阵
- 第三讲:对表达量矩阵用GSEA软件做分析
- 第四讲:根据分组信息做差异分析
- 第五讲:对差异基因结果做GO/KEGG超几何分布检验富集分析
- 第六讲:指定基因分组boxplot指定基因list画热图
感兴趣可以细读表达芯片的公共数据库挖掘系列推文 ;
- 解读GEO数据存放规律及下载,一文就够
- 解读SRA数据库规律一文就够
- 从GEO数据库下载得到表达矩阵 一文就够
- GSEA分析一文就够(单机版+R语言版)
- 根据分组信息做差异分析- 这个一文不够的
- 差异分析得到的结果注释一文就够
代码我们以一个新的方式给到大家
因为我们生信技能树公众号表现良好,可以开通付费阅读功能,正好体验一下!应该是花3元就可以打开我们隐藏好的代码,详见:https://mp.weixin.qq.com/s/_iOit9MtjE3dNSWDuj1W3A
代码文件左边是行数:
149 kegg_and_go_up_and_down.R
57 step0-install.R
87 step1-download.R
77 step2-check.R
141 step3-DEG.R
62 step4-anno-go-kegg.R
51 step5-anno-GSEA.R
118 step6-anno-GSVA.R
59 step7-visualization.R
谢谢大家参与哦,付费人数足够,我们才可以正式加入腾讯优秀自媒体阵营,谢谢你的参与和支持!
温馨提示
里面的代码,用到了一个数据库文件,在我的电脑里面是:~/biosoft/MSigDB/symbols/ 当然,你可以去官网下载最新的哦。
文件如下:
238K Jul 25 2018 c1.all.v6.2.symbols.gmt
3.2M Jul 25 2018 c2.all.v6.2.symbols.gmt
1.2M Jul 25 2018 c3.all.v6.2.symbols.gmt
601K Jul 25 2018 c4.all.v6.2.symbols.gmt
4.7M Jul 25 2018 c5.all.v6.2.symbols.gmt
207K Jul 25 2018 c6.all.v6.2.symbols.gmt
6.4M Jul 25 2018 c7.all.v6.2.symbols.gmt
47K Jul 25 2018 h.all.v6.2.symbols.gmt
然后你可能会遇到GEO数据库的GSE数据集下载困难这个门槛,所以呢,我有一个GEO数据库中国区镜像横空出世 ,理论上是能够帮助你的。
最后,如果是你自己的数据集,有可能是芯片比较偏僻。我也有一个暂时托管在GitHub平台的包可以帮助你, 在:芯片探针ID的基因注释以前很麻烦 和 :芯片探针序列的基因注释已经无需你自己亲自做了, 里面详细介绍了。最重要的是idmap函数,安装方法说到过:芯片探针序列的基因注释已经无需你自己亲自做了, 使用起来也非常简单:
library(AnnoProbe)
ids=idmap('GPL570',type = 'soft')
head(ids)
并不是所有的平台都是有soft注释,也不是所有的平台都被我的这个工具囊括哦。
仅仅是一句话,就拿到了这个平台的探针的注释信息。需要注意的是,这个函数的type参数,其实是有3个选择,这里我演示的是选择soft这个来源的基因注释信息。