都介绍到单细胞转录组数据处理之细胞亚群比例比较部分了,10讲就告一段落了,大家可以回看仔细品读。后面的分析其实都是个性化的了,取决于课题设计,假说,生物学背景知识,而且需要学习大量的R包。
既然是个性化分析,理论上就是无穷无尽的,而且我在 有一种生意双方都觉得亏 提到过,专业的工程师觉得为客户学习一个R包收费2000合情合理,但是委托者觉得一个项目全套分析收2000才合理。也就是说大部分情况下,大家自己的课题的个性化需求,是很难找到合适的合作者来代替你完成的,不得不学习多种多样的R包算法,而且通常是找不到交流者。
我们就以中山大学的最新研究《一个人的15个器官单细胞测序数据 》,链接是:https://www.biorxiv.org/content/10.1101/2020.03.18.996975v1.full.pdf 为例子,看看多少种个性化分析吧。
拟时序分析
通常是monocle流程,也可以是diffusion map等等,我们以前其实分享过很多教程了。

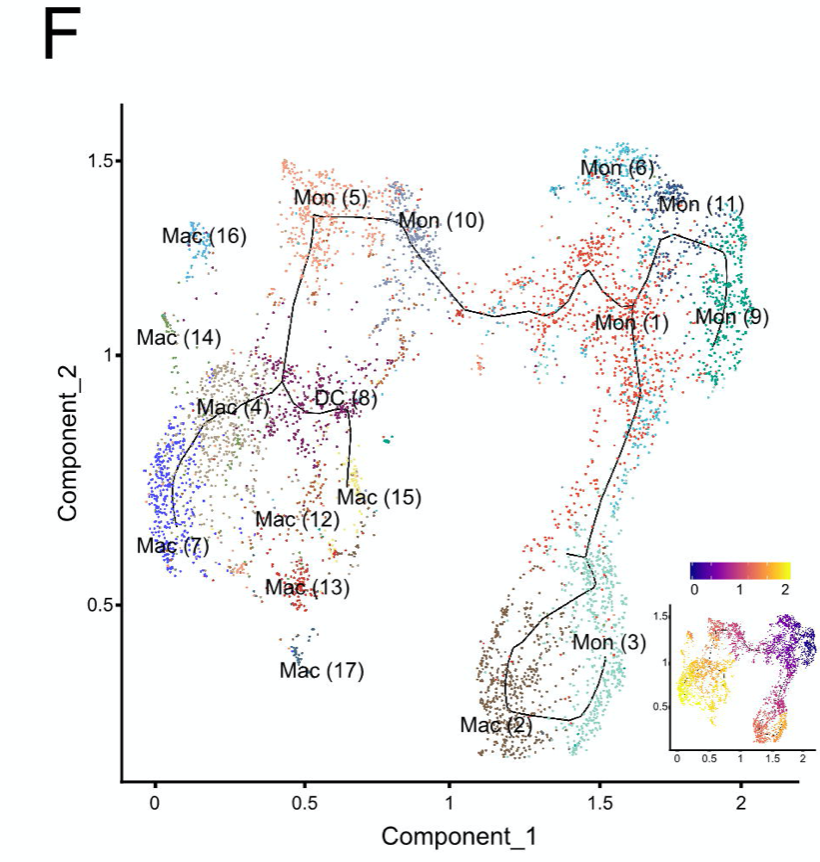
在文章里面主要是figures4F,图例是:
F. Pseudo-time trajectory analysis of all myeloid cells with high variable genes.
- Each dot represents a single cell and is colored according to their clustering in A.
- The inlet t-SNE plot at each plot shows each cell with a pseudo-time score from dark blue to yellow, indicating early and terminal states, respectively.
如下:
GO和KEGG等生物学数据库注释
主要是在figures5D-E,图例是:D, E. Gene Ontology enrichment analysis results of each epithelial cell cluster in digestive organs (D) and non-digestive organs (E). Cell clustered as numbered below were colored according to their -log10P values. Only the top 20 significant terms (p-value < 0.05) were shown.
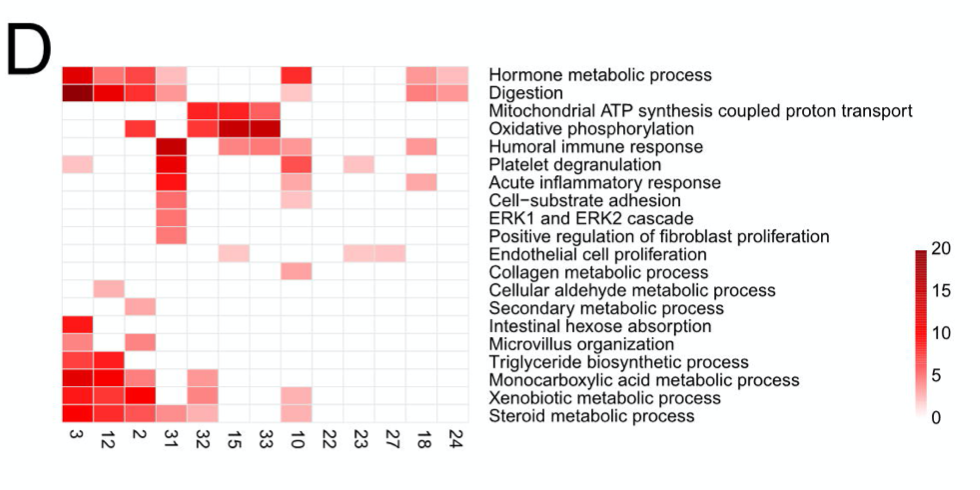
这个虽然是个性化分析,但是在常规转录组里面已经烂大街了,需要注意的是GO和KEGG等生物学数据库条目非常多,如果生物学背景不够,大部分情况下是出图后就无动于衷。
比如这篇文章就是有偏向的关注 GO Biological Processes ,而不是CC或者MF,甚至也不是KEGG数据库。

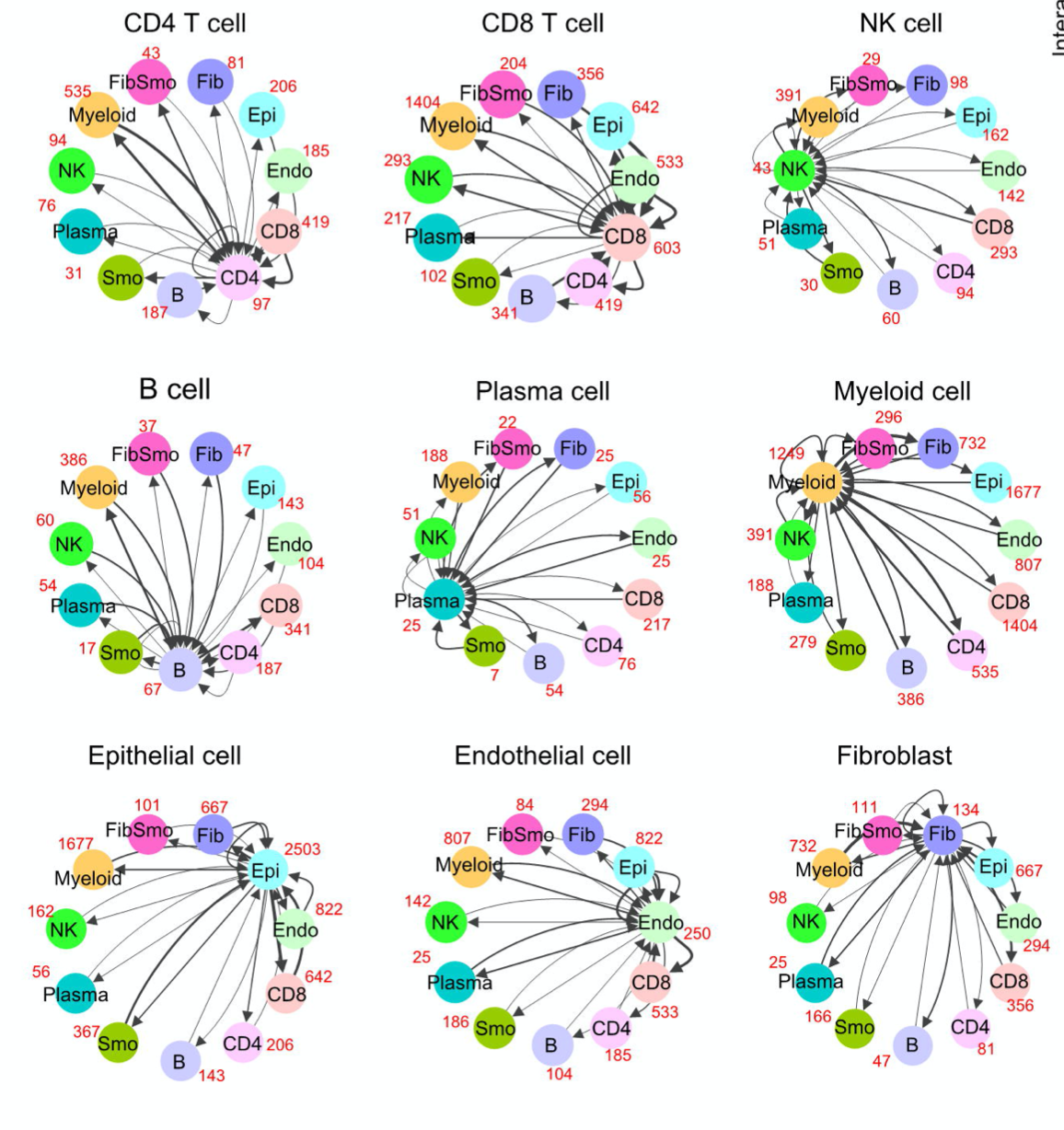
细胞通讯CellphoneDB
该工具算法正式发表于26 February 2020 ,链接是:https://www.nature.com/articles/s41596-020-0292-x ,代码在 https://github.com/Teichlab/cellphonedb ,流行程度尚可,所以写关于它教程的很多。

重要就是给11种主要的细胞亚群,进行关系配对的计算。 - CD4, CD4+ T cells;
- CD8, CD8+ T cells
- B, B cells
- Plasma, plasma cells
- Myeloid, myeloid cells
- NK, NK cells
- Epi, epithelial cells
- Fib, fibroblasts
- Smo, smooth muscle cells
- FibSmo, FibSmo cells
- Endo, endothelial cells)
Numbers in red indicate the counts of ligand-receptor pairs for each intercellular link.
使用SCENIC进行转录因子调控分析
SCENIC发表要早于前面的CellphoneDB,是2017年的Nature methods文章,链接: https://www.nature.com/articles/nmeth.4463 主要是根据表达矩阵来计算每个细胞可能的调控基因。

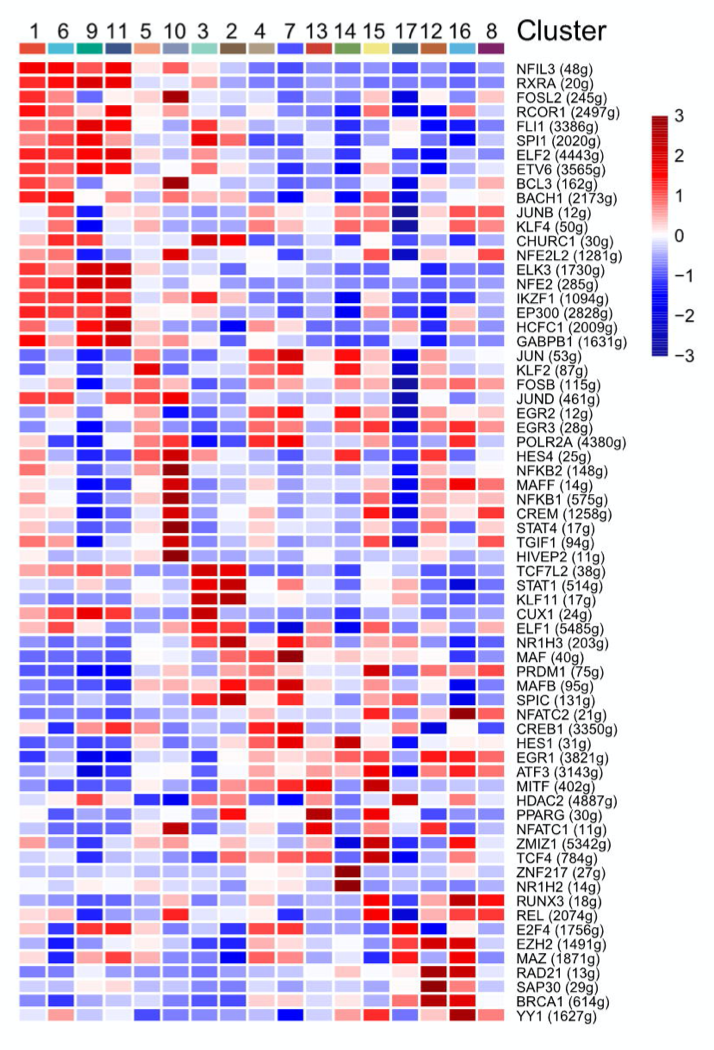
图例是:F. Heatmap of the active scores of epithelial cell subtypes as numbered on top, of which expression was regulated by transcription factors (TFs), as estimated using SCENIC analysis.
Shown are the top 10 TFs having the highest difference in expression regulation estimates between each cluster and all other cells, tested with a Wilcoxon rank-sum test.
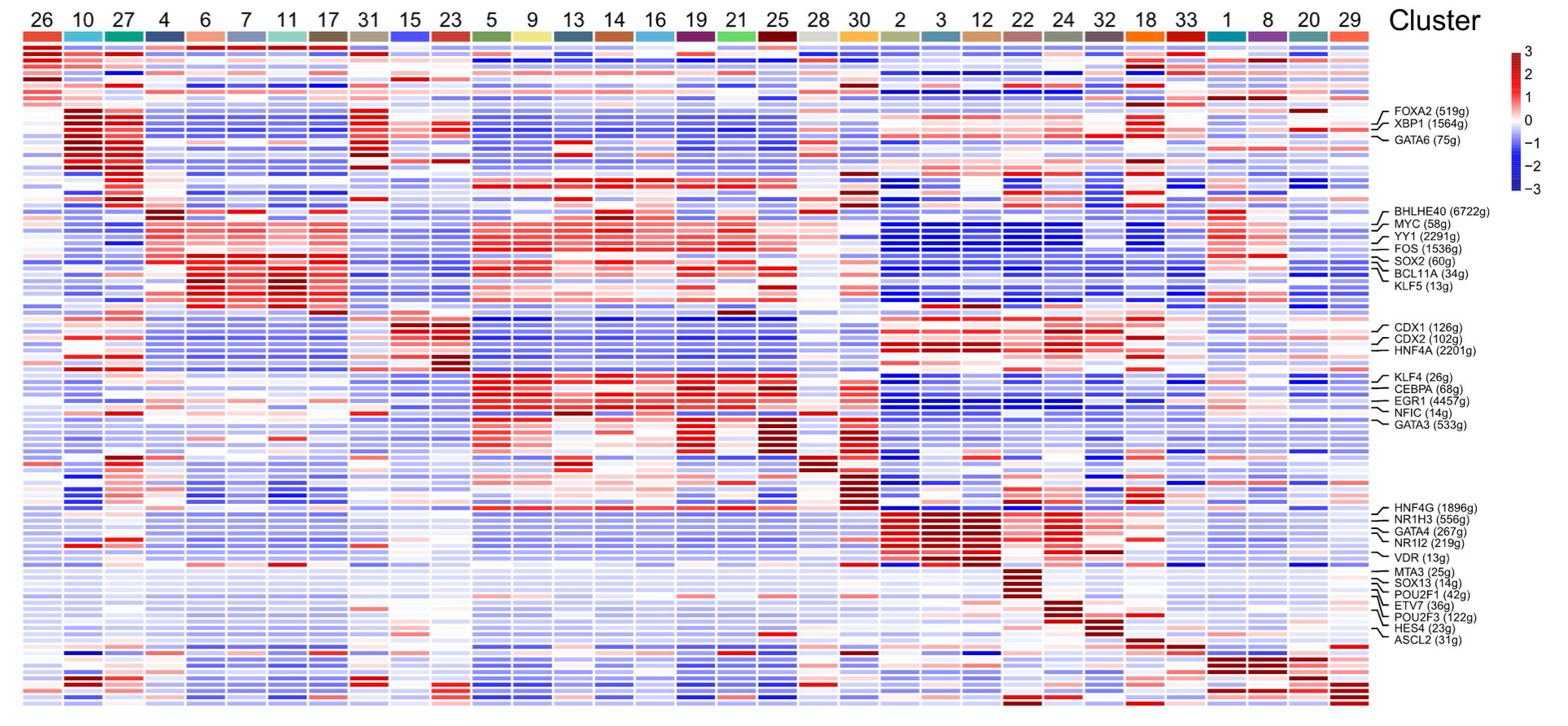
另外一个关于 myeloid cell subtype 的SCENIC分析结果如下:
解锁更多分析
如果你看的单细胞转录组数据分析文章足够多, 就可以把分析点收集齐全,通常也只有科研服务公司的单细胞研发团队才会做这样的努力。
比如我们生信菜鸟团分享的:Cell | 单细胞技术揭示人肝细胞图谱 提到的10372个人正常细胞、1052个类器官细胞、1282个肝癌细胞和311个移植后小鼠细胞项目。使用的是RaceID3(FateID包)进行后续分析;并标准化,使用的是destiny包做伪时间分析和自组织映射。
再比如2020年3月25日,郭国骥团队在Nature杂志 上发表文章 Construction of a Human Cell Landscape at Single-cell Level ,对60种人体组织样品和7种细胞培养样品进行了Microwell-seq高通量单细胞测序分析,系统性地绘制了跨越胚胎和成年两个时期,涵盖八大系统的人类细胞图谱。建立了70多万个单细胞的转录组数据库,鉴定了人体100余种细胞大类和800余种细胞亚类。基于该数据库,团队开发了scHCL单细胞比对系统用于人体细胞类型的识别,并搭建了人类细胞蓝图网站http://bis.zju.edu.cn/HCL/(国家基因库镜像https://db.cngb.org/HCL/)。
通常是细胞图谱类文章涉及到的新工具比较多。第二期单细胞视频笔记汇总
免疫治疗
- 10X scRNA免疫治疗学习笔记1-前言
- 10X scRNA免疫治疗学习笔记-2-配置Seurat的R语言环境
- 10X scRNA免疫治疗学习笔记-3-Seurat标准流程
- 10X scRNA免疫治疗学习笔记-4-细胞亚群的生物学命名
- 10X scRNA免疫治疗学习笔记-5-差异分析及可视化
- 10X scRNA免疫治疗学习笔记-6-marker基因的表达量可视化
- 10X scRNA免疫治疗学习笔记-7-条条道路通罗马—单细胞分群分析
发育
- scRNA小鼠发育Smartseq2流程—前言及上游介绍
- 根据表达矩阵进行分群-1
- 根据表达矩阵进行分群-2
- 标记基因可视化
- 差异分析及功能注释(上)
- 差异分析及功能注释(下)
- 发育谱系推断及可视化
- 不同谱系的差异基因分类注释
单细胞基因组
- 单细胞基因组拷贝数变异流程
其它R包及笔记
- M3Drop
- 单细胞的qPCR
- 比较5种scRNA鉴定HVGs方法
- 单细胞分群后继续分亚群的一些例子
文末友情宣传
强烈建议你推荐给身边的博士后以及年轻生物学PI,多一点数据认知,让他们的科研上一个台阶:
- 全国巡讲全球听(买一得五)第3期(4月6日开始) ,你的生物信息学入门课。
- 数据挖掘线上班来袭(两天变三周,实力加量),医学生/医生首选技能提高课。
- 生信技能树的2019年终总结 ,你的生物信息学成长宝藏
- 2020学习主旋律,B站74小时免费教学视频为你领路