前面我们分享了:microRNAs靶基因数据库哪家强,提到了综合了12个网页工具的miRWalk,以及整合了7个工具的miRSystem,但是最后我们仍然是推荐R包multiMiR作为提取miRNA的预测靶基因结果的解决方案,除了因为它综合了14个网页工具,而且还是这个月更新过,还有一个很大的原因是,我喜欢编程,喜欢R语言!
今天我们比较一下另外的一个miRNAtap包,与multiMiR的结果的一致性,以及两个数据库的差异!首先安装miRNAtap包,代码如下:
options(BioC_mirror="https://mirrors.tuna.tsinghua.edu.cn/bioconductor/")
options("repos" = c(CRAN="http://mirrors.cloud.tencent.com/CRAN/"))
options(download.file.method = 'libcurl')
options(url.method='libcurl')
BiocManager::install("miRNAtap",ask = F,update = F)
BiocManager::install("topGO",ask = F,update = F)
BiocManager::install("miRNAtap.db",ask = F,update = F)
在miRNAtap包说明书很清楚的写着,它其实就是综合另外5个流行的数据源,然后总结一下,而且仅仅是支持人和鼠这两个物种哦!

首先看看,对指定的一个miRNA进行靶基因提取:
rm(list = ls())
library(miRNAtap)
library(topGO)
library(org.Hs.eg.db)
mir = 'miR-10b'
predictions = getPredictedTargets(mir, species = 'hsa',
method = 'geom', min_src = 2)
head(predictions)
predictions_min = getPredictedTargets(mir, species = 'hsa',
method = 'min', min_src = 2)
head(predictions_min)
两个总结5个数据库结果的方法差异在于靶基因的排名:
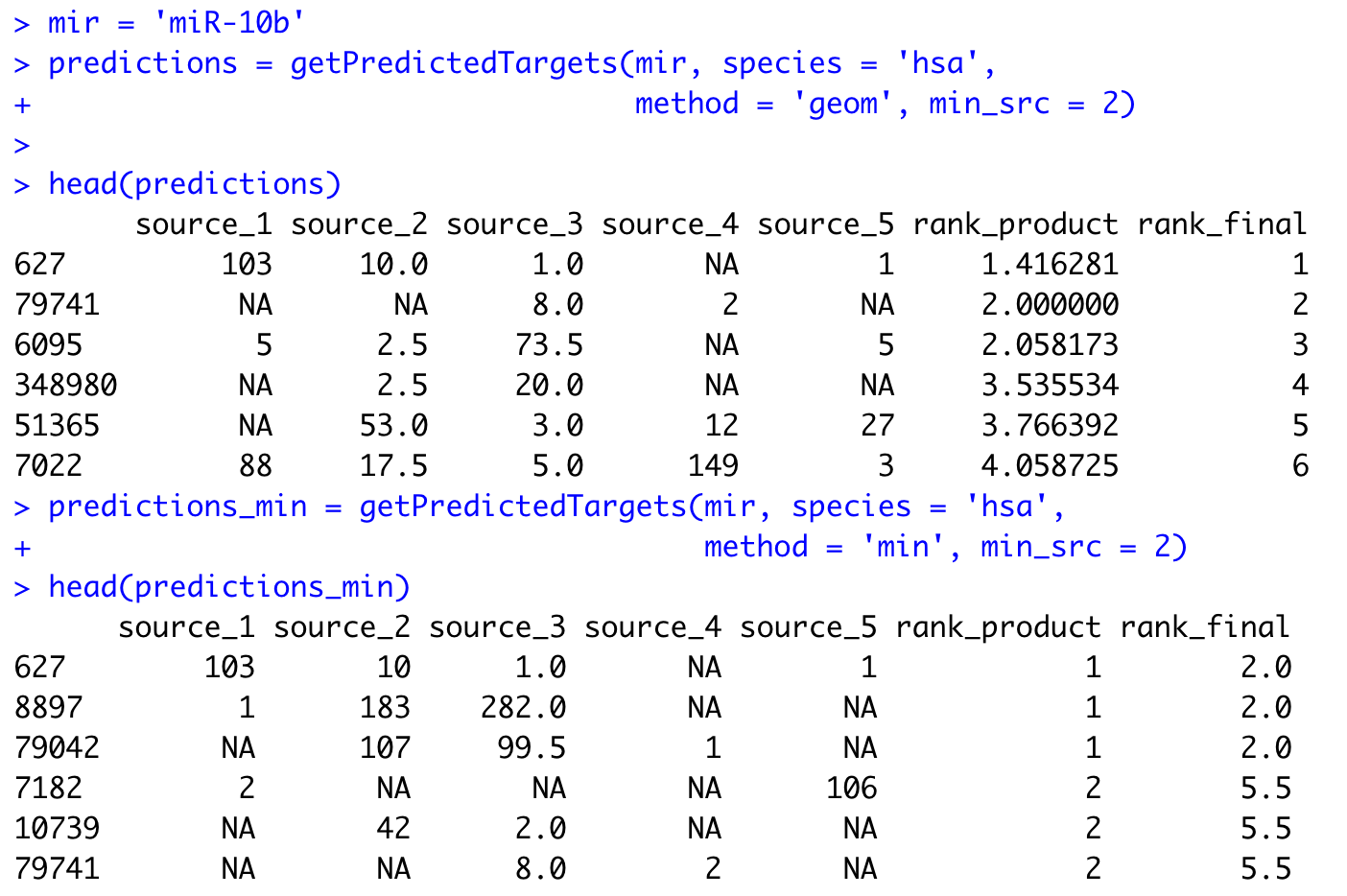
值得注意的是,默认是5个数据库都采用, i.e. c('pictar','diana','targetscan','miranda','mirdb'),不过也有可能某些miRNA在某些数据库其实是没有记录的,然后算法有3种:
min' is a minimum of ranks, 'max' is a maximum of ranks, and default 'geom'is based on geometric mean of the ranks which proves to be the most accurate method
所以我们就选择默认的geom即可,反正靶基因列表是一样的。
首先看miRNAtap包与multiMiR的结果的一致性
我们这里分别测试人和鼠的各一个miRNA,代码如下:
rm(list = ls())
library(miRNAtap)
library(topGO)
library(org.Hs.eg.db)
mir = 'hsa-miR-18a-3p'
tmp1 = getPredictedTargets(mir, species = 'hsa',
method = 'geom', min_src = 2)
head(tmp1)
library(multiMiR)
example1 <- get_multimir(mirna = 'hsa-miR-18a-3p', summary = TRUE)
tmp2=example1@data
intersect(rownames(tmp1),tmp2$target_entrez)
mir = 'mmu-miR-9-5p'
tmp1 = getPredictedTargets(mir, species = 'mmu',
method = 'geom', min_src = 2)
library(multiMiR)
example1 <- get_multimir(mirna = mir, org = 'mmu',summary = TRUE)
tmp2=example1@data
table(tmp2$database)
intersect(rownames(tmp1),tmp2$target_entrez)
简单看了看,两个R包里面记录的同一个miRNA的靶基因重合度还挺高的!
再看看它与miRSystem网页工具结果的差异
进入 http://mirsystem.cgm.ntu.edu.tw/ ,值得注意的是,该工具顺便对靶基因进行了生物学功能数据库的注释
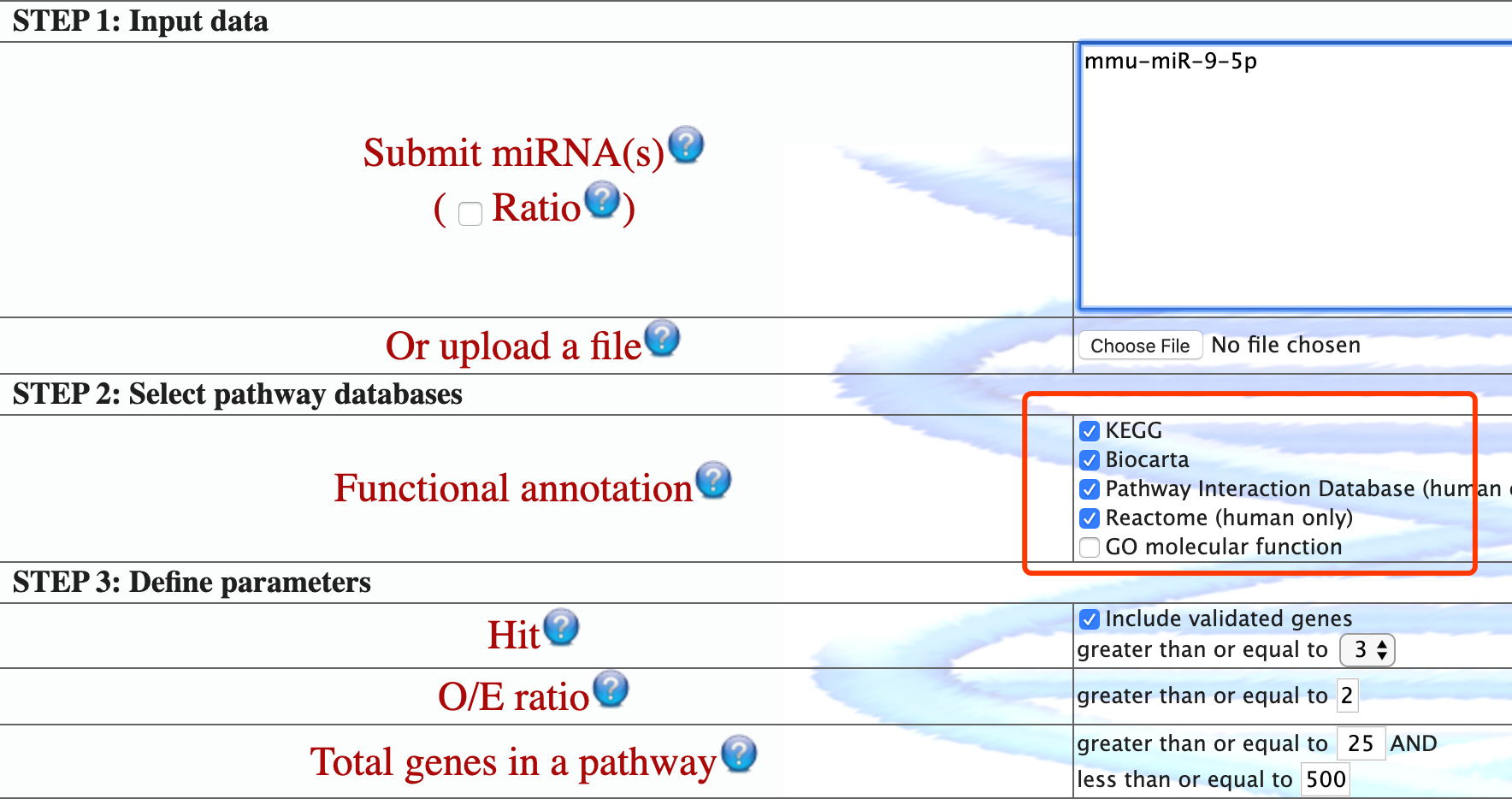
可以看到预测的靶基因是836个,有趣的是我们明明输入的是小鼠的miRNA,理论上靶基因应该是小鼠的,但是这个网页工具似乎是把人和鼠的基因模糊处理了。
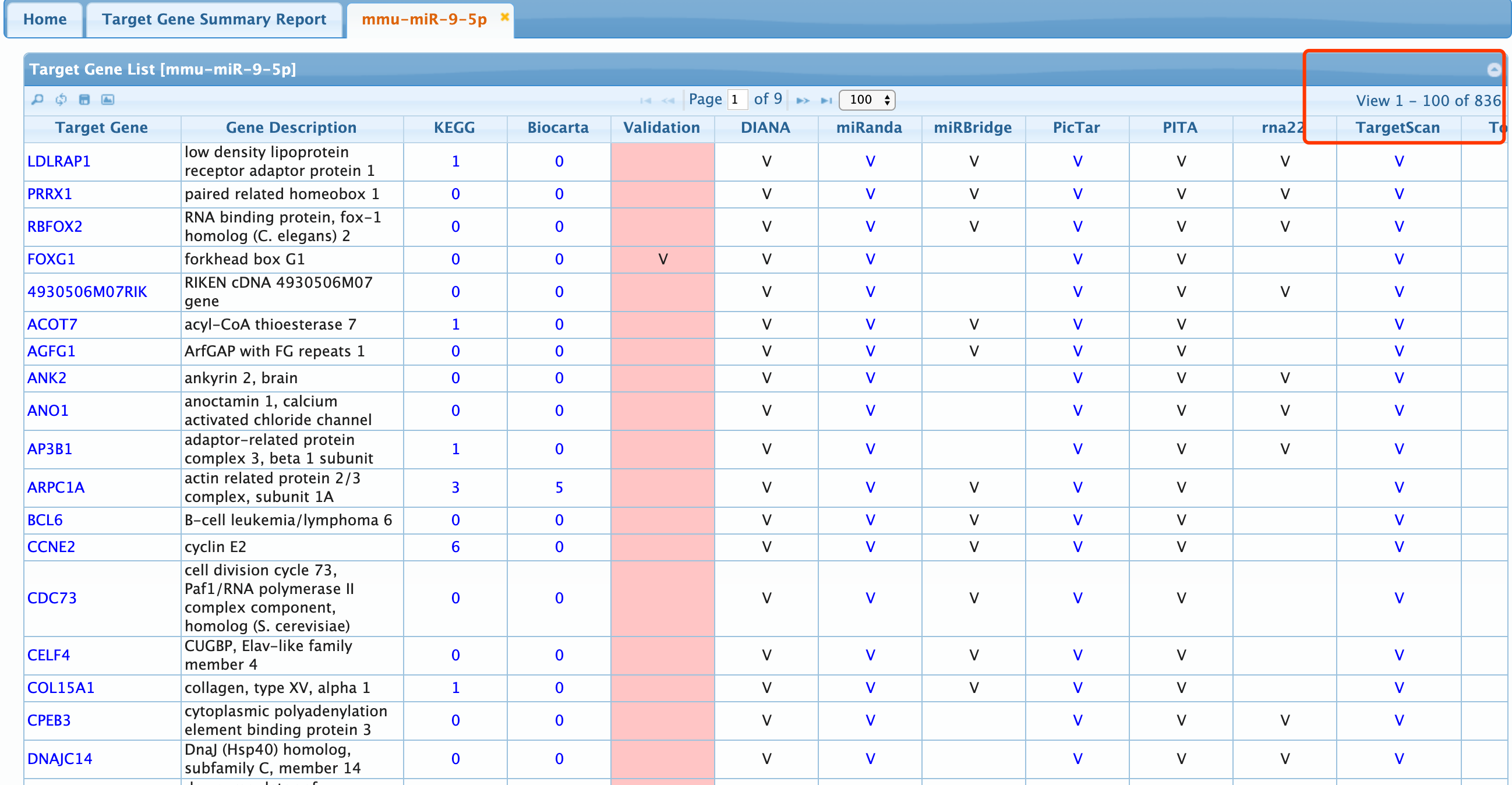
同样的,检查可以看到miRSystem网页工具结果和R包也是约30%的重合。
既然是预测,就不可能多个工具完全一致,所以目前主流的做法是,选择5个以上数据库支持的靶基因作为该miRNA的最后列表。
文末友情宣传
强烈建议你推荐我们生信技能树给身边的博士后以及年轻生物学PI,帮助他们多一点数据认知,让科研更上一个台阶:
- 生信爆款入门-全球听(买一得五)(第4期),你的生物信息学入门课
- 数据挖掘第2期(两天变三周,实力加量),医学生/临床医师首选技能提高课
- 生信技能树的2019年终总结 ,你的生物信息学成长宝藏
- 2020学习主旋律,B站74小时免费教学视频为你领路,还等什么,看啊!!!