不知道多少人看完了全网第一个单细胞课程(免费基础课程),免费学习地址在B站:https://www.bilibili.com/video/av38741055 而且完成结业考核20题:https://mp.weixin.qq.com/s/lpoHhZqi-_ASUaIfpnX96w ,作业里面有一个头颈癌单细胞的:
课程配套资料(主要是代码和PPT)文档在:https://docs.qq.com/doc/DT2NwV0Fab3JBRUx0
发表于2017年12月,在CELL杂志:Single-Cell Transcriptomic Analysis of Primary and Metastatic Tumor Ecosystems in Head and Neck Cancer 的文献,找到单细胞转录组表达矩阵,数据公布在 GSE103322 ,文件是 GSE103322_HNSCC_all_data.txt.gz,有86.0 Mb。
- 研究人员用
Smart-seq2建库方法得到的单细胞转录组数据经过QC后,留下了来自18名患者的5,902个细胞。 - 首先使用inferCNV可以分成2215个恶性细胞和3363个非恶性细胞
- 对进行3363个非恶性细胞表达矩阵分群,根据已知标记基因(自行搜索)的表达,注释并且找到B细胞,巨噬细胞,树突状细胞,肥大细胞,内皮细胞,成纤维细胞和肌细胞这8类!
重新看这个文章的时候,发现里面有这样的一个图表,如下:
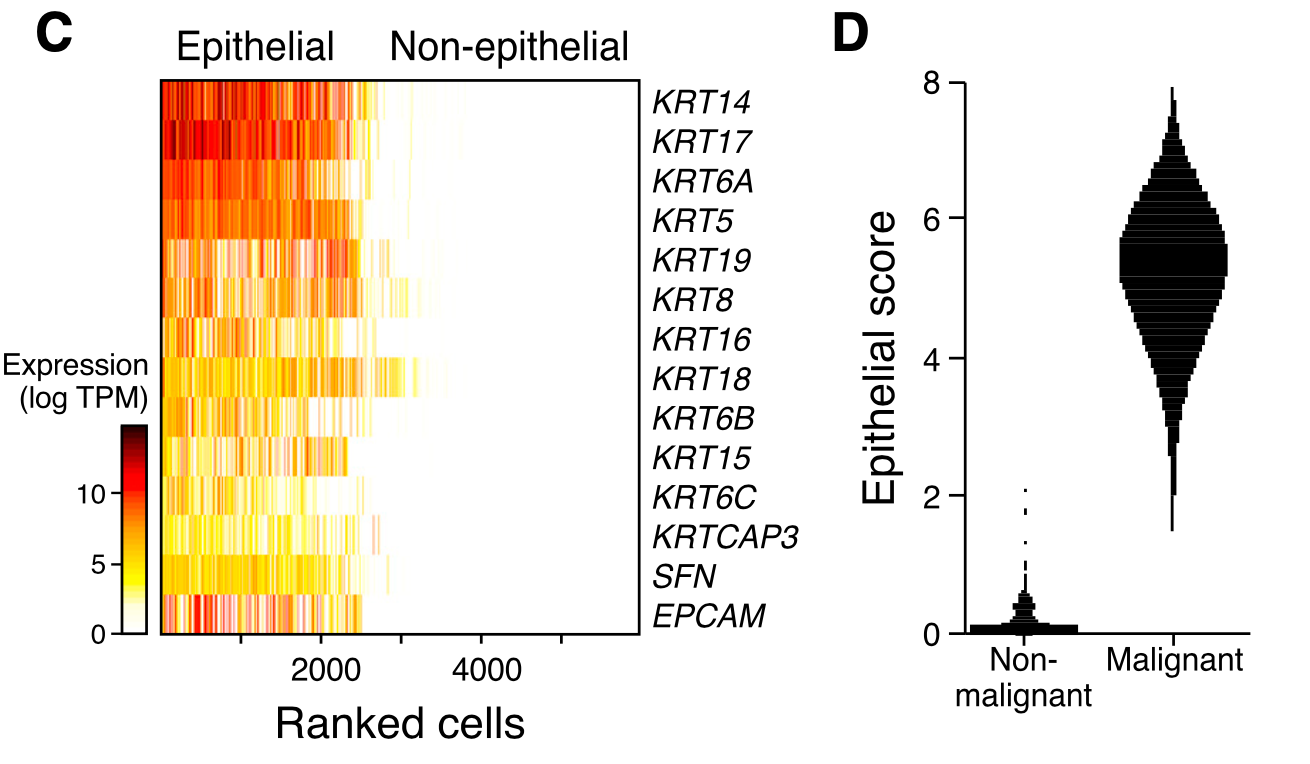
上皮细胞的标记基因如上所示,它们居然表达量如此相关,而且恰好与inferCNV的恶性与否对应上。所以我安排这个学徒作业,大家下载GSE103322_HNSCC_all_data.txt.gz文件,绘制上面这个图。
其中inferCNV区分细胞恶性与否,如果你对原理感兴趣可以看我以前在单细胞天地的教程:使用inferCNV分析单细胞转录组中拷贝数变异 ,但是第一步3个输入文件就制作失败,值得单独写教程强调一下这个解决方案。当然了,如果你还卡在第一步安装R包,请看我昨天在生信菜鸟团的教程:有些R包是你的电脑操作系统缺东西,但也有一些不是 。
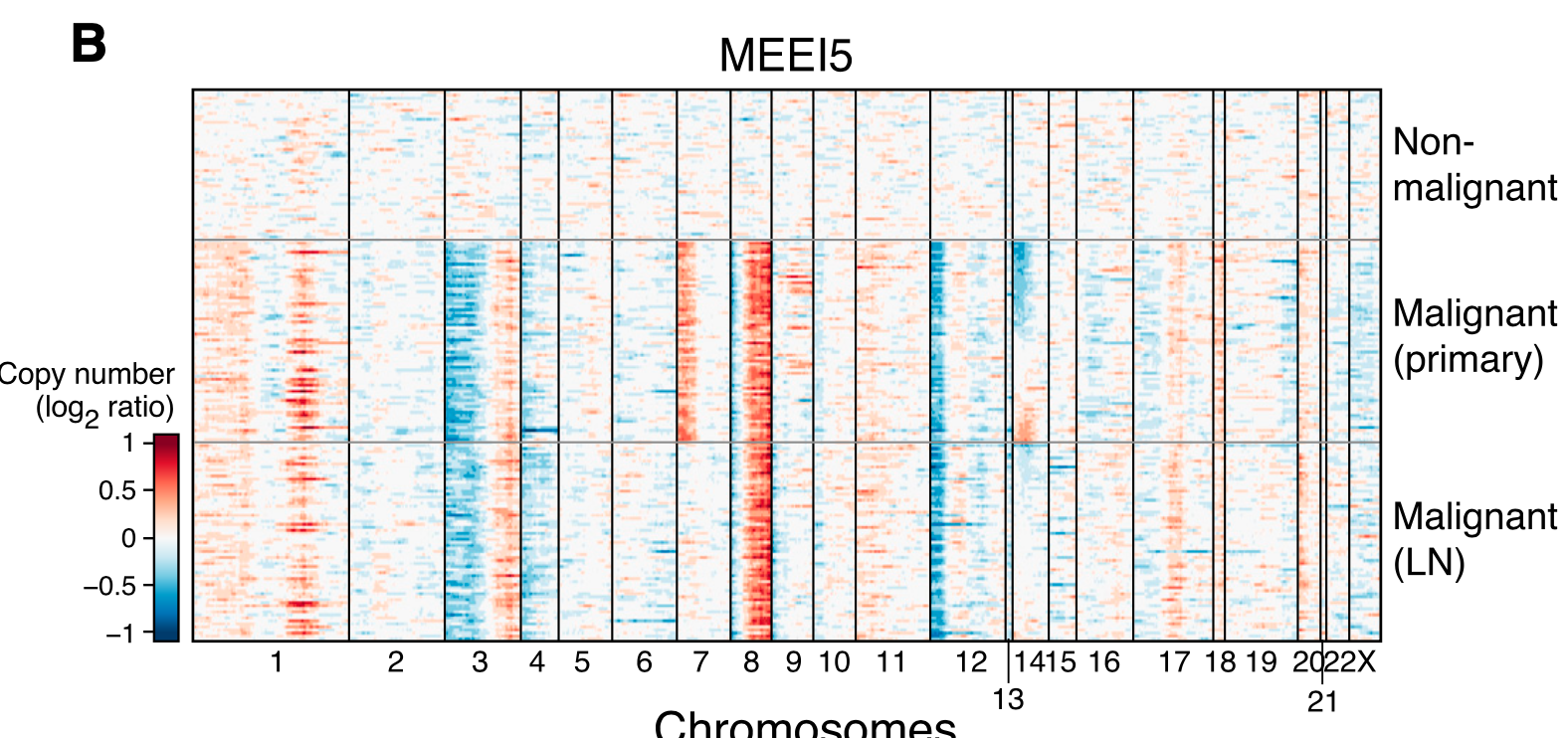
当然了,inferCNV非常难,你很大概率上是搞不定的哈,但是我相信这篇文章肯定是有inferCNV结果,你可以直接使用即可。文末友情宣传
强烈建议你推荐我们生信技能树给身边的博士后以及年轻生物学PI,帮助他们多一点数据认知,让科研更上一个台阶:
- 生信爆款入门-全球听(买一得五)(第4期),你的生物信息学入门课
- 数据挖掘第2期(两天变三周,实力加量),医学生/临床医师首选技能提高课
- 生信技能树的2019年终总结 ,你的生物信息学成长宝藏
- 2020学习主旋律,B站74小时免费教学视频为你领路,还等什么,看啊!!!