做知识分享这六年,太多粉丝需要帮助,所以我选择了全部无偿公开我所有技能,不仅仅是R和Linux基础,还包括多种ngs技术数据处理,甚至单细胞,这些教学视频都在B站。这样我就可以轻松上阵,但是千分之一左右粉丝是有科研经费,而且也没有时间精力慢慢看教程钻研摸索的,所以我们仍然是提供一点点学习辅导服务:
- 生信爆款入门-全球听(买一得五)(第4期),你的生物信息学入门课
- 数据挖掘第2期(两天变三周,实力加量),医学生/临床医师首选技能提高课
但是今早在粉丝交流群,看到这样的提问:
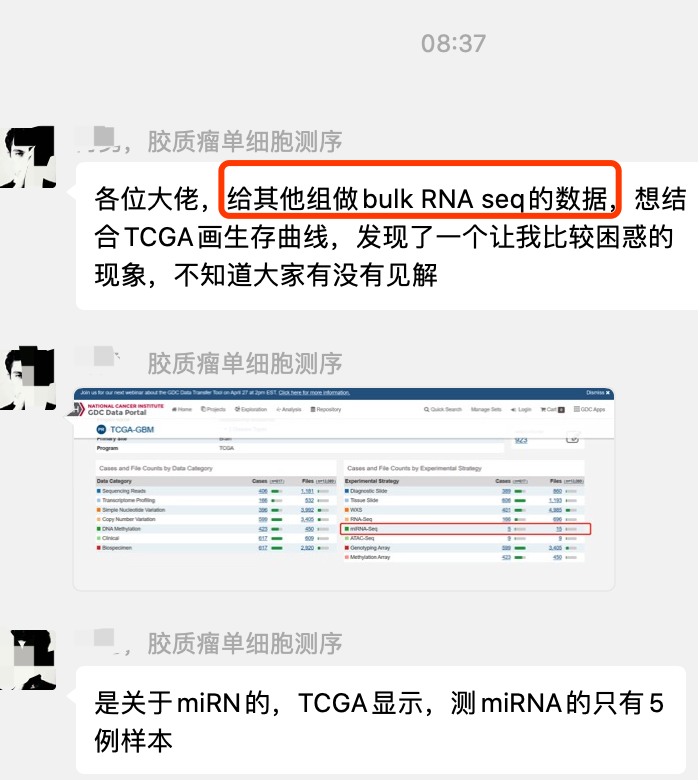
我心里一紧,这个最基础的学习辅导服务,大家也可以互相帮忙搞定了,不过善良的我还是回答了这个问题。
这个miRNA Expression Quantification (n=5) GDC Hub 是没有问题哦,之所以他想授课的文献里面有几百个人的miRNA数据,其实是表达芯片啦。有意思的是gdc官网不提供miRNA-array下载了,而且ucsc的xena里面也没有。不过,好在我写的教程足够多,其实还可以选择R语言的RTCGA包获取TCGA数据。
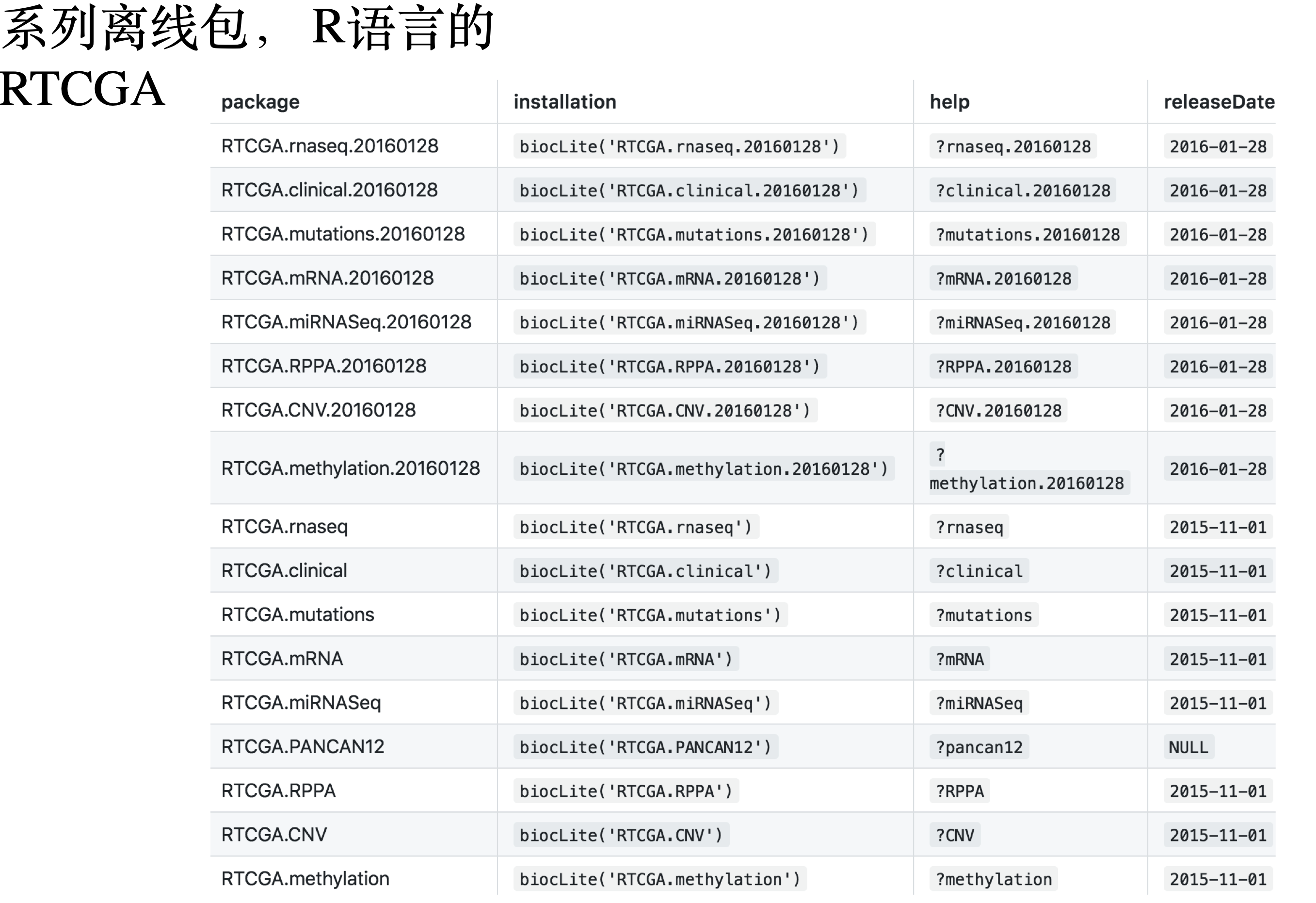
关于TCGA数据库
众所周知,TCGA数据库是目前最综合最全面的癌症病人相关组学数据库,包括:
- DNA Sequencing
- miRNA Sequencing
- Protein Expression array
- mRNA Sequencing
- Total RNA Sequencing
- Array-based Expression
- DNA Methylation
- Copy Number array
知名的肿瘤研究机构都有着自己的TCGA数据库探索工具,比如: - Broad Institute FireBrowse portal, The Broad Institute
- cBioPortal for Cancer Genomics, Memorial Sloan-Kettering Cancer Center
所以我挑选了部分,写了6个数据下载系列教程: - TCGA的28篇教程- 使用R语言的cgdsr包获取TCGA数据(cBioPortal)
- TCGA的28篇教程- 使用R语言的RTCGA包获取TCGA数据 (离线打包版本)
- TCGA的28篇教程-使用R语言的RTCGAToolbox包获取TCGA数据(FireBrowse portal)
- TCGA的28篇教程- 批量下载TCGA所有数据 ( UCSC的 XENA)
- TCGA的28篇教程-数据下载就到此为止吧
如果大家觉得自己在授课的时候耗费太多时间备课,而且还没有把握,可以考虑直接让你们的朋友参加我们的数据挖掘第2期(两天变三周,实力加量),医学生/临床医师首选技能提高课。全年滚动开课。我们全职的讲师和助教团队,可不能生锈和吃灰啊!文末友情宣传
强烈建议你推荐我们生信技能树给身边的博士后以及年轻生物学PI,帮助他们多一点数据认知,让科研更上一个台阶:
- 生信爆款入门-全球听(买一得五)(第4期),你的生物信息学入门课
- 数据挖掘第2期(两天变三周,实力加量),医学生/临床医师首选技能提高课
- 生信技能树的2019年终总结 ,你的生物信息学成长宝藏
- 2020学习主旋律,B站74小时免费教学视频为你领路,还等什么,看啊!!!