《道德经》“玄之又玄,众妙之门”
gsea和gsva算法大家应该是都很熟悉了,我也多次讲解:
不过里面有一个算法表格很难理解:
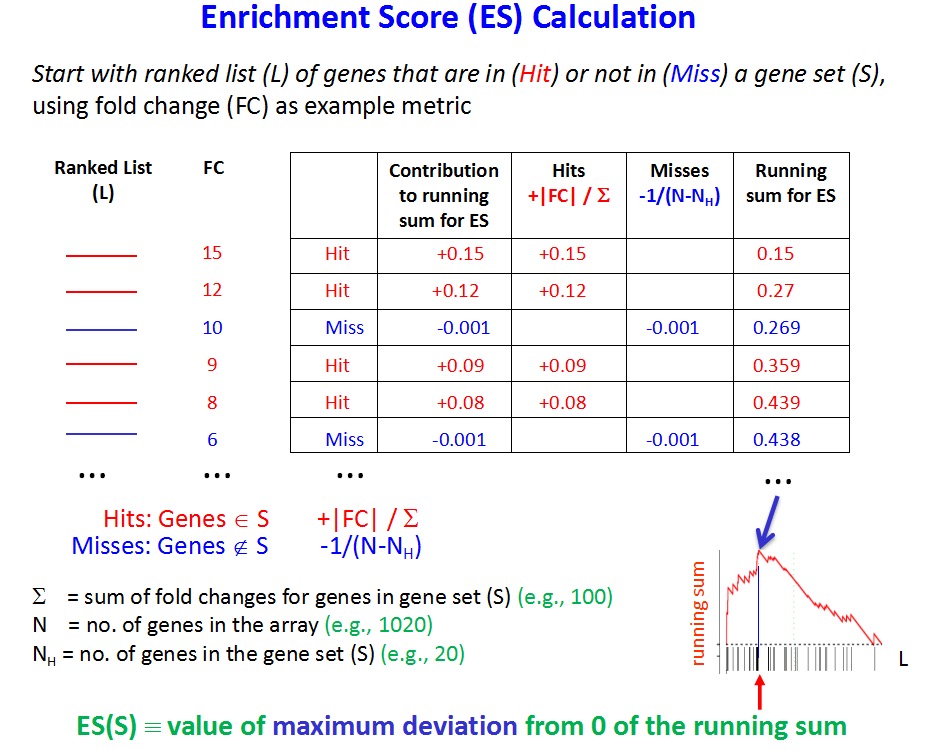
所以最近又有学徒咨询这个细节了,她的疑问是,为什么ssGSEA得分,跟其基因集里面的每个基因的表达量,并不是完全相关的,我这次写了一个例子来讲解。
模拟表达矩阵后,获得基因排序表格
比如我们模拟一下 26个基因的4个样本的表达量矩阵,然后查看自己感兴趣的3个基因在4个样本的顺序。
dat=data.frame(s1=rnorm(26),
s2=rnorm(26),
s3=rnorm(26),
s4=rnorm(26))
rownames(dat)=LETTERS
dat['A',]
pos=match(c('D','G','Z'),rownames(dat))
apply(dat, 2, function(x){
order(x,decreasing = T)[pos]
})
这里面的rnorm函数是随机的,所以你复制粘贴运行我这个代码,结果会有点不一样。
然后对它进行ssGSEA算法计算,如下:
library(GSVA)
library(GSEABase)
gmtFile='test.gmt'
tmp=paste(c('test','test','D','G','Z'),sep = '\t')
write.table(t(as.data.frame(tmp)),
file = gmtFile,sep = '\t',quote = F,row.names = F,col.names = F)
geneSet=getGmt(gmtFile,
geneIdType=SymbolIdentifier())
geneSet
ssgseaScore=gsva(as.matrix(dat),
geneSet, method='ssgsea', kcdf='Gaussian', abs.ranking=TRUE)
ssgseaScore
结果如下:
> ssgseaScore
s1 s2 s3 s4
test 0.03809468 0.2781415 -0.7218585 -0.6190109
> apply(dat, 2, function(x){
+ sort(order(x,decreasing = T)[pos])
+ })
s1 s2 s3 s4
[1,] 17 2 18 13
[2,] 23 23 20 17
[3,] 26 25 25 21
可以看到总共是26个基因的表达量,我们感兴趣的是3个基因,使用ssGSEA后:
- s1样本的ssGSEA打分是0.04,基因都排在后面
- s2样本的ssGSEA打分是0.27,因为有一个基因排的非常靠前!
- s3样本的ssGSEA打分是 -0.72, 因为3个基因都排在最后面
- s4样本的ssGSEA打分是 -0.62,同样的,3个基因都排在后面,但是略高于s3样本。
总之,如果要ssGSEA打分高,你的基因需要尽可能表达量排序靠前,反之,需要靠后使得ssGSEA打分降低,甚至为负值。
也就是说,仅仅是关心基因的表达量排序,而不是表达量本身。
真实数据
取一个项目的表达矩阵,然后提取11个基因组成的基因集的表达量热图,加上这11个基因的ssGSEA在这个表达量矩阵计算结果。
热图可视化如下:
n=t(scale(t( mat[treg,] )))
n[n>2]=2
n[n< -2]= -2
n[1:4,1:4]
ac=data.frame(gsea=as.numeric(ssgseaScore))
rownames(ac)=colnames(n)
pheatmap(n,annotation_col = ac,
show_colnames =F,show_rownames = T)
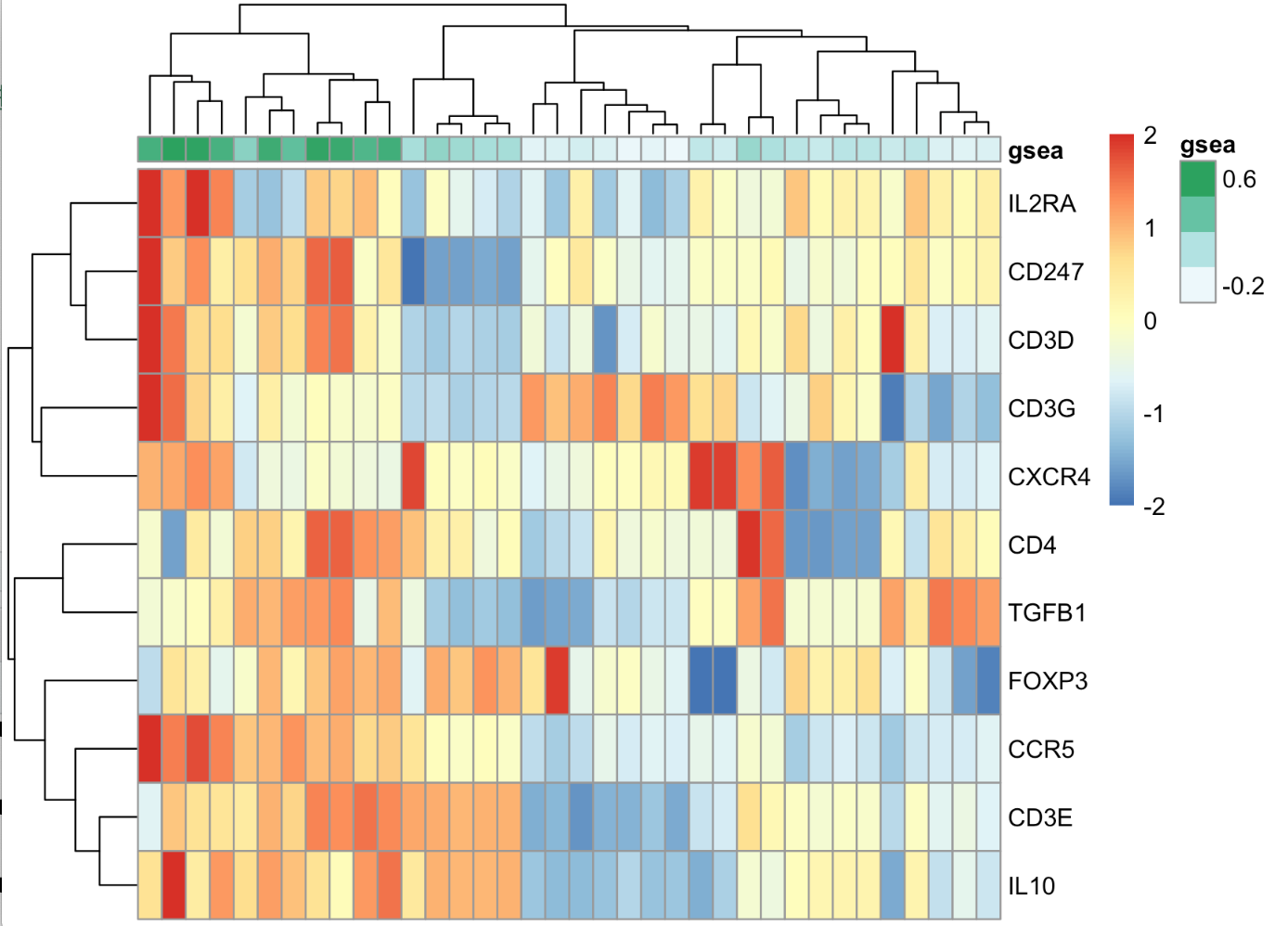
可以看到,ssGSEA得分,跟基因表达量,还是有关系的,毕竟表达量高基因排名也高啊!
你大概率上无需把最开始那个算法PPT里面的公式写出来
首先FC在单个样本里面是不存在的,但是可以把表达量进行zscore,代入公式,看看能不能计算得到差不多的ssGSEA值。
欢迎邮件交流你的代码,我的邮箱 jmzeng1314@163.com
文末友情宣传
强烈建议你推荐我们生信技能树给身边的博士后以及年轻生物学PI,帮助他们多一点数据认知,让科研更上一个台阶:
- 生信爆款入门-全球听(买一得五)(第4期),你的生物信息学入门课
- 数据挖掘第2期(两天变三周,实力加量),医学生/临床医师首选技能提高课
- 生信技能树的2019年终总结 ,你的生物信息学成长宝藏
- 2020学习主旋律,B站74小时免费教学视频为你领路,还等什么,看啊!!!