最近看到某公司宣传他们的产品,是lncRNA的芯片,文章是2015发表的,研究思路很清晰:
- 119个食管癌病人的肿瘤组织和配对样品的lncRNA芯片数据,在GSE53624
- 芯片平台比较老旧了,是Agilent human lncRNA+mRNA array V.2.0
- 把119个食管癌病人数据拆分成为 training (n=60) and test (n=59) 数据集
- 在训练集里面,筛选 4874 lncRNAs表达量有变化的,然后差异分析得到909个有表达差异的lncRNAs。
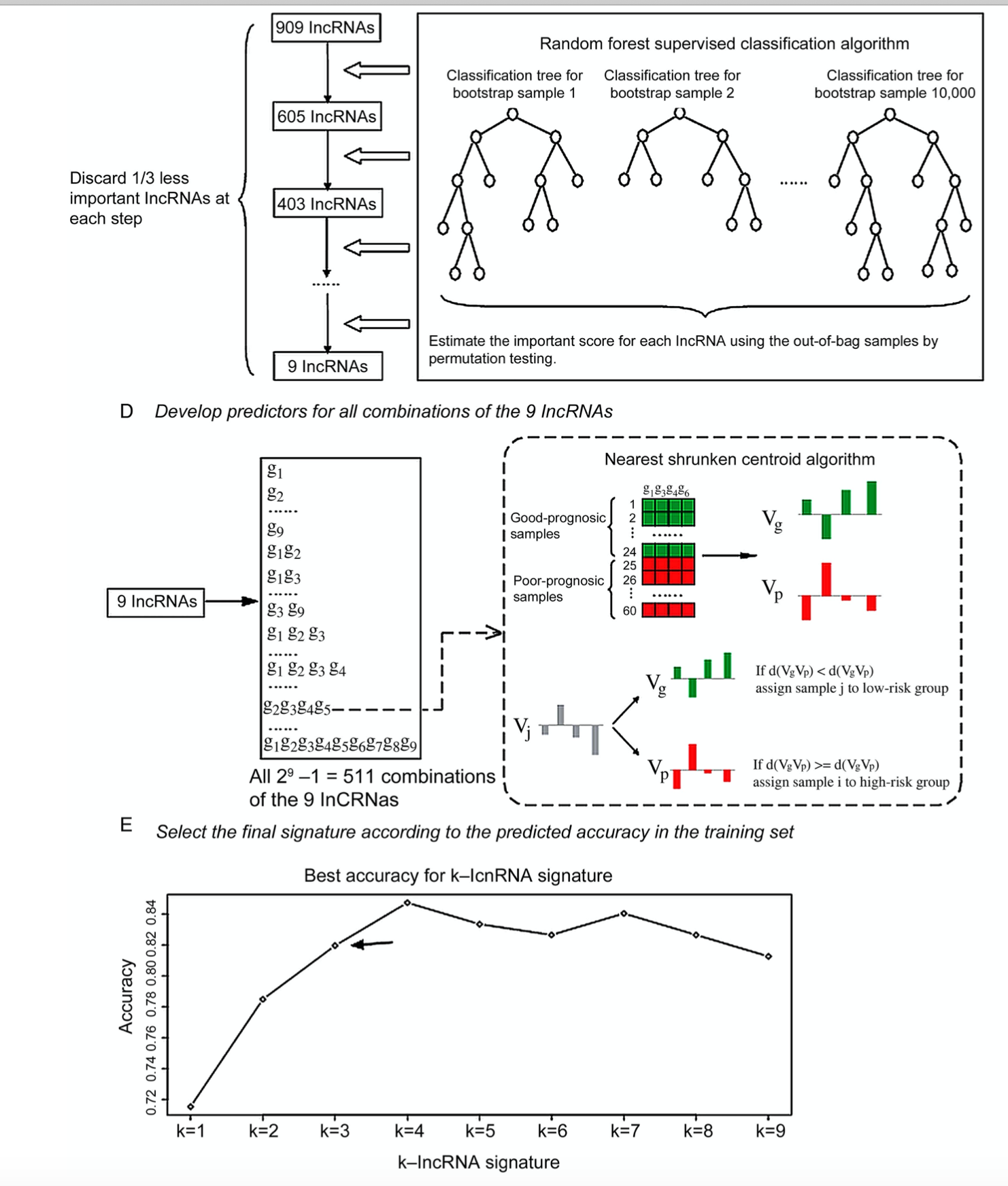
- 随机森林,筛选9个跟生存最相关的,然后排列组合,定下来3个lncRNAs。
- 寻找3个lncRNAs所调控的蛋白编码基因并且进行注释。
看起来似乎是Agilent和CBC公司合作,所以芯片平台是:Agilent-038314 CBC Homo sapiens lncRNA + mRNA microarray V2.0 (Feature Number version) ,从有表达差异的基因列表里面筛选到最后的3个lncRNA组成的食管癌诊断分类器基因集,过程比较复杂,如下:
有趣的是,这篇文章的数据非常多,还有个:https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE53622
所以很多数据挖掘文章重新分析了它,比如: - Immune signature profiling identified predictive and prognostic factors for esophageal squamous cell carcinoma. Oncoimmunology 2017;6(11):e1356147. PMID: 29147607
- AJUBA promotes the migration and invasion of esophageal squamous cell carcinoma cells through upregulation of MMP10 and MMP13 expression. Oncotarget 2016 Jun 14;7(24):36407-36418. PMID: 27172796
学徒作业
大家可以去tcga数据库下载食管癌的转录组数据,提取分离lncRNA的部分,走同样的诊断建模流程,看看得到的lncRNA是否作者的3个lncRNA有交叉。这里面变量很多:
- 首先,两个队列的人群地域差异
- 其次,lncRNA芯片和测序技术差异
- 还有,肿瘤组织和癌旁配对问题,两个组数据量问题
对大家来说,比较难的地方就是如何取最小可诊断的lncRNA集合。
可以参考我的4个小时TCGA肿瘤数据库知识图谱视频教程,其中总共使用了四种算法构建模型: - cox(可做单因素和多因素)
TCGA的cox模型构建和风险森林图 - lasso回归
用lasso回归构建生存模型+ROC曲线绘制 - 随机森林
听起来很霸气用起来并不难的随机森林 - 支持向量机
听起来很霸气用起来并不难的 支持向量机
不管用了那种算法,核心都只是几句代码而已。
点击下面的阅读原文,马上就可以学习起来啦~
https://www.bilibili.com/video/BV1db411L7GX文末友情宣传
强烈建议你推荐我们生信技能树给身边的博士后以及年轻生物学PI,帮助他们多一点数据认知,让科研更上一个台阶:
- 生信爆款入门-全球听(买一得五)(第4期),你的生物信息学入门课
- 数据挖掘第2期(两天变三周,实力加量),医学生/临床医师首选技能提高课
- 生信技能树的2019年终总结 ,你的生物信息学成长宝藏
- 2020学习主旋律,B站74小时免费教学视频为你领路,还等什么,看啊!!!