但是,能走通流程,并不代表可以有好的生物学解释
前些天我发布了 cellranger更新到4啦,提到了可以免费做一下10X的单细胞转录组数据上游分析,反正刚刚购买的服务器闲着也是闲着。然后,马上就有粉丝寄给我一个2.4T硬盘,里面有24个10X单细胞转录组原始测序数据,真的是超级大,两个星期过去了,我还没有跑完。可能是自己的服务器有点辣鸡吧,或者说因为我使用的是Windows的ubuntu子系统,而不是纯正的ubuntu这样的Linux?
当然,这不是重点,麻烦的事情是粉丝仅仅寄给我数据和md5,我校验后文件都是完整的,就直接上流程了,结果,打开一个运行日志后才发现不太对劲额。
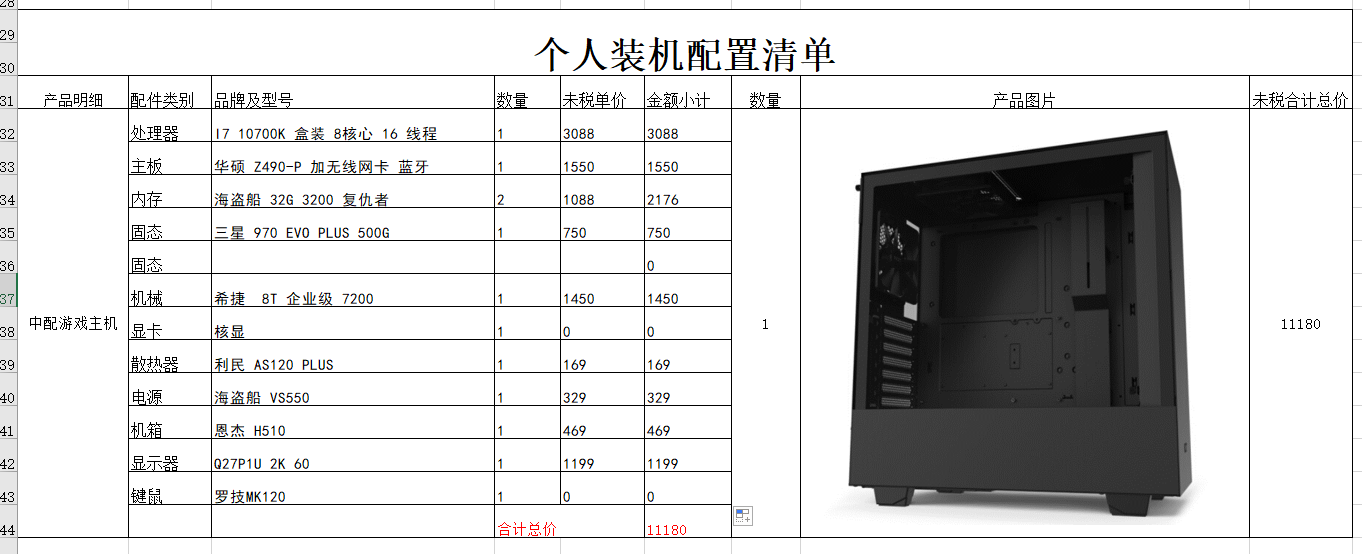
粗看起来是很漂亮的结果,本次10x单细胞转录组得到的细胞数量是5548,符合3到5千个细胞的预期,然后每个细胞的平均测序reads数量是56K,也符合预期,就是每个细胞的平均基因数量有点少额。(当时我没有意识到有问题)
其实后续seurat流程也很顺利,很漂亮,但是呢,没有线粒体基因,所以我回过去再看了看10x的cellranger count 流程报告,有一个很小的警告,如下:

我略微思考了一下,因为是比对,所以参考基因组的版本是无所谓的,比对不到参考基因组,肯定是物种选错了。果然,回邮件询问了一下粉丝,把小鼠单细胞数据错当做了是人类数据,就匆匆忙忙跑流程了。唉···
但是,为何我用错了参考基因组,把小鼠的单细胞转录组测序数据比对到人类上面,但是仍然是有10%的成功率呢?而且前面的表达矩阵,后续seurat流程,都有模有样啊, 分群也挺好的,高表达量基因都没有问题,如果不回过头检查,难道就这样将错就错了吗?
当然了,后面我仍然是选择了正确的参考基因组,重新跑了一下流程,报告如下:
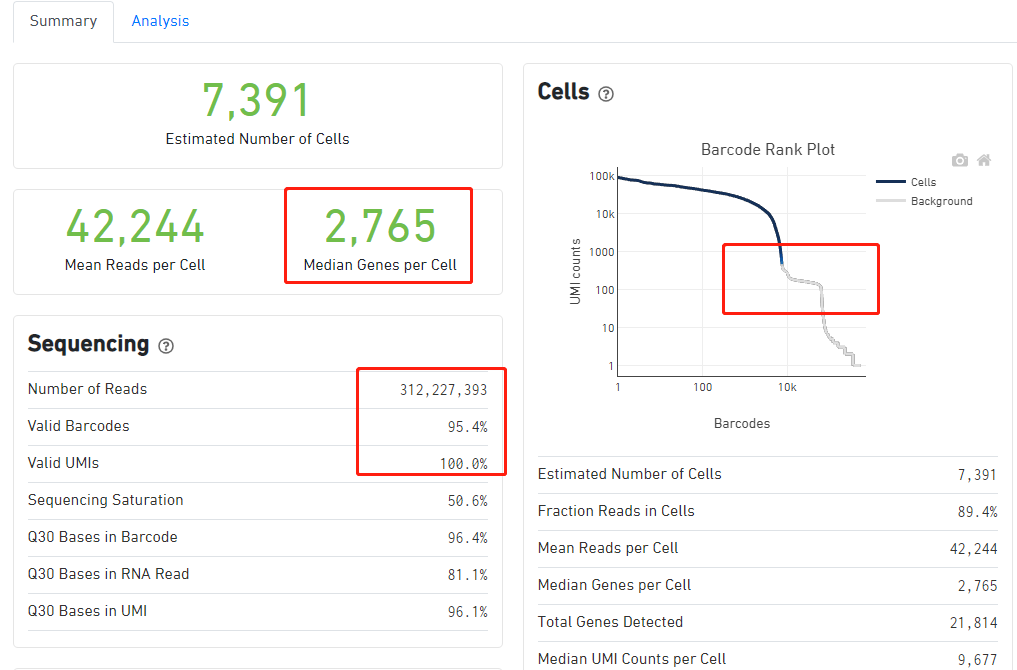
首先,细胞数量由5000多增加到了7000多,然后每个细胞的平均检测到的基因数量由600多变成了2700多。
一些思考
- 小鼠单细胞转录组数据里面的10%的reads是可以比对到人的参考基因组,那么做PDX模型这样的人鼠混合数据的时候就必然有一些reads天然就多比对咯?
- 小鼠单细胞转录组数据里面的10%的reads是可以比对到人的参考基因组,这些reads落入了几百个基因里面,这些基因进行生物学功能基因集富集,是不是有一些含义?
不知道是否有人愿意花费时间去具体探索它,号外:生信技能树知识整理实习生招募,长期招募,我觉得就可以加入这个小课题哦。