看到一个比较古老的生物学领域的研究文献,文章标题是:《Analysis of CUL-5 expression in breast epithelial cells, breast cancer cell lines, normal tissues and tumor tissues》,于2003年发表在 Molecular Cancer 杂志,如题,研究者就分别在乳腺癌和正常乳腺的细胞系里面测定了几个基因的表达量情况。
- The Journal Impact 2019-2020 of Molecular Cancer is 11.350, which is just updated in 2020.
研究课题涉及到的癌症和正常细胞系是:
- breast epithelial cells (HMEC, MCF-10A),
- breast cancer cells (MCF-7, MDA-MB-231)
检测的基因是 CUL-5: 以及 other CUL family members (CUL-1, -2, -3, -4A, and -4B) 。实际上哺乳动物有: seven related cullins (Cul1, Cul2, Cul3, Cul4A, Cul4B, Cul5 and Cul7)
也有临床队列验证:
- The cancer profiling array revealed that 82% (41/50) of the breast cancers demonstrated a decrease in CUL-5 expression versus the matched normal tissue.
- For the 50 cases of matched breast tissue there was a statistically significant ~2.2 fold decreased expression of CUL-5 in tumor tissue versus normal tissue (P < 0.0001).
技术是 RT-PCR :,使用 :
- a 411-bp PCR product for ribosomal protein S9
- 905-bp mouse GAPDH cDNA (Ambion)
- actin (~42 kDa)
实际上看表达量远不止 RT-PCR 技术,在临床病人队列,现在用得更多的是: Tissue microarray (TMA) 和 immunohistochemistry analysis (IHC)。而且文章里面的 RT-PCR ,进化成为了 Quantitative real-time polymerase chain reaction (qPCR) ,实时荧光定量PCR.值得注意的是不同物种的不同组织器官样品的qRT-PCR内参基因也不一样。
我前些天在《生信技能树》介绍过一个网页工具DrBioRight ,见:聊个天就把生存分析给做了?,现在演示一下聊个天就把这个课题做了,进入主页:(https://drbioright.org),然后尝试使用以下对话进行聊天:
CUL5 gene expression in breast related cellline of CCLE database
发现这个网页工具DrBioRight 简直就是一个人工智障啊,还不如自己去CCLE数据库官网去查询:
https://portals.broadinstitute.org/ccle/page?gene=CUL5
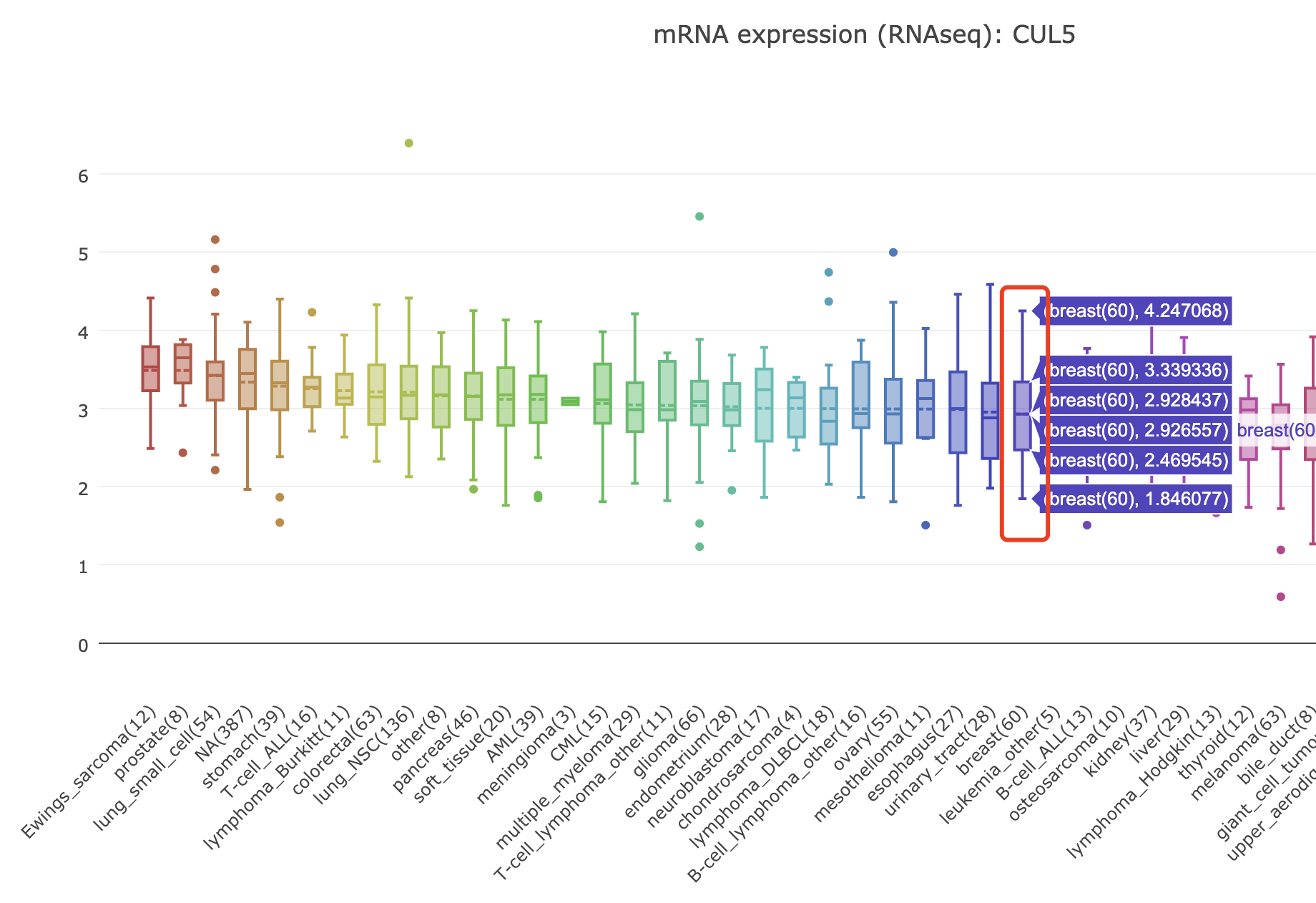
而且可以下载数据自己去R里面进行统计可视化。
不知道是不是我还是不是很会玩那个网页工具DrBioRight ,目前看来我的需求是很难通过聊天被满足的。我仍然是推荐大家自己学习R语言把R的知识点路线图搞定,如下:
- 了解常量和变量概念
- 加减乘除等运算(计算器)
- 多种数据类型(数值,字符,逻辑,因子)
- 多种数据结构(向量,矩阵,数组,数据框,列表)
- 文件读取和写出
- 简单统计可视化
- 无限量函数学习
起码需要一个月!!!详见:《生信分析人员如何系统入门R(2019更新版)》。
同样的策略今年仍然是有效
我看到公众号《上海国际乳腺癌论坛(SIBCS)》发布的 :《切断三阴性乳腺癌细胞生长的营养来源》就提到了一个类似的研究:
比如2020年10月8日,英国癌症研究基金会《英国癌症杂志》在线发表英国牛津大学、诺丁汉大学、伦敦大学癌症研究院的研究报告,探讨了不同谷氨酰胺转运蛋白对不同乳腺癌细胞的影响。就是分析了5种钠偶联中性氨基酸转运蛋白(SLC1A4、SLC1A5、SLC7A5、SLC38A1、SLC38A2)在谷氨酰胺缺乏或药物刺激下对6种乳腺癌细胞(激素受体阳性MCF7、激素受体阳性T47D、HER2阳性SKBR3、三阴性HCC1806、三阴性MDA-MB-231、三阴性MDA-MB-468)的作用和分布。
结果,SLC38A2(又称SNAT2)被确定为高表达于6种乳腺癌细胞的氨基酸转运蛋白。通过自噬作用,SLC38A2可被溶酶体降解。对于谷氨酰胺敏感细胞,敲低SLC38A2编码基因表达可减少谷氨酰胺摄入、诱发自噬作用、引起氧自由基产生、抑制乳腺癌细胞生长。
142例SLC38A2高表达与1338例SLC38A2低表达乳腺癌患者相比,乳腺癌相关生存显著较差(P=0.004);其中,33例SLC38A2高表达与75例SLC38A2低表达三阴性乳腺癌患者相比,乳腺癌相关生存显著较差(P=0.02)。
该研究结果表明,SLC38A2高表达可促进乳腺癌(尤其三阴性乳腺癌)细胞对谷氨酰胺的依赖性和抗氧化应激,而且患者生存结局较差,SLC38A2有望成为抑制谷氨酰胺依赖性乳腺癌(尤其三阴性乳腺癌)生长的新靶点。
学徒作业目录如下:
- 生信编程直播课程优秀学员作业展示1
- 生信编程直播课程优秀学员学习心得及作业展示3
- 生信编程直播课程优秀学员作业展示2
- 给学徒的GEO作业
- 这个WGCNA作业终于有学徒完成了!
- 上次说的gmt函数(学徒作业)
- 拖后腿学徒居然也完成作业,理解RNA-seq数据分析结果
- 肿瘤外显子视频课程小作业
- ChIPseq视频课程小作业
- Agilent芯片表达矩阵处理(学徒作业)
- 学徒作业:TCGA数据库单基因gsea之COAD-READ
- 学徒作业-在CCLE数据库里面根据指定基因在指定细胞系里面提取表达矩阵
- 学徒作业-指定基因在指定组织里面的表达量热图
- 学徒作业-我想看为什么这几个基因的表达量相关性非常高
- 学徒作业:给你8个甲基化探针, 你在tcga数据库进行任意探索
- 学徒作业-根据我的甲基化视频教程来完成2015-NPC-methy-GSE52068研究
- RNA芯片和测序技术的比较(学徒作业)
- 学徒作业-单基因的tcga数据挖掘分析
- ATCC终于出来了organoids资源
- 拿到7个DDR通路的基因集-学徒作业
- 绘图本身很简单但是获取数据很难
- 都说lncRNA只有部分具有polyA尾结构,请证明
- 学徒作业-hisat2+stringtie+ballgown流程
- 学徒任务-探索DNA甲基化的组织特异性
- 用WES和RNA-Seq数据提取到的somatic SNVs不一致
- 《GEO数据挖掘课程》配套练习题