文章《BRCA1 Promoter Methylation Status in 1031 Primary Breast Cancers Predicts Favorable Outcomes Following Chemotherapy》,链接是:https://academic.oup.com/jncics/article/4/2/pkz100/5673392 我看到文章里面提到了乳腺癌患者的1031 primary tumors的测序数据,下意识的以为是一个TCGA数据库挖掘。
本来呢,还在奇怪,TCGA数据库里面的乳腺癌患者的放化疗信息应该是没有那么全吧。等我看完摘要才明白,原来是研究者自己招募的病人队列,来自于Iceland between 1976 and 2007. 而且Chemotherapy信息很丰富,包括 cyclophosphamide, methotrexate, and fluorouracil 。
这个研究,科学家们想解决的问题是;whether BRCA1 promoter methylation translates to clinical benefits from the use of DNAdamaging agents in patients.
招募的病人队列
- 1031 primary tumors
- carries of germline mutations in either BRCA1 (n = 5) or BRCA2 (n = 61)
- BRCA1 methylation was identified in 29 tumors
- along with 91 normal breast tissue samples
涉及到的甲基化位点,是 Four CpG sites were analyzed at genetic positions:
- chr17: 43125409(GRCh38.p7),
- chr17: 43125411(GRCh38.p7),
- chr17: 43125419(GRCh38.p7),
- chr17: 43125427(GRCh38.p7).
这里定义 BRCA1 methylated 样品的标准是: Tumor samples were considered to be BRCA1 methylated at median greater than 10% methylation across the four CpGs (median values).
文章的图表超级简陋,我感觉的R语言初学者使用base plot绘制而成,如下所示:
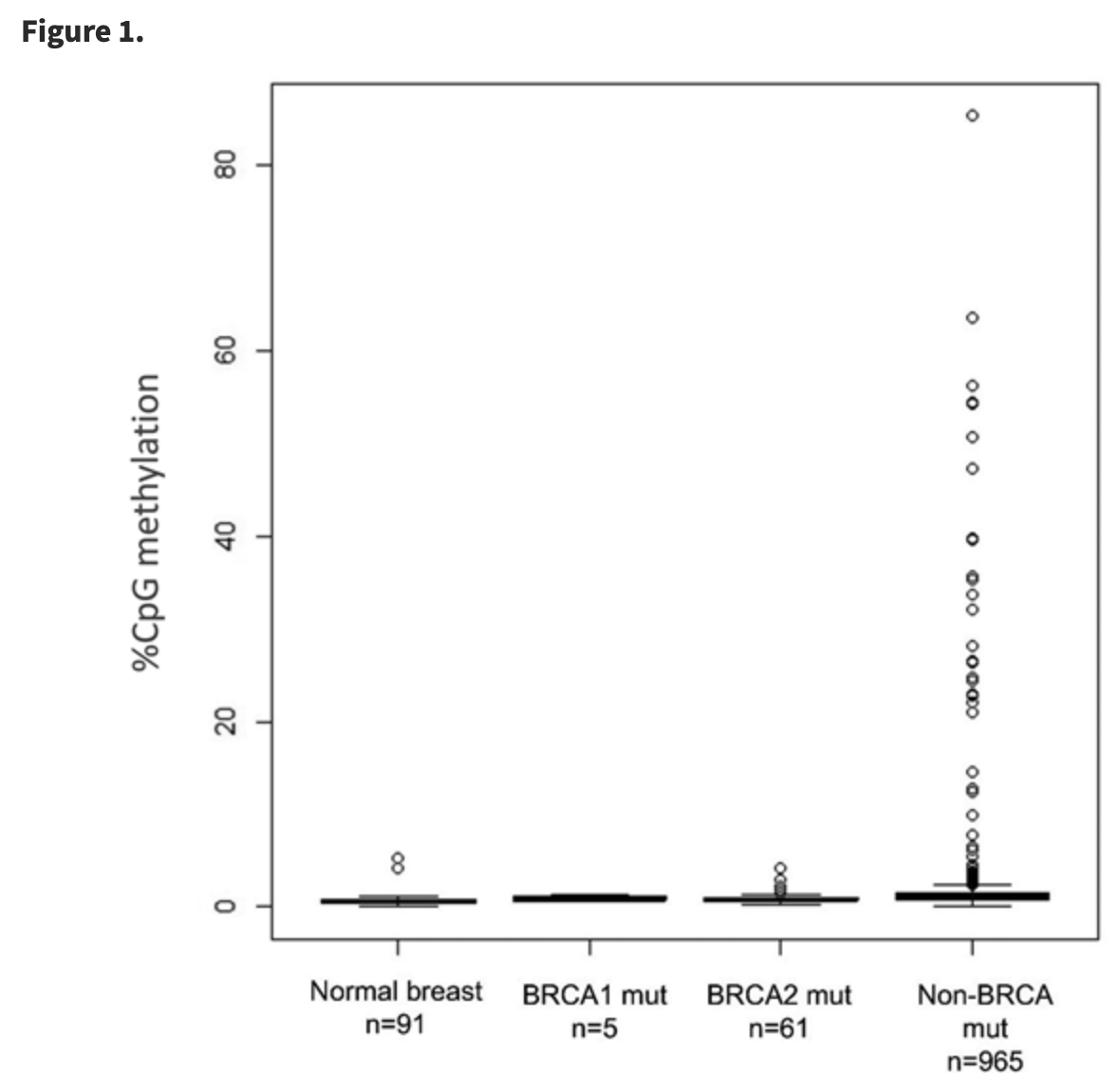
这个是作者最重要的研究发现,不同分组的样品的 BRCA1–promoter CpG methylation 百分比不一样。
临床属性关联
其实不管是自己医院收集的病人队列,还是TCGA的,临床信息都是可以做到很完善。
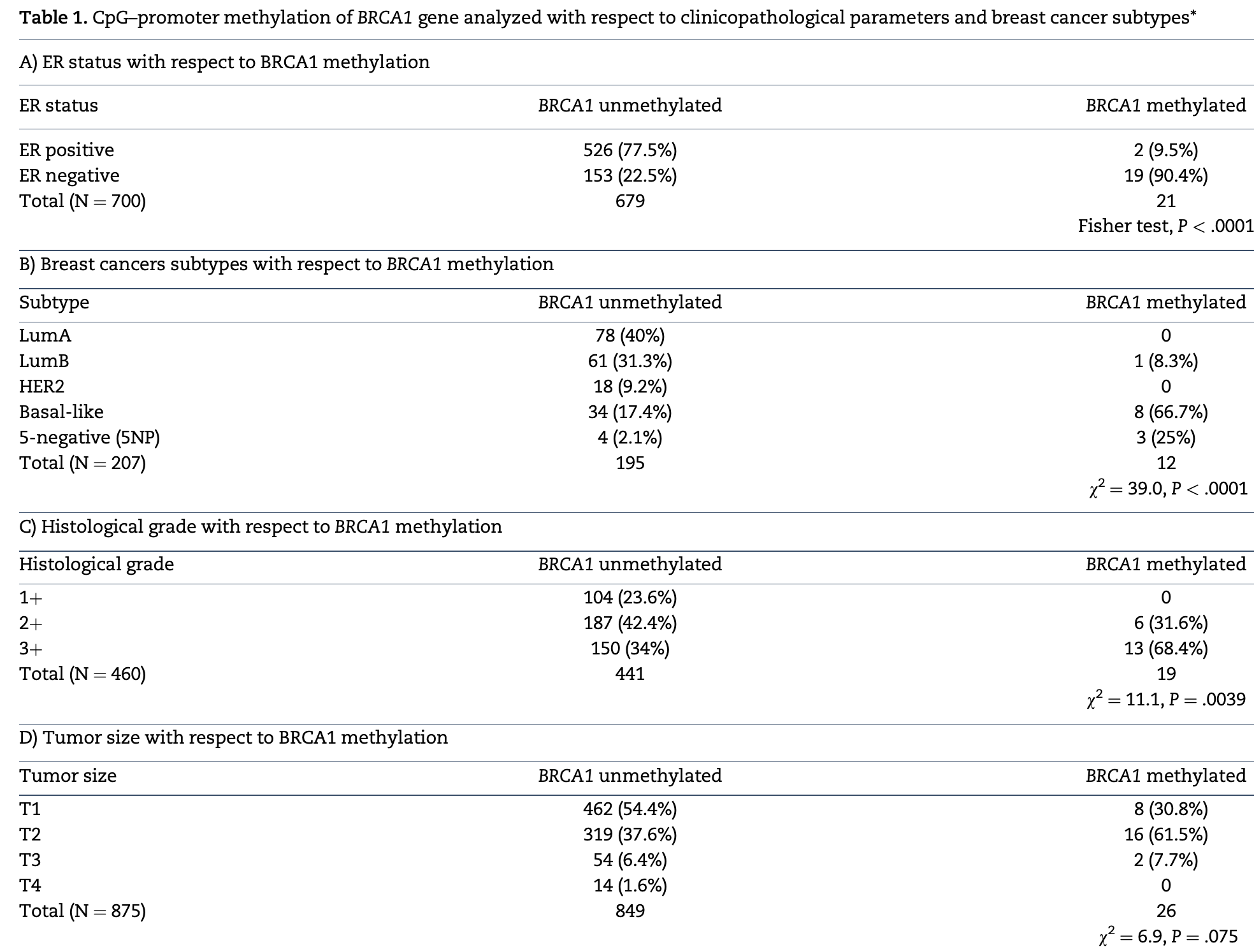
这里,研究者们列出来了常见的临床属性分组后的 BRCA1–promoter CpG methylation 情况。
临床意义
主要就是生存分析啦,这里可以看到 BRCA1–promoter CpG 有甲基化的不管是否经受了化疗,都要生存情况好一点。

不过,BRCA1–promoter CpG 有甲基化的病人数量有点太少了。
学徒作业
如果要得出文章的研究结果,其实大可不必费劲招募一千多个乳腺癌患者,完完全全是可以进行tcga数据库挖掘的。这里就布置成为一个学徒作业哈:
需要去TCGA数据库下载乳腺癌患者队列的临床信息,以及甲基化信号值矩阵,通常是贝塔值,通常是按照2-8原则把BRCA1–promoter CpG 甲基化信号值进行分类。如果你不理解甲基化数据呢,可以读一下我在生信技能树的甲基化系列教程,目录如下:
- 01-甲基化的一些基础知识.pdf
- 02-甲基化芯片的一般分析流程.pdf
- 03-甲基化芯片数据下载的多种技巧.pdf
- 04-甲基化芯片数据下载如何读入到R里面.pdf
- 05-甲基化芯片数据的一些质控指标.pdf
- 06-甲基化信号值矩阵差异分析哪家强.pdf
- 07-甲基化芯片信号值矩阵差异分析的标准代码.pdf (微信交流群在这里)
- 08-TCGA数据库的各个癌症甲基化芯片数据重新分析.pdf
- 09-TCGA数据库的癌症甲基化芯片数据重分析.pdf
- 10-TCGA数据辅助甲基化区域的功能研究.pdf
- 11-按基因在染色体上的顺序画差异甲基化热图.pdf
- 850K甲基化芯片数据的分析.pdf
- 使用DSS包多种方式检验差异甲基化信号区域.pdf
然后就可以看我在B站免费分享的视频课程《甲基化芯片(450K或者850K)数据处理 》
- 教学视频免费在:https://www.bilibili.com/video/BV177411U7oj
- 课程配套思维导图:https://mubu.com/doc/1cwlFgcXMg
- 甲基化芯片相关资料,https://share.weiyun.com/42a9e78c2dd5367f3427e86c5c99baa1 按需下载,无需整个文件夹全部下载
- 表观全部资料,https://share.weiyun.com/5tg6pIn 同样是按需下载,无需整个文件夹全部下载
需要做同样的生存分析,我在生信技能树多次分享过生存分析的细节;
- 基因表达量高低分组的cox和连续变量cox回归计算的HR值差异太大?
- 学徒作业-两个基因突变联合看生存效应
- TCGA数据库里面你的基因生存分析不显著那就TMA吧
- 对“不同数据来源的生存分析比较”的补充说明
- 批量cox生存分析结果也可以火山图可视化
- 既然可以看感兴趣基因的生存情况,当然就可以批量做完全部基因的生存分析
- 多测试几个数据集生存效应应该是可以找到统计学显著的!
- 我不相信kmplot这个网页工具的结果(生存分析免费做)
- 为什么不用TCGA数据库来看感兴趣基因的生存情况
- 200块的代码我的学徒免费送给你,GSVA和生存分析
- 集思广益-生存分析可以随心所欲根据表达量分组吗
- 生存分析时间点问题
- 寻找生存分析的最佳基因表达分组阈值
- apply家族函数和for循环还是有区别的(批量生存分析出图bug)
- TCGA数据库生存分析的网页工具哪家强
- KM生存曲线经logRNA检验后也可以计算HR值
生存分析是目前肿瘤等疾病研究领域的点睛之笔!