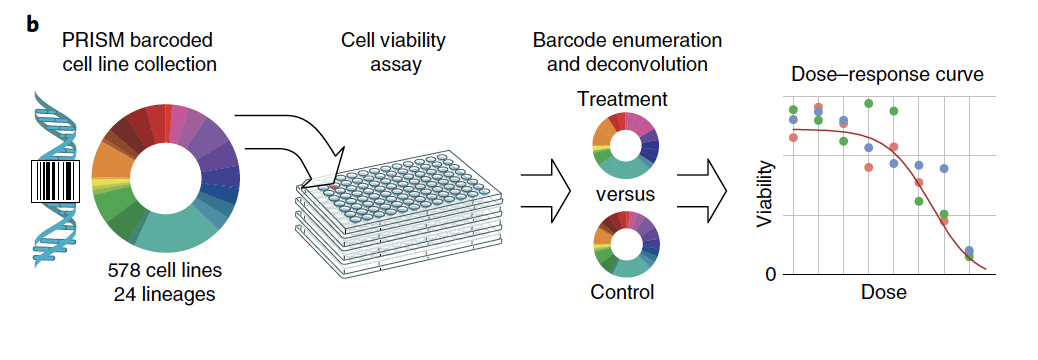
最近在Twitter看到新闻:https://www.biospace.com/article/almost-50-non-oncology-drugs-found-to-kill-cancer-cells/ , 很有意思,提到了MIT的broad研究所和哈佛的研究人员,测试了4518种药物在578个癌症细胞系的效果,然后发现了居然有50种药物虽然很明显并不是抗癌药物,但是杀伤癌细胞的效果出奇的好!
我根据关键词定位到了发表于2020年1月的文章:《Discovering the anticancer potential of non-oncology drugs by systematic viability profiling》,链接是:https://www.nature.com/articles/s43018-019-0018-6 ,4518种药物(都是已获FDA批准或已在临床试验中证明是安全的现有药物和化合物)和578个癌症细胞系,必然得上高通量筛选的实验手段啦,如下所示:
借鉴单细胞转录组的可视化方法
研究人员测试4518种药物在578个癌症细胞系的效果,首先,药物分成了,known oncology (n = 997 compounds) and non-oncology (n = 3,443 compounds)
这个UMAP可视化方法在单细胞转录组数据分析应用非常广泛了。
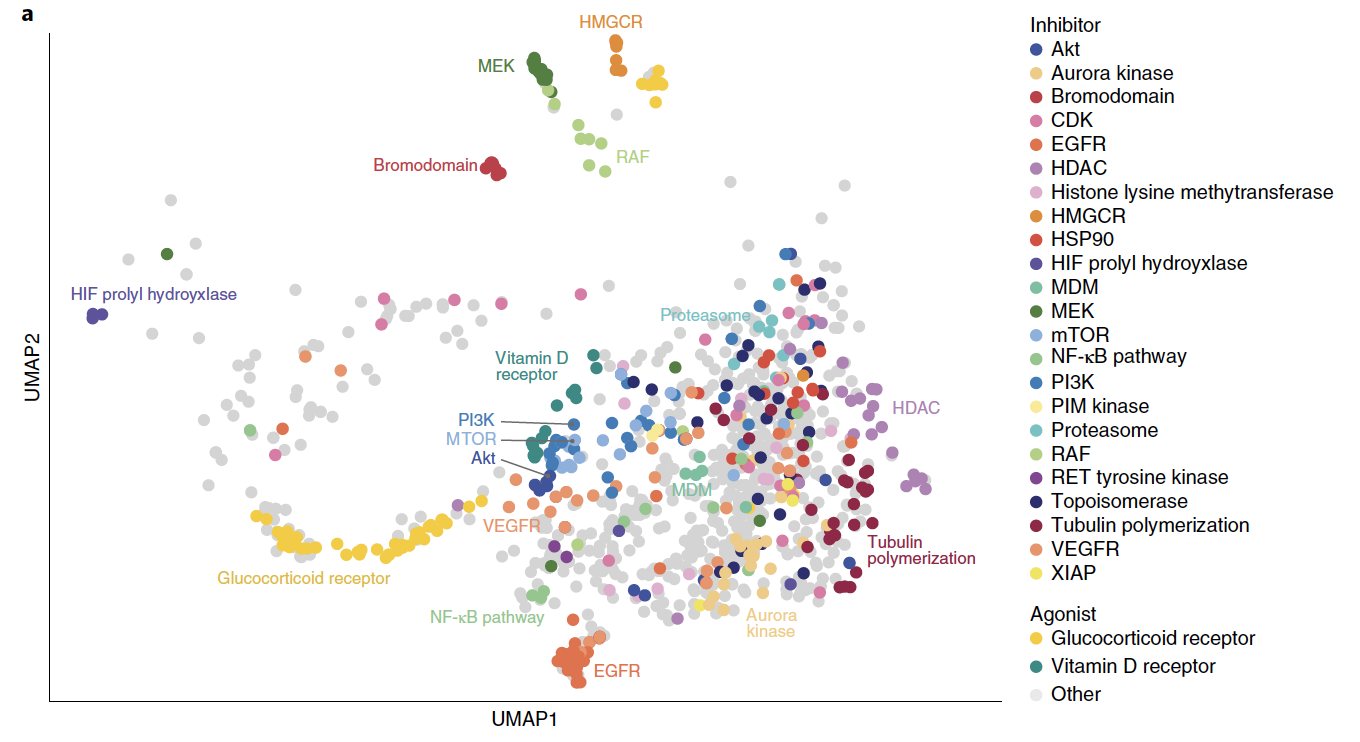
- Two-dimensional UMAP projection of 990 drug killing profiles by cosine similarity.
- Compounds with a shared annotated mechanism of action are labeled by color (n = 640 compounds).
- Compounds with an average Pearson correlation below 0.25 across three independent plates in one PRISM screen are not shown.
这个图里面的细节知识点还挺多的,包括cosine similarity,drug killing profiles 等概念。不过,跟我们这次的新闻报道关系不大,最重要的是用于糖尿病、炎症、酒精中毒甚至用于治疗犬关节炎的药物也可以在实验室条件下杀死癌细胞。这些化合物以意想不到的方式杀死癌细胞。“大多数现有的抗癌药物都是通过阻断蛋白质发挥作用的,但是我们发现这些化合物可以通过其他机制起作用,” 达娜·法伯(Dana-Farber)的人类癌症遗传学研究人员Todd Golub 说。他和他的同事们确定的四种药物中的某些似乎不是通过抑制蛋白质而起作用,而是通过激活蛋白质或稳定蛋白质与蛋白质的相互作用
Corsello说:“基因组特征为我们提供了有关药物作用方式的一些初步假设,然后我们可以将其运回实验室进行研究。我们对这些药物如何杀死癌细胞的理解为我们开发新疗法提供了一个起点。”数据下载链接
- https://depmap.org/repurposing/
- https://figshare.com/articles/PRISM_Repurposing_19Q3_Primary_Screen/9393293
- https://figshare.com/articles/Cell_line_feature_sets_used_in_Corsello_et_al_2020/10277810
- RNA-seq data have been deposited with the Gene Expression Omnibus (accession number GSE133299).