目前大家都在集中精力在单细胞转录组表达矩阵的质控降维聚类分群和注释,其实这个数据分析思路并不仅仅是在单细胞这个当红炸子鸡上面才有。
降维聚类分群是一个非常普遍的信息提炼方式,在TCGA多组学领域都有应用,众所周知,TCGA数据库是目前最综合最全面的癌症病人相关组学数据库,包括:
- DNA Sequencing
- miRNA Sequencing
- Protein Expression array
- mRNA Sequencing
- Total RNA Sequencing
- Array-based Expression
- DNA Methylation
- Copy Number array
但绝大部分朋友们都不会去花时间细看过去的十五年TCGA计划的成果了,往往是奢求一个直接的答案,比如想知道他的基因在某个癌症的某个亚型里面到底有没有临床意义?
实际上,越是急躁的寻求最终结果,越是痛苦,还不如静下心来一个个组学一个个分析细节慢慢攻克。而且这些分析并不仅仅是在TCGA计划及其附属成果里面应用起来了的,大家只需要看文献,就会有新发现,就能学到知识点。比如发表在《cancer cell》杂志的文章《Genomic and Transcriptomic Landscape of Triple-Negative Breast Cancers: Subtypes and Treatment Strategies》,就是一个小TCGA计划。近日有粉丝就咨询我他们文献里面的CNV是如何进行拷贝数全景图聚类分群找差异。
其实文章说的很清楚:Using k-means clustering and consensus clustering, methods similar to mRNA clustering, six clusters based on CNA peaks were identified (Figure 3B; Table S8):
主要是下面是Figure 3B展现的拷贝数全景图聚类分群,粗略看起来确实是杂乱无章,如下所示:
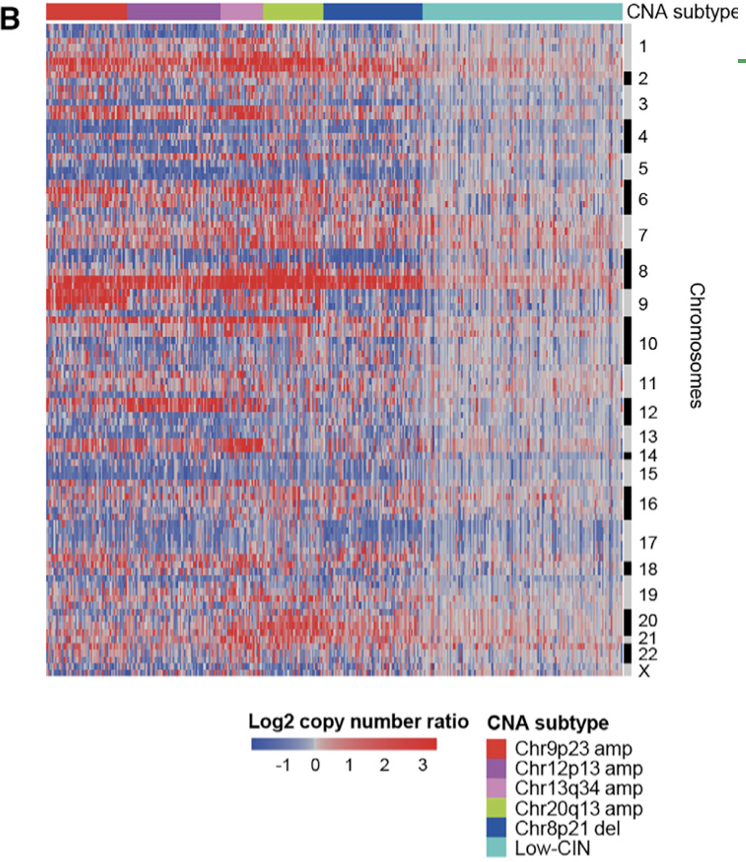
可以看到是6个病人亚群分组,然后就可以总结一下6个病人亚群CNV特性,如下所示: - CNA subtype 1, frequent 9p23 amplification (Chr9p23 amp);
- CNA subtype 2, frequent 12p13 amplification (Chr12p13 amp);
- CNA subtype 3, frequent Chr13q34 amplifications (Chr13q34 amp);
- CNA sub- type 4, frequent Chr20q13 amplification (Chr20q13 amp);
- CNA subtype 5, frequent Chr8p21 loss (Chr8p21 del);
- CNA subtype 6, somatic CNA lacking a CN cluster but with low chromosomal instability (CIN) (low CIN).
多个CNV的比较的表格展示数值差异
在 Table S8, related to Figure 3. Enriched copy number alteration (CNA) events in each CNA subtype 可以看到具体的值:
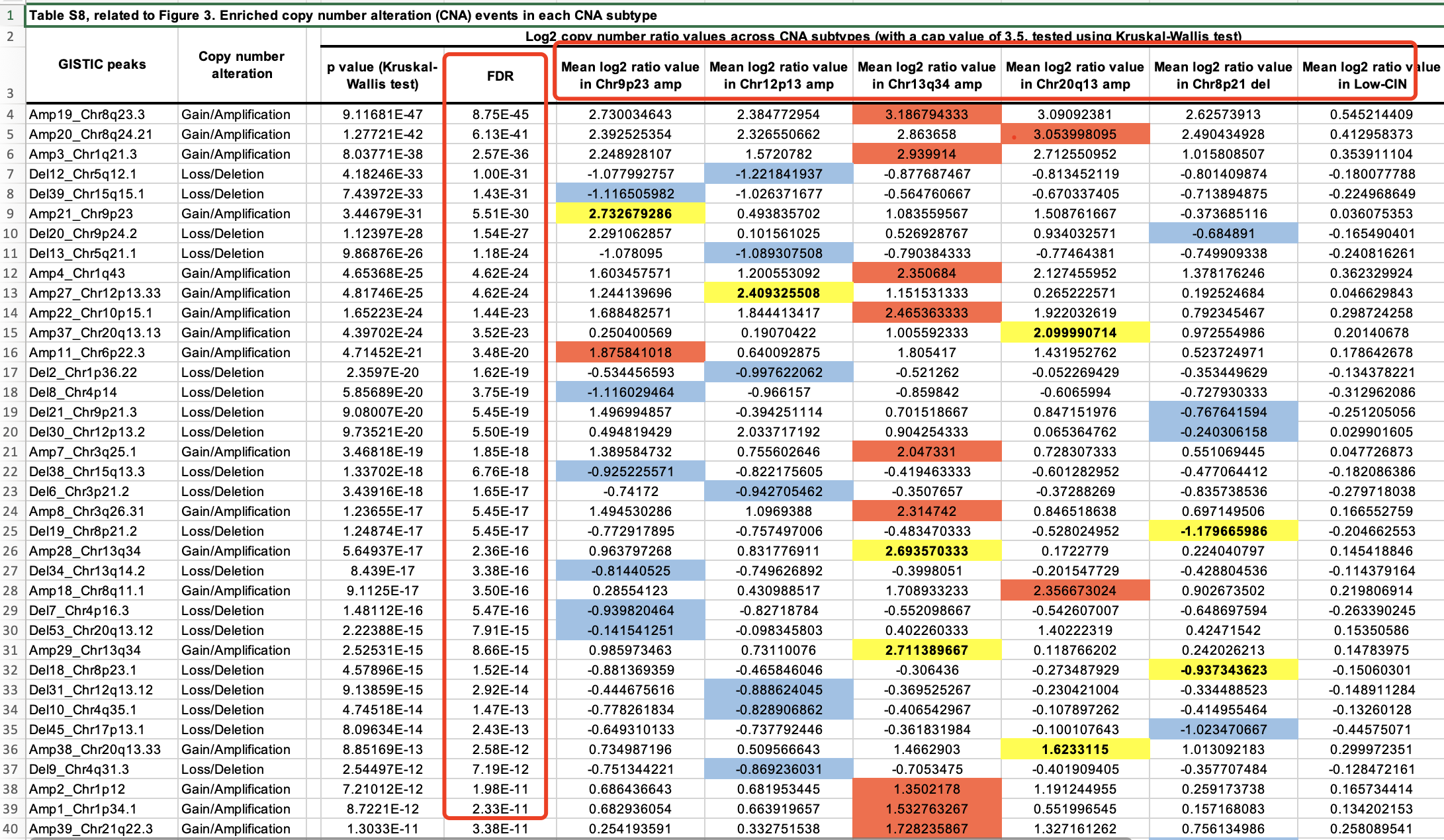
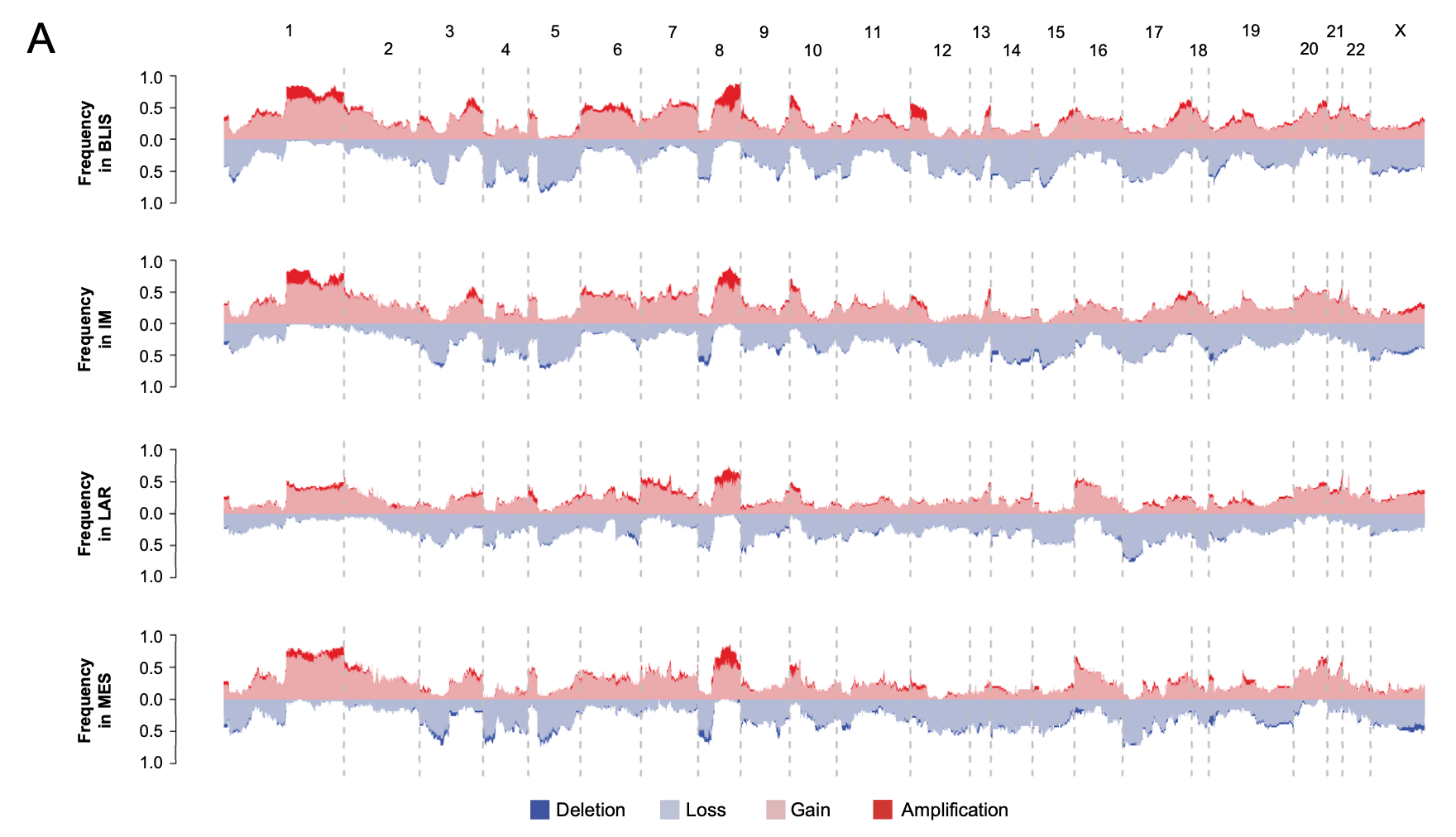
除了上面的 Log2 copy number ratio values across CNA subtypes,还有 CNA frequency in each subtype 的差异。多个CNV的比较的波峰图展示差异
其实有比表格更好的展现方式:
如果是比较两个亚群的CNV全景图
可以使用下面的图:
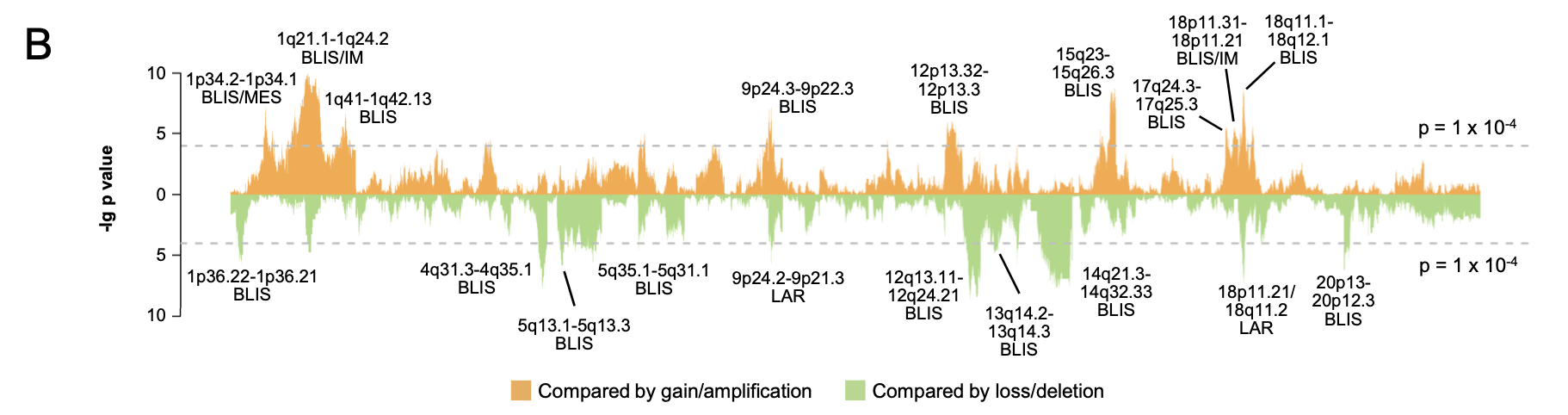
首先需要CNV矩阵
如果大家走肿瘤的拷贝数教程,通常是cnvkit等软件,可以拿到bed格式的CNV信息文件,然后就可以走一下GISTIC2的流程,就可以拿到具体的每个基因在每个样本的拷贝数啦,这个时候的拷贝数通常是 -2,-1,0,1,2 这样的5个可能性而已。
有了每个基因在每个样本的拷贝数矩阵,就可以做热图,聚类分群。分群后具体的每个亚群的bed格式的CNV信息文件,又可以走一下GISTIC2的流程重新拿到各自的CNV全景图,如何读入maftools,就可以做另外的波峰图展示差异。学徒作业
在TCGA计划里面寻找到BRCA数据,拿到其CNV信息文件,然后根据PAM50进行分类后,走我上面提到的数据分析流程并且拿到对应的图表!