我前面吐槽了这个2020年10月发表在cancer cell杂志的文章《Senescence Reprogramming by TIMP1 Deficiency Promotes Prostate Cancer Metastasis》,链接是:https://doi.org/10.1016/j.ccell.2020.10.012 ,详见:转录组数据挑选下游基因能不能不要这么随便啊 ,我提到了它的槽点非常多,我们再吐一个哈!
这个研究做了 10X单细胞转录组,但是正文里面基本上没有描述这个单细胞转录组数据集的质控降维聚类分群和细胞亚群注释的方法和结果,可以说是非常的诡异了。如果大家感兴趣这个文章的数据集,可以去 https://www.ebi.ac.uk/ena/browser/view/PRJEB40397 ,就能看到作者上传的单细胞转录组数据集,是10X技术的,4只小鼠,两个不同的 genetically engineered mouse models (GEMMs) 。
而且,我反复查看了该文章的正文,居然,单细胞数据方面的结果不配出现!说实话,我还是很不能接受这个事实,现在才2021,单细胞大火事情就是2018-2020,今年2021热点虽然说转移到了空间单细胞,但也不至于怎么瞧不起自己的单细胞转录组数据吧!
我去附件看了看,FACS-sorted Epcam+, CD45+ and Epcam-/CD45- prostate cells were used in equal ratio as the input source for the scRNA-seq. 也就是说,我前面的 肿瘤单细胞数据第一次分群通用规则 就这样被吊打了,因为他们在还没有做单细胞的时候,就已经预分选好了。但是为什么为什么配平后这3种细胞的比例仍然是不一致?
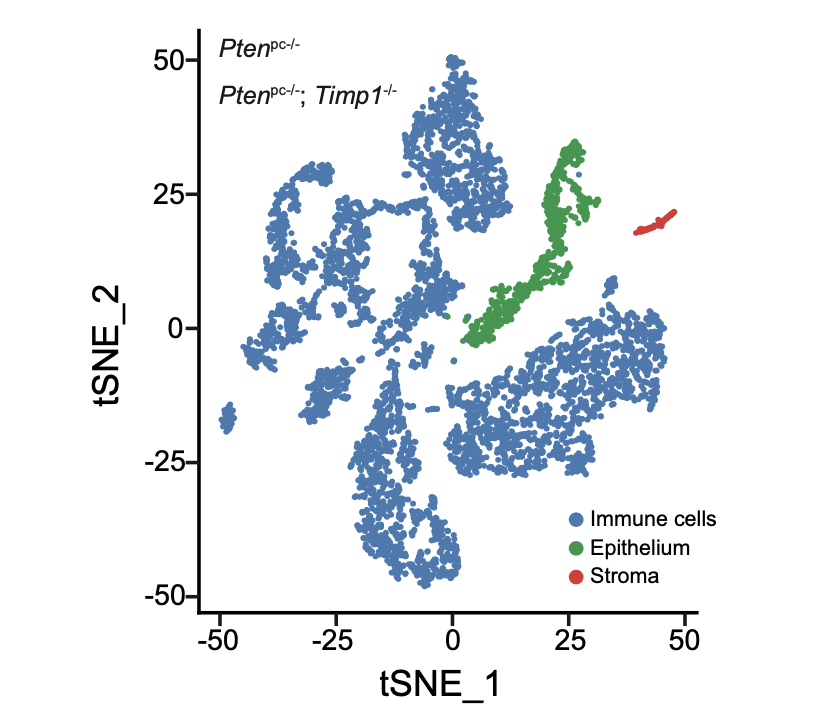
是有什么其它神奇的操作被我漏掉了吗?
更诡异的是tSNE坐标居然取子集了就平移中心点?
大家注意看,前面的初步分群 :
- immune (CD45+,PTPRC),
- epithelial/cancer (EpCAM+,EPCAM),
- stromal (CD10+,MME,fibo or CD31+,PECAM1,endo)
首先,epithelial 细胞亚群比例超级低,就很诡异了。但是呢,更诡异的操作是,作者在取epithelial子集的时候,居然直接就平移了tSNE坐标系!!!
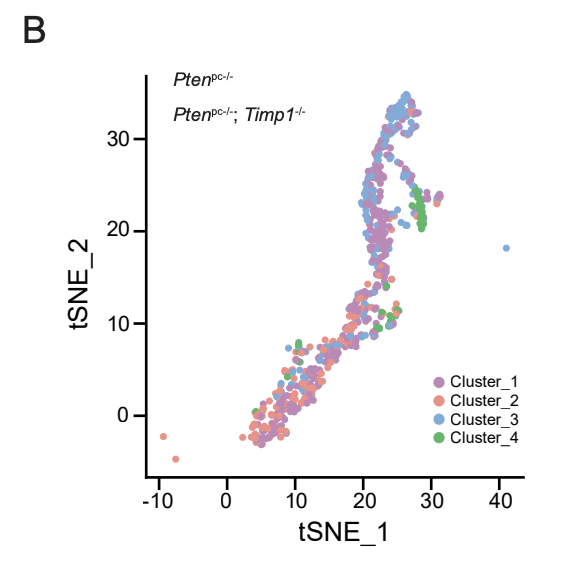
真的是亮瞎了我的24K纯钛合金狗眼!而且关于这个图例,也着实让人费解啊: - (B) t-SNE plots showing the segregation of epithelial tumor single cells into 4 clusters based on hierarchical clustering.
层次聚类区分好的4个亚群,在tSNE坐标系完全混为一群,也是很有意思哦!我还是去原文看了看他关于这个层次聚类的描述:
In the epithelial compartment (Epcam+ cells), we used AddModuleScore function to define senescence score (based on p16, p21, p27, p19 and Pai-1 genes expression). We performed hierarchical clustering, an algorithm that clusters similar data into groups (method = ‘‘complete’’ in phetmap function), and we divided the epithelium population into four main groups, Cluster_3 included cells with high senescence score.
也就是说,并不是全部的表达量矩阵在进行细胞距离计算以及聚类,而且仅仅采用了跟senescence score 相关的基因的表达量矩阵进行进行细胞距离计算以及聚类。这样就能说得通,为什么层次聚类区分好的4个亚群,在tSNE坐标系完全混为一群。毕竟大家使用的基因都不一样。
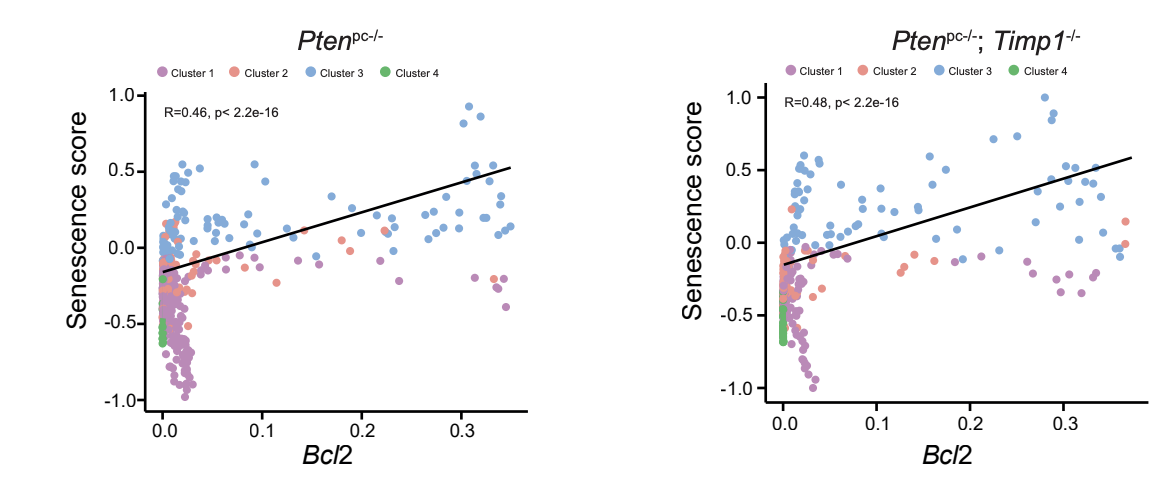
但是你这样搞,仍然不是一个值得鼓励的行为,我要是审稿人,肯定不会让这样的图堂而皇之的发表。最关键的结论是Bcl2基因和senescence score的相关性
图C表现的还不错,可以看到在两个基因型背景的小鼠的单细胞转录组数据里面,都是有 第3群细胞的senescence score最高,而且对应的Bcl2基因表达量也是最高。
(C) Heat map showing expression of Bcl2 in cluster 3 of tumor epithelial cells with high senescence score in Ptenpc-/- and Ptenpc-/- ; Timp1-/- tumors.

图D呢,也是类似的结论,只不过是换了个散点图的展现方式,这个无可厚非。
(D) t-SNE plots show the epithelial cells based on the senescence score, on the expression of Bcl2 and the overlap of cell expressing high senescence score and high Bcl2.
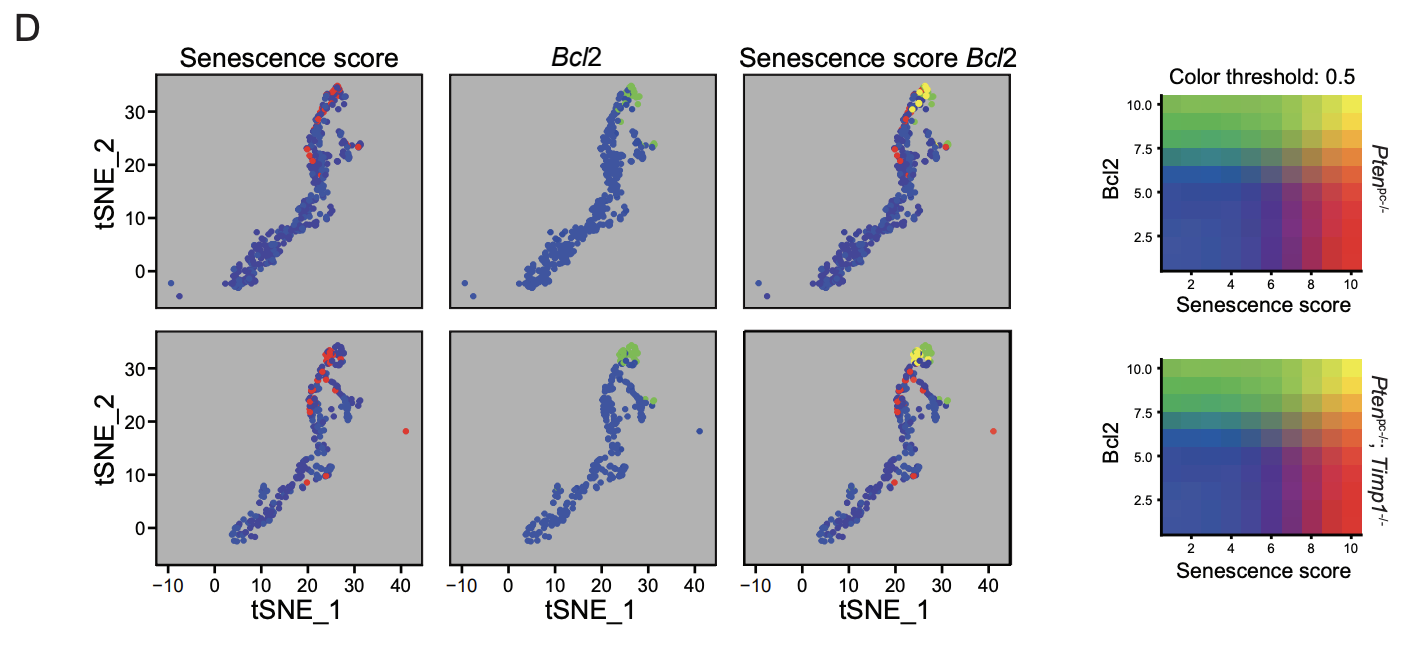
图E基本上说的也是同样的事情,仍然是表现是Bcl2基因和senescence score的相关性。
(E) Scatter plot representing correlation analysis between senescence score and Bcl2 expression in Ptenpc-/- (R= 0.46, p< 2.2e-16) and Ptenpc-/- ; Timp1-/- tumors (R = 0.48, p < 2.2e-16).