传统的分子生物学机制机理研究到最后往往是某个基因在某个癌症等疾病研究具备非常重要的生物学意义,结合目前比较流行的ngs数据资源来说,就可以做大量的临床图表,我这里简单举3个例子抛砖引玉。
### 该基因在肿瘤组织和正常组织的表达量差异箱线图
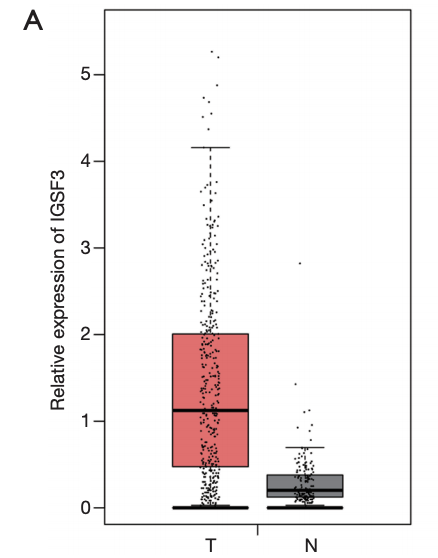
图来源于:doi: 10.21037/atm.2020.02.14
可能的变种:
- 比如可以只挑取严格配对的病人
- 挑选具备某基因突变的病人
该基因的临床预后价值
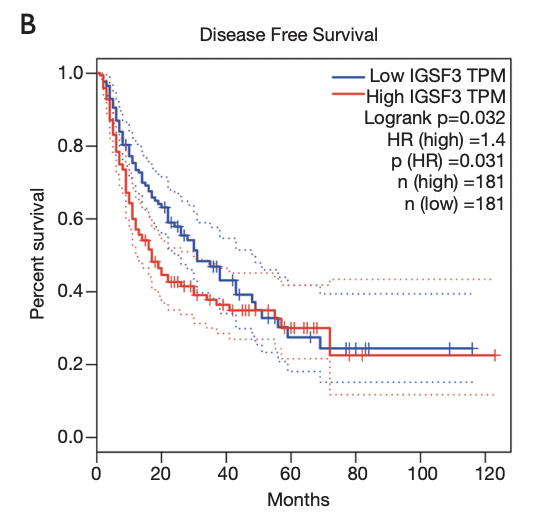
图来源于:doi: 10.21037/atm.2020.02.14
可能的变种:
- 考虑其它结局事件,详见:人人都可以学会生存分析(学徒数据挖掘)
生存分析是目前肿瘤等疾病研究领域的点睛之笔,我在生信技能树多次分享过生存分析的细节;
- 人人都可以学会生存分析(学徒数据挖掘)
- 学徒数据挖掘之谁说生存分析一定要按照表达量中位值或者平均值分组呢?
- 基因表达量高低分组的cox和连续变量cox回归计算的HR值差异太大?
- 学徒作业-两个基因突变联合看生存效应
- TCGA数据库里面你的基因生存分析不显著那就TMA吧
- 对“不同数据来源的生存分析比较”的补充说明
- 批量cox生存分析结果也可以火山图可视化
- 既然可以看感兴趣基因的生存情况,当然就可以批量做完全部基因的生存分析
- 多测试几个数据集生存效应应该是可以找到统计学显著的!
- 我不相信kmplot这个网页工具的结果(生存分析免费做)
- 为什么不用TCGA数据库来看感兴趣基因的生存情况
- 200块的代码我的学徒免费送给你,GSVA和生存分析
- 集思广益-生存分析可以随心所欲根据表达量分组吗
- 生存分析时间点问题
- 寻找生存分析的最佳基因表达分组阈值
- apply家族函数和for循环还是有区别的(批量生存分析出图bug)
- TCGA数据库生存分析的网页工具哪家强
- KM生存曲线经logRNA检验后也可以计算HR值
肿瘤分期等临床条件差异下的表达量差异小提琴图
临床条件太多了,肿瘤分期分级,放化疗与否,耐药与否,复发转移与否,都可以看:
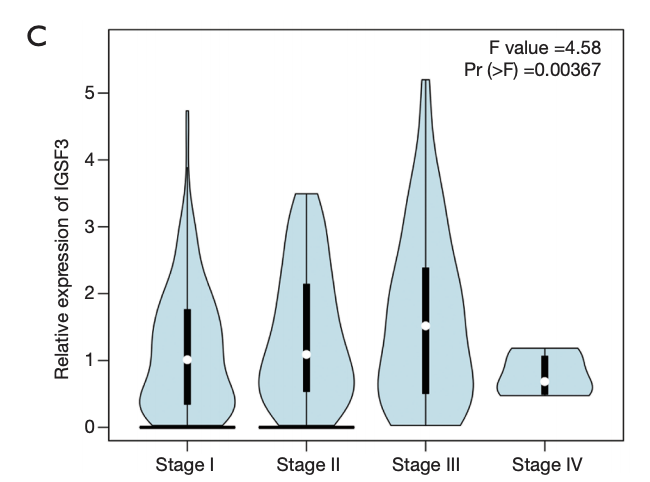
图来源于:doi: 10.21037/atm.2020.02.14
更多图表
我喜欢把TCGA数据库的应用划分为8个领域:
- 1、探索各类肿瘤不同临床特征(性别、年龄、种族、临床分期)的预后(生存曲线)
- 2、探索各类肿瘤与对照的单个分子(mRNA,lncRNA,miRNA,甲基化,蛋白)水平的差异情况(箱线图)
- 3、探索各类肿瘤与对照的全局(mRNA,lncRNA,miRNA,甲基化,蛋白)水平的差异情况(差异分析流程)
- 4、探索各类肿瘤中两个分子(mRNA,lncRNA,miRNA,甲基化,蛋白)水平相关性(散点图)
- 5、探索各类肿瘤中多个分子(mRNA,lncRNA,miRNA,甲基化,蛋白)水平总结(热图)
- 6、探索各类肿瘤中单个分子(mRNA,lncRNA,miRNA,甲基化,蛋白)与所有其它分子相关性并且排序
- 7、探索各类肿瘤中单个基因突变或者单个分子(mRNA,lncRNA,miRNA,甲基化,蛋白)水平的预后(生存曲线)
- 8、探索各类肿瘤不同临床特征(性别、年龄、种族、临床分期)分组后的单个分子(mRNA,lncRNA,miRNA,甲基化,蛋白)特性的分布
这些应用都是有对应的网页工具或者R代码可以实现!