最近在整理新冠疫情相关的单细胞文章,尤其是那些提供了数据集的而且还有配套GitHub代码的,超级棒的学习资料。
比如北京大学张泽民教授的实验室发表在 Nat Med. 2020 Jun的文章《Single-cell landscape of bronchoalveolar immune cells in patients with COVID-19》,链接是:https://www.nature.com/articles/s41591-020-0901-9 ,全部的代码在https://github.com/zhangzlab/covid_balf.数据集在 GSE145926。
有条件的可以考虑下载全部的单细胞转录组数据,涉及到12个人,6 severe and 3 moderate COVID-19 patients and 3 healthy control,都是取 bronchoalveolar lavage fluid (BALF) 做单细胞,同时做了 (scRNA-seq)/TCR-seq,都是10X仪器。
初始的分群如下所示:
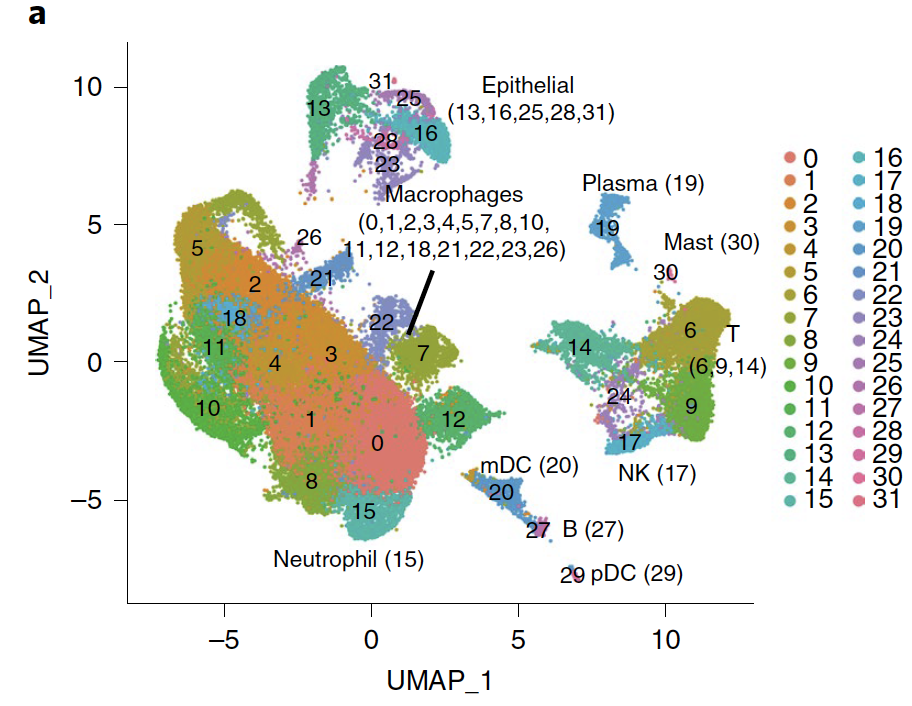
我看了看文章里面对这些大群的描述,主要是项目的这些标记基因:
- 巨噬细胞(CD68)和中性粒细胞(FCGR3B)
- 髓样树突状细胞(mDCs)(CD1C,CLEC9A)
- 血小板样树突状细胞(pDCs)(LILRA4)
- 自然杀伤(NK)细胞(KLRD1)、T细胞(CD3D)
- B细胞(MS4A1)、浆细胞(IGHG4)
- 上皮细胞(TPPP3,KRT18)
使用下面的代码就可以检测:
library(ggplot2)
genes_to_check = c( 'CD68','FCGR3B','CD1C','CLEC9A','LILRA4','KLRD1','CD3D',
'MS4A1','IGHG4','TPPP3','KRT18'
)
library(stringr)
#genes_to_check=unique(str_to_title(genes_to_check))
genes_to_check
p <- DotPlot(sce.all, features = genes_to_check,
assay='RNA' ) + coord_flip()
p
可以看到,这个时候的T细胞主要是 (NK)细胞(KLRD1)和T细胞(CD3D),并不是我们前两天提到的:T细胞可以简单分成4类,有意思的是,这篇文章的T细胞也是区分为4个大类:
- CD4+ T cells (upper),
- CD8+ T cells (lower),
- innate T cells (left)
- proliferating T cells (right).
如下所示:
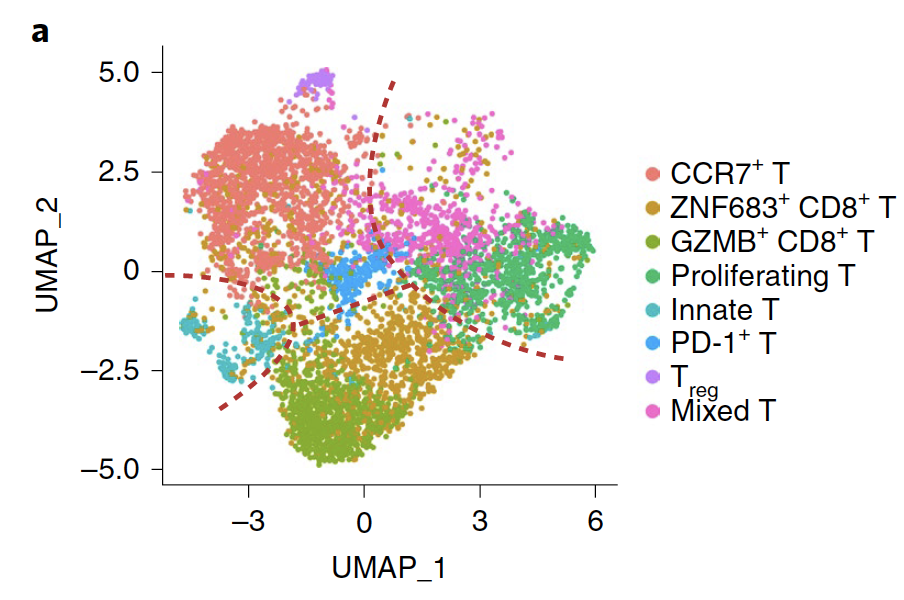
这就非常尴尬了,基本上每个文章都有自己的T细胞分类系统啊!
另外,值得一提的是研究者们提供了一个网页工具来探索他们的单细胞数据集:https://cells.ucsc.edu/?ds=covid19-balf
全部的R代码也是公开的:https://github.com/zhangzlab/covid_balf
R script for the covid balf data analysis. (Liao M. et al, Single-cell landscape of bronchoalveolar immune cells in patients with COVID-19, Nature Medicine, 2020. https://www.nature.com/articles/s41591-020-0901-9)