在我们的单细胞交流群看到了这样的提问《singleR注释小鼠免疫细胞用哪个参考集效果比较好》:

让我想起来了被免疫细胞里面的淋巴系和髓系的细胞细分群及注释支配的恐惧,虽然在前面的例子:人人都能学会的单细胞聚类分群注释 ,我们演示了第一层次的分群,但是第二层次的分群就很麻烦。绝大部分文献基本上没有参考价值,比如发表于2020年8月的文章是:《Single-cell RNA sequencing uncovers heterogenous transcriptional signatures in macrophages during efferocytosis》,链接在:https://www.nature.com/articles/s41598-020-70353-y ,做了6只 C57BL/6J品系的小鼠的单细胞,每个小鼠平均1400个单细胞,合起来还不到一万个单细胞,让我们来来看看作者是如何对细胞亚群进行注释:
To unbiasedly identify resident peritoneal cells present in the dataset, SingleR (v1.0.5) was employed. Briefly, SingleR infers the origin of each individual cell by referencing transcriptomic datasets of pure cell types. We utilized the ImmGen database, which contains normalized expression values for immune cells from 830 murine microarrays to ID our peritoneal cell types. These classifications were confirmed with canonical immune cell markers.
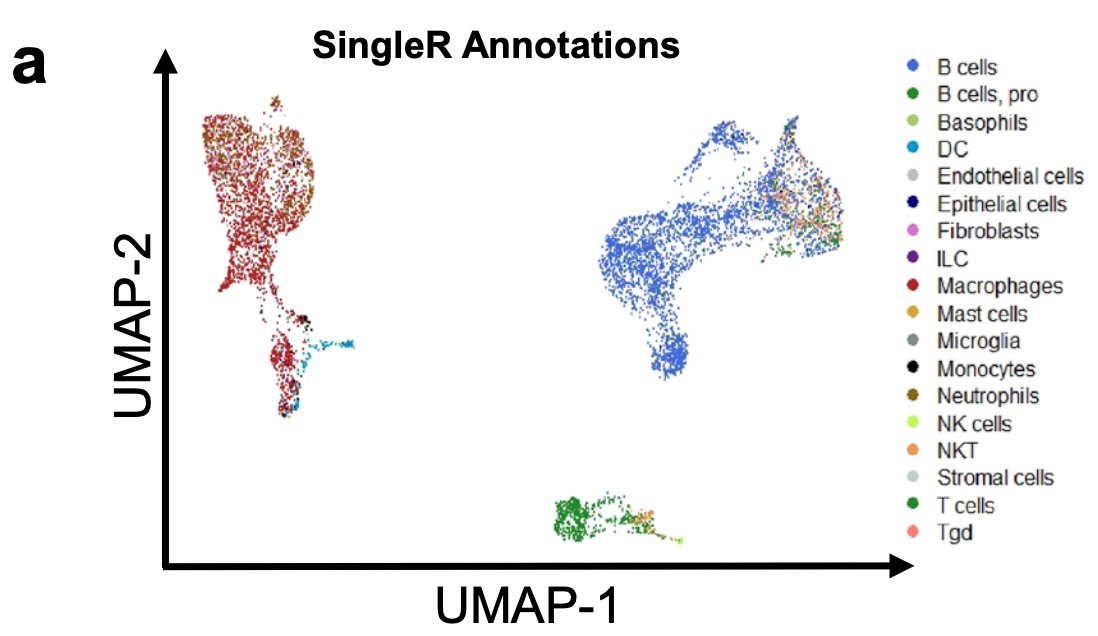
可以看到,非常的奇怪,超级多的亚群,其实并没有太大的意义。
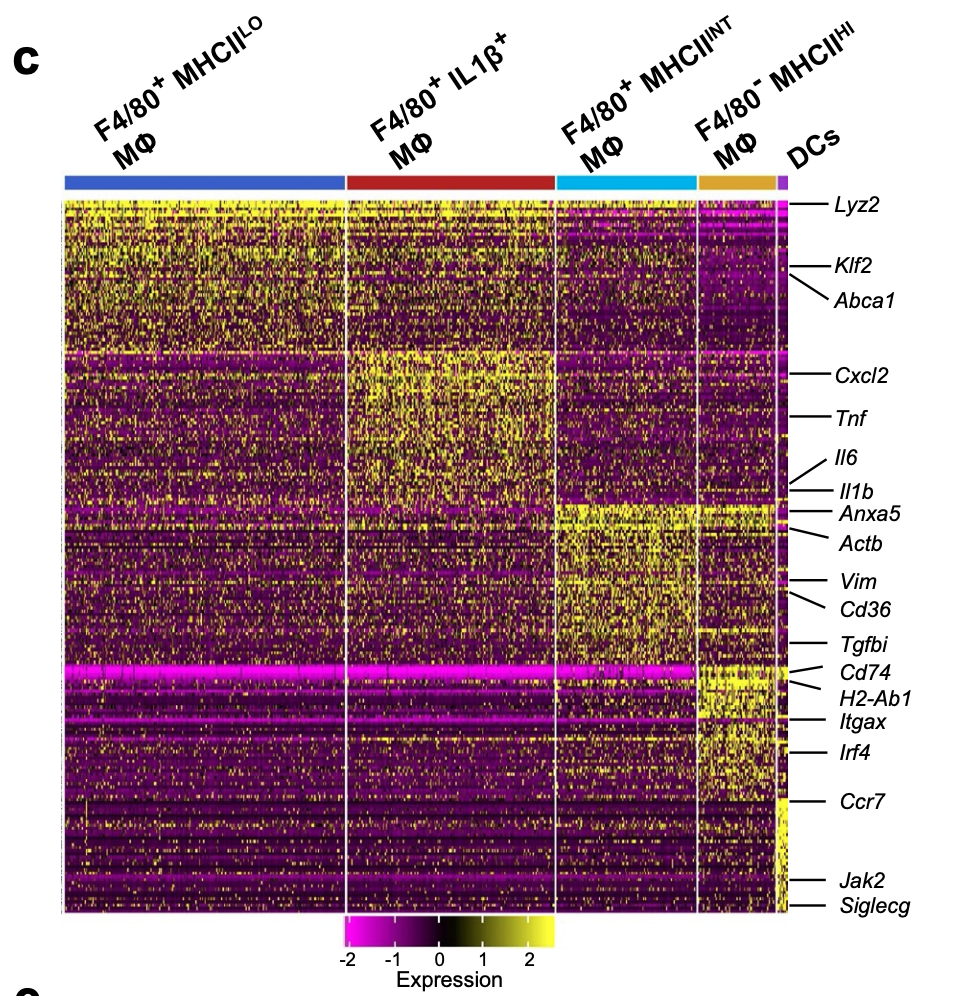
然后研究者们着重关注了 macrophage and dendritic cell ,它们都是髓系来源的免疫细胞:
High-Resolution UMAP dimensional reduction of macrophage and dendritic cell (DC) partitioned into 5 distinct clusters.
数据在:https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE156234
GSM4736404 Peritoneum - WT Control
GSM4736405 Peritoneum - WT 2 hr Effero
GSM4736406 Peritoneum - WT 6 hr Effero
GSM4736407 Peritoneum - MerKD Control
GSM4736408 Peritoneum - MerKD 2 hr Effero
GSM4736409 Peritoneum - MerKD 6 hr Effero
我下载了这个数据集,然后走了一下单细胞流程,非常的奇怪哦,跟文章基本上对不上!
我这里主要是展示如何使用SingleR
前面的构建单细胞对象,以及安装各个R包就不再赘述啦!
library(SingleR)
sce
sce_for_SingleR <- GetAssayData(sce, slot="data")
clusters=sce@meta.data$seurat_clusters
mouseImmu <- ImmGenData()
pred.mouseImmu <- SingleR(test = sce_for_SingleR, ref = mouseImmu, labels = mouseImmu$label.main,
method = "cluster", clusters = clusters,
assay.type.test = "logcounts", assay.type.ref = "logcounts")
mouseRNA <- MouseRNAseqData()
pred.mouseRNA <- SingleR(test = sce_for_SingleR, ref = mouseRNA, labels = mouseRNA$label.fine ,
method = "cluster", clusters = clusters,
assay.type.test = "logcounts", assay.type.ref = "logcounts")
cellType=data.frame(ClusterID=levels(sce@meta.data$seurat_clusters),
mouseImmu=pred.mouseImmu$labels,
mouseRNA=pred.mouseRNA$labels )
这里我使用了两个小鼠数据库哦,是ImmGenData() 和MouseRNAseqData() 两个函数,独立注释如下:
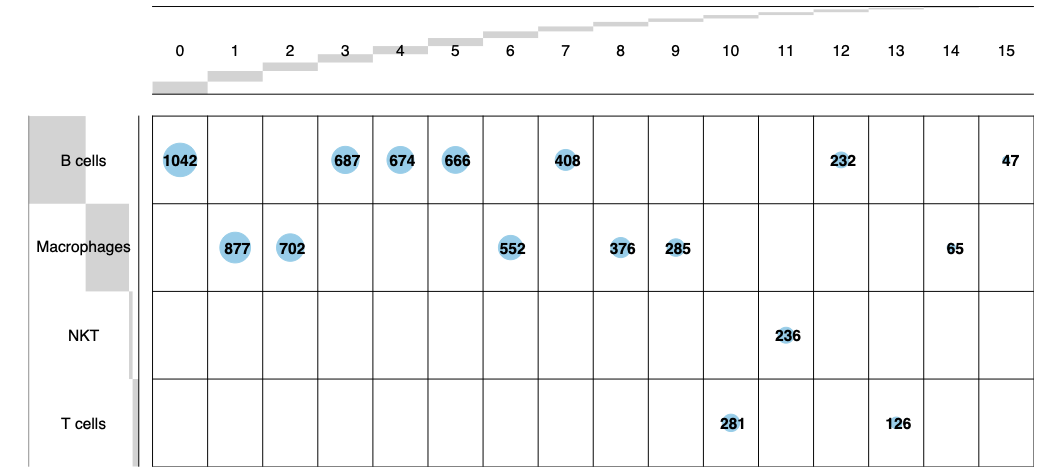
> cellType
ClusterID mouseImmu mouseRNA
1 0 B cells B cells
2 1 Macrophages Macrophages
3 2 Macrophages Macrophages
4 3 B cells B cells
5 4 B cells B cells
6 5 B cells B cells
7 6 Macrophages Macrophages
8 7 B cells B cells
9 8 Macrophages Macrophages activated
10 9 Macrophages Macrophages
11 10 T cells T cells
12 11 NKT T cells
13 12 B cells B cells
14 13 T cells T cells
15 14 Macrophages Macrophages
16 15 B cells B cells
也就是说,都是注释到了 Macrophages ,并没有 dendritic cell (DC) 的事情。我自己绘图如下:
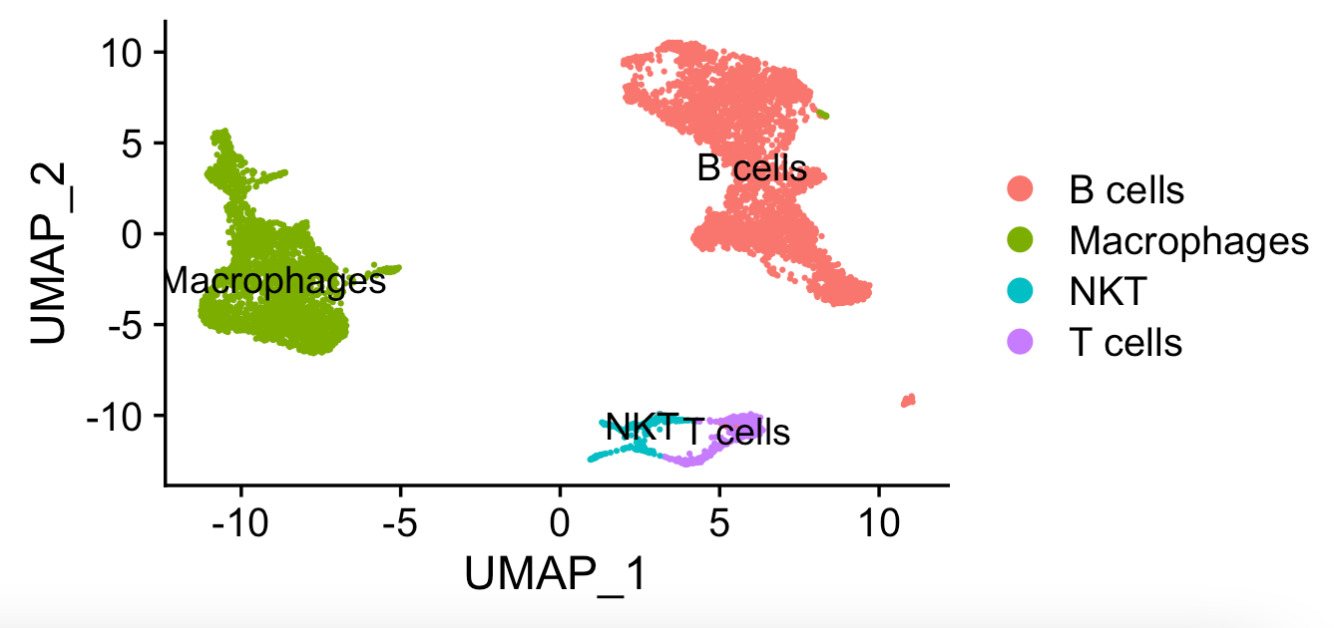
可以看到 NKT 细胞亚群和 T cells确实能区分开来,而且呢B细胞跟它们也很不一样,当然了,它们都是可以继续细分的,不过我们这里先不看这么细。
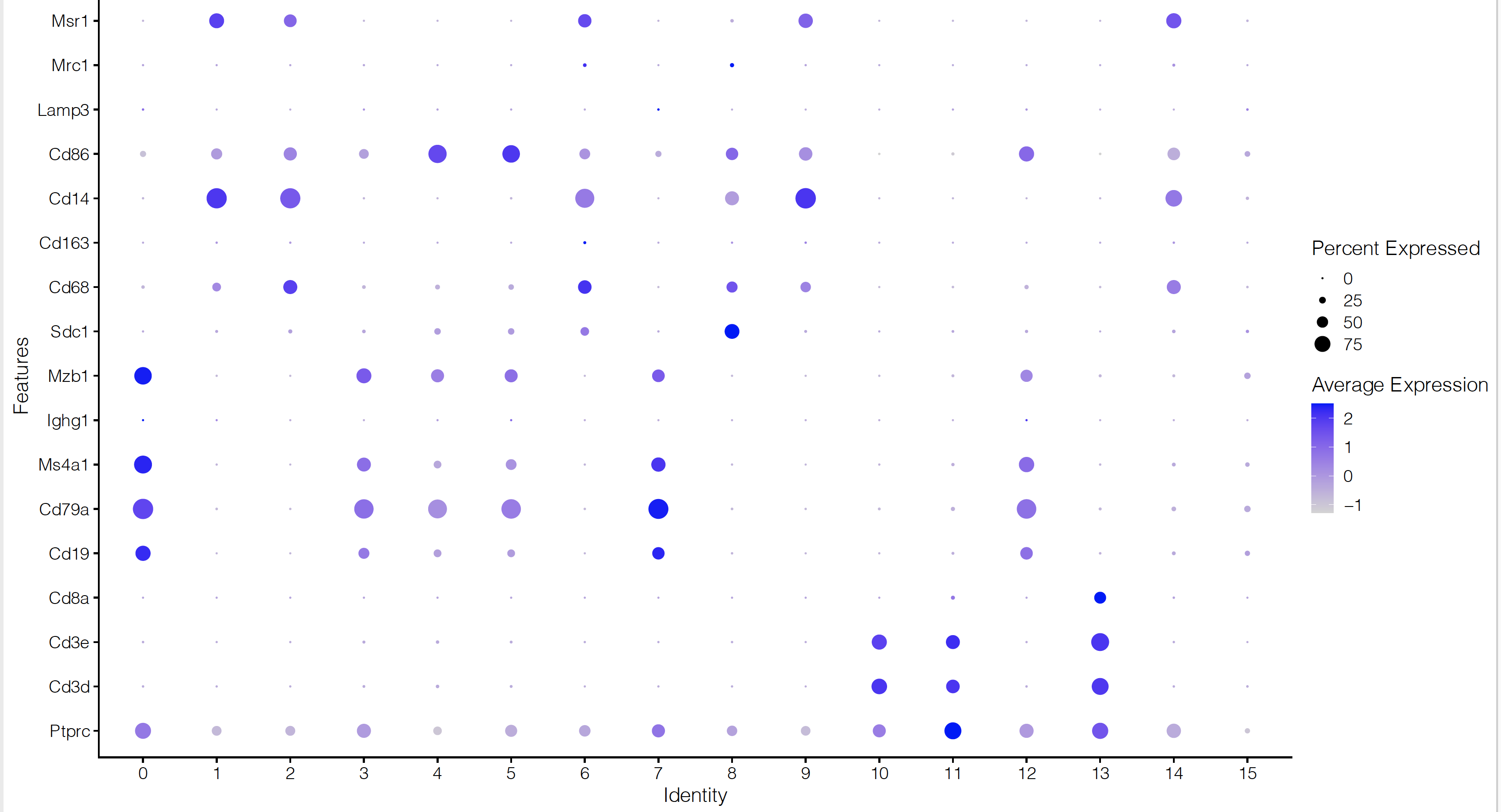
不同亚群对应到生物学注释,如下:
很明显我没有加入 NKT和T细胞的标记基因,最近看到了发表在 Nat Med. 2020 Jun的文章《Single-cell landscape of bronchoalveolar immune cells in patients with COVID-19》,链接是:https://www.nature.com/articles/s41591-020-0901-9 ,全部的代码在https://github.com/zhangzlab/covid_balf.数据集在 GSE145926. 有如下所示的标记:
# 巨噬细胞(CD68)和中性粒细胞(FCGR3B)
# 髓样树突状细胞(mDCs)(CD1C,CLEC9A)
# 血小板样树突状细胞(pDCs)(LILRA4)
# 自然杀伤(NK)细胞(KLRD1)、T细胞(CD3D)
# B细胞(MS4A1)、浆细胞(IGHG4)和上皮细胞(TPPP3,KRT18)
但是人和鼠又很不一样,真麻烦!
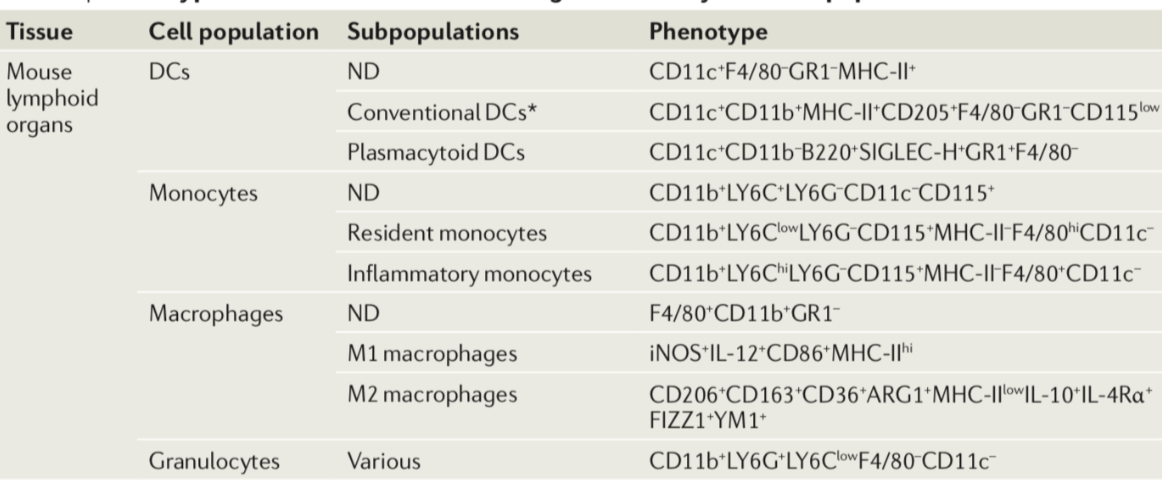
另外,出一个思考题:myeloid里面似乎是包含了 monocyte,macrophage,Dendritic cells,Neutrophils ,Granulocytes, 那么这5个该如何区分呢?在人和鼠里面是不是不一样?能找到一个含有 这5个细胞的数据集,然后使用 Mark基因,给它绘制一个气泡图吗?