我在CNS图表复现09—上皮细胞可以区分为恶性与否提到了一个很有意思的现象,就是把上皮细胞分群后,可以看到有一些亚群是具有病人特异性,但是也有不少亚群是跨越了病人存在的。
而我展现这一现象使用的可视化方法就是balloonplot函数啦,它来自于gplots这个包!
首先创造一个数据
n=10*sample(100,9);n
cancer=rep(paste0('c',1:9),n)
table(cancer)
p=c(rep(paste0('p',1:5),n[1:5]),
sample(paste0('p',1:5),sum(n[6:9]),replace = T))
table(p)
table(cancer,p)
可以看到是如下所示:
> table(cancer,p)
p
cancer p1 p2 p3 p4 p5
c1 890 0 0 0 0
c2 0 420 0 0 0
c3 0 0 330 0 0
c4 0 0 0 800 0
c5 0 0 0 0 730
c6 155 170 180 184 181
c7 197 194 189 194 176
c8 70 64 54 56 56
c9 116 106 102 129 107
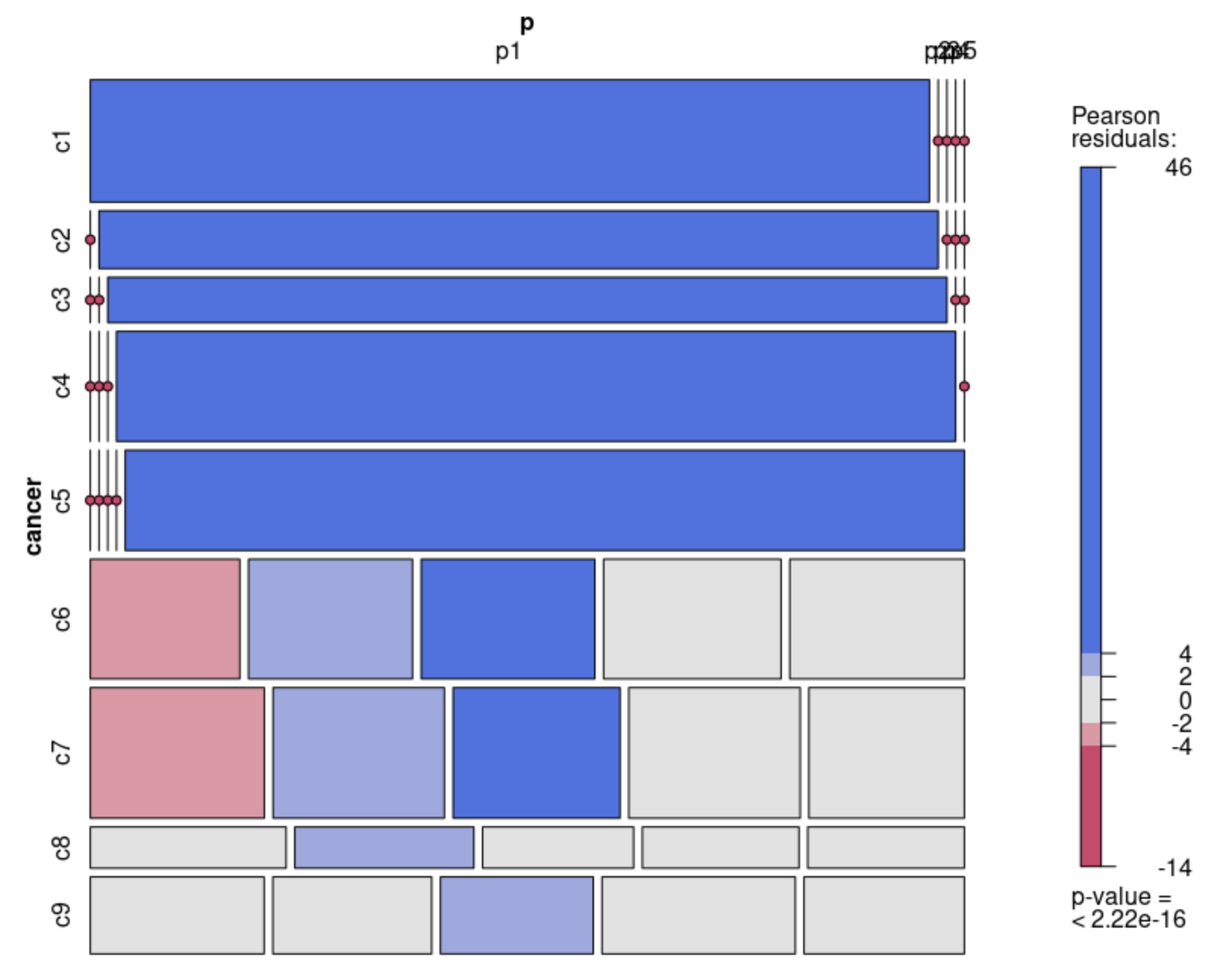
前面的c1到c5,都是病人特异性的,每个病人一个独立单细胞亚群,通常是恶性细胞。但是后面的 c6和c9都是涵盖了5个病人的正常细胞。
在肺癌领域,恶性和细胞正常细胞各自的标记基因如下所示:
- epi or tumor (EPCAM, KRT19, PROM1, ALDH1A1, CD24).
- alveolar type I cell (AT1; AGER+)
- alveolar type II cell (AT2; SFTPA1)
- secretory club cell (Club; SCGB1A1+)
- basal airway epithelial cells (Basal; KRT17+)
- ciliated airway epithelial cells (Ciliated; TPPP3+)
balloonplot可视化
前面我们提到过可视化方法就是balloonplot函数啦,它来自于gplots这个包!
library(gplots)
balloonplot(table(cancer,p))
效果如下所示:

马赛克图可视化
代码如下:
library(vcd)
dat=table(cancer,p)
mosaic(dat,shade=T,legend=T)
还有一个更优秀的可视化方法
就是桑基图,明天再介绍它!