我在CNS图表复现09—上皮细胞可以区分为恶性与否提到了一个很有意思的现象,就是把上皮细胞分群后,可以看到有一些亚群是具有病人特异性,但是也有不少亚群是跨越了病人存在的。
当时我展现这一现象使用的可视化方法就是balloonplot函数啦,它来自于gplots这个包!而且我还创造了一个模拟数据,如下所示;
n=10*sample(100,9);n
cancer=rep(paste0('c',1:9),n)
table(cancer)
p=c(rep(paste0('p',1:5),n[1:5]),
sample(paste0('p',1:5),sum(n[6:9]),replace = T))
table(p)
table(cancer,p)
library(gplots)
balloonplot(table(cancer,p))
library(vcd)
dat=table(cancer,p)
mosaic(dat,shade=T,legend=T)
dat
使用这个模拟数据,比较了balloonplot和马赛克图的可视化结果,见:展示细胞比例变化之balloonplot和马赛克图
但是有不少粉丝留言说,两个图都不好看。其实是他们完全搞错了我教程的重心,绘图根本就是小儿科的事情,最重要的是上面的数据模拟代码,这个才是技术含量!
如果你仅仅是需要好看的图,大把的成熟的R包,供你使用,比如 ggalluvial 可以绘制桑基图,代码如下:
df=data.frame( cancer,p)
head(df)
# 两列数据,互相之间有对应关系,可以绘制桑基图
library(ggplot2)
library(ggalluvial)
options(stringsAsFactors = F)
gg <- ggplot(df,
aes(axis1 = cancer, axis2 = p ));gg
gg <- gg +
geom_alluvium(aes(fill = as.factor(cancer)),
width = 2/5, discern = FALSE);gg
gg <- gg +geom_stratum(width = 2/5, discern = FALSE);gg
#添加文本
gg <- gg +geom_text(stat = "stratum", discern = FALSE,
aes(label = after_stat(stratum))) ;gg
gg <- gg +
theme(legend.position = "none",#去除刻度线和背景颜色
panel.background = element_blank(),
axis.ticks = element_blank(),
axis.text.y = element_blank(),
axis.text.x = element_text(size =15,colour = "black"),#坐标轴名
axis.title = element_blank()) +
scale_x_discrete(position = "top");gg #坐标轴位置
关键是两列数据,互相之间有对应关系,可以绘制桑基图,效果如下所示:
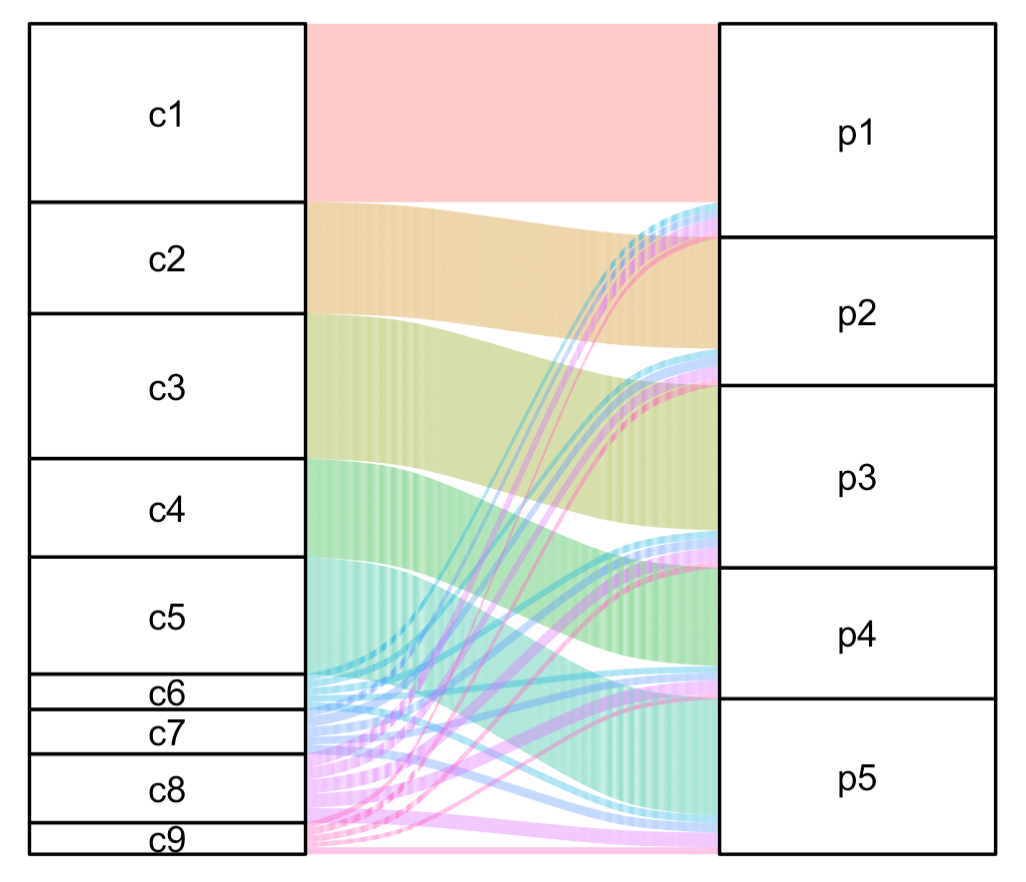
当然了,我这个是模拟数据,前面的c1到c5,都是病人特异性的,每个病人一个独立单细胞亚群,通常是恶性细胞。
但是后面的 c6和c9都是涵盖了5个病人的正常细胞,从上面的桑基图可以很清晰的看出来这一点,是不是非常直观!!!
桑基图上镜率持续走高
比如2021的纯生物信息学文献:《Conserved pan-cancer microenvironment subtypes predict response to immunotherapy》,就有一个看起来超级复杂的桑基图,图例:(C) Sankey plot showing antigenicity and TMB (left) per TME subtype linked to mutation group (right) across TCGA patients at the pan-cancer level.
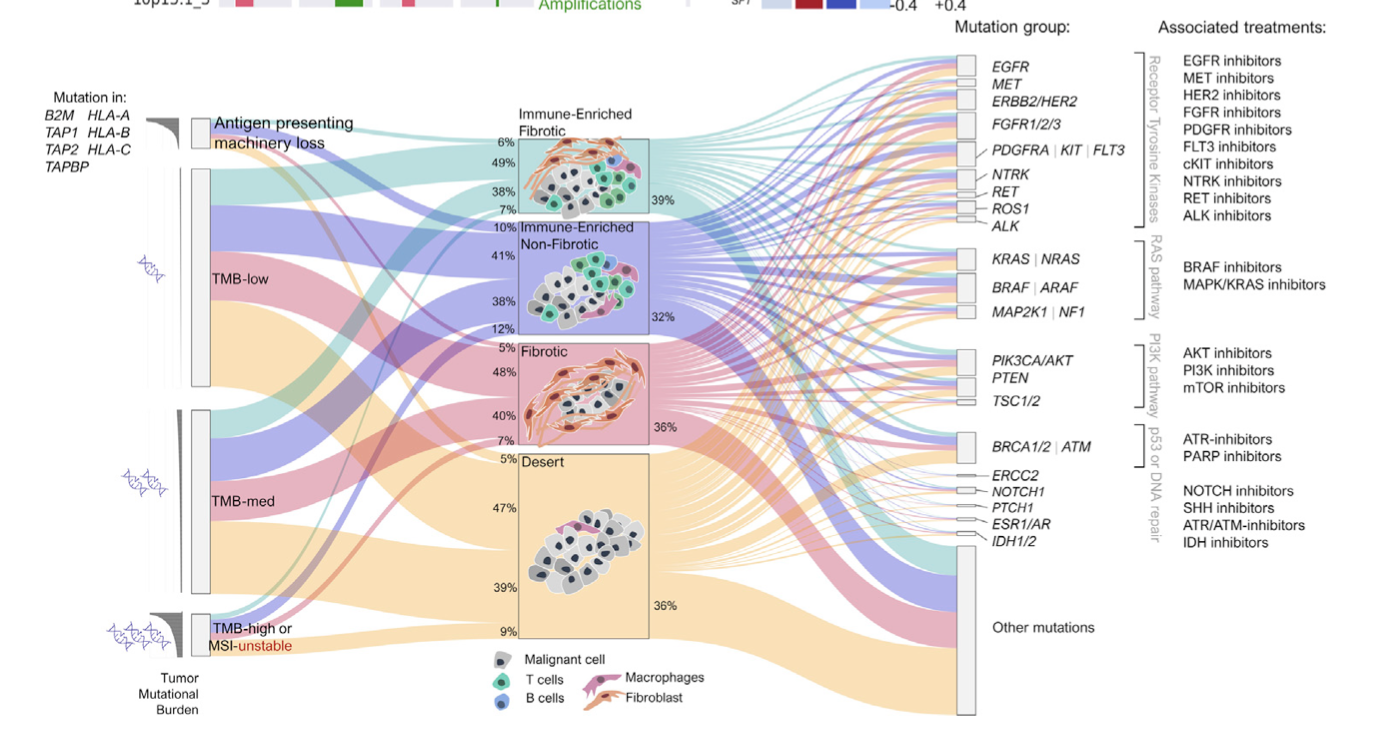
这个时候 ggalluvial 可以绘制桑基图,但是它只能是绘制一个框架,这里面的大量的插图都是AI制作和拼接。
现在,桑基图你知道它需要的输入数据了吗?你会画了吗?我每个教程给出来的代码都是复制粘贴即可运行,如果你都不去动手, 是永远不可能学会哦!