在 NCBI的ftp里面关于人的一些基因信息, 在 : ftp://ftp.ncbi.nlm.nih.gov//gene 下载即可!
其中 gene2pubmed.gz 这个是NCBI的entrez ID号对应着该基因发表过的文章的ID号
- 链接是: ftp://ftp.ncbi.nlm.nih.gov//gene/DATA/gene2pubmed.gz
这个 gene2pubmed.gz 约50M,网络后的话,几分钟就下载OK了。
整理数据文件
简单的使用bing搜索一下关键词:word clound in r ,就可以找到解决方案,第一个链接就是:http://www.sthda.com/english/wiki/text-mining-and-word-cloud-fundamentals-in-r-5-simple-steps-you-should-know,代码分成5个步骤。
- Step 1: Create a text file
- Step 2 : Install and load the required packages
- Step 3 : Text mining
- Step 4 : Build a term-document matrix
- Step 5 : Generate the Word cloud
一般来说,会R基础的朋友们很容易看懂,如果你还不会R语言,建议看:
把R的知识点路线图搞定,如下:
- 了解常量和变量概念
- 加减乘除等运算(计算器)
- 多种数据类型(数值,字符,逻辑,因子)
- 多种数据结构(向量,矩阵,数组,数据框,列表)
- 文件读取和写出
- 简单统计可视化
- 无限量函数学习
核心代码就是wordcloud函数,但是这个wordcloud函数要求的输入数据就需要认真做出来。
library(data.table)
a=fread('gene2pubmed.gz',data.table = F)
head(a)
# https://www.uniprot.org/taxonomy/9606
a=a[a$`#tax_id`==9606,]
library("tm")
library("SnowballC")
library("wordcloud")
library("RColorBrewer")
tb=as.data.frame(table(a$GeneID))
head(tb)
tb=tb[order(tb$Freq,decreasing = T),]
tb=head(tb,100)
colnames(tb)[1]='gene_id'
set.seed(1234)
如下所示:
> head(tb)
gene_id Freq
5608 7157 10251
5580 7124 5966
1550 1956 5929
5827 7422 4771
2778 3569 4746
292 348 4544
可以看到,关联到pubmed文献基因最多的是 7157和 7124,当然了,这个时候它仍然是 Entrez Gene
- 2005 https://pubmed.ncbi.nlm.nih.gov/15608257/
- 2007 https://www.ncbi.nlm.nih.gov/pmc/articles/PMC1761442/
- 2011 https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3013746/
但是已经可以绘图了
第一次绘图
绘图是R语言里面最简单但是如果要深入又最折磨人的,比如这个词云,就一个函数即可:
wordcloud(words = tb$gene_id, freq = tb$Freq, min.freq = 1,
max.words=200, random.order=FALSE, rot.per=0.35,
colors=brewer.pal(8, "Dark2"))
可以看到, 基因根据其热门程度,以不同的大小显示出来了:
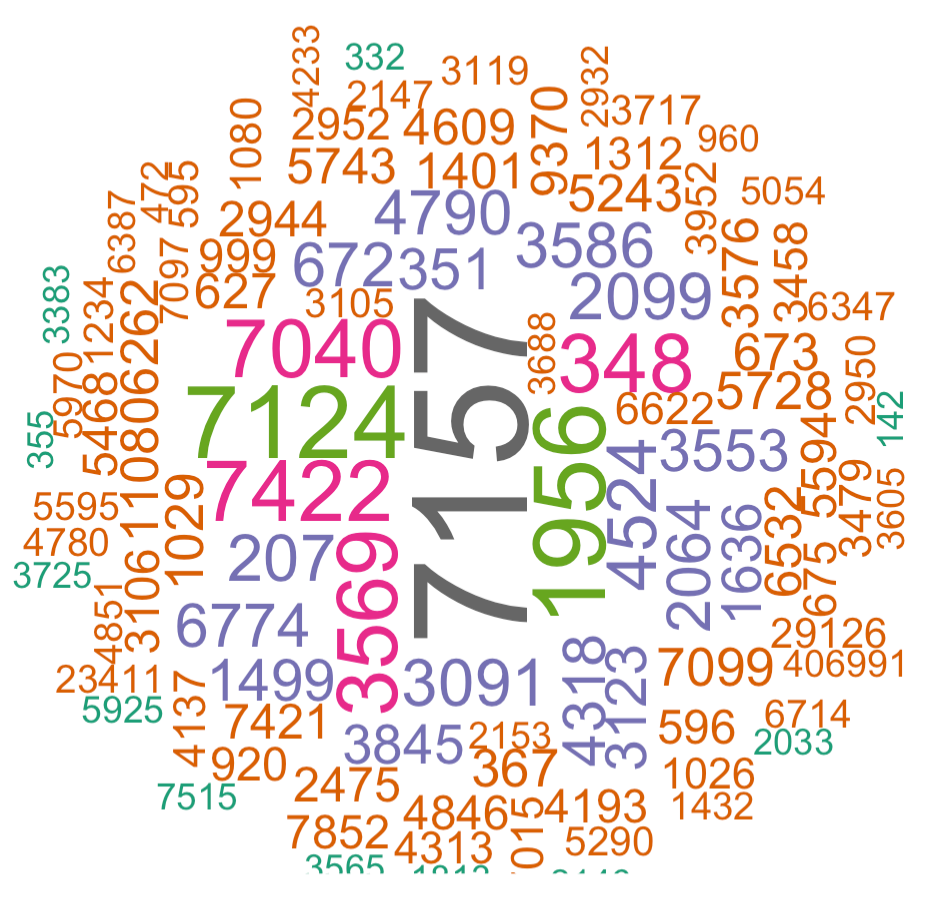
第二次绘图
因为前面的基因是 Entrez Gene,绝大部分人是没办法一目了然的去认识它,比如这个时候虽然知道研究最多的是7157和 7124 这两个基因
所以,我们需要理所当然的做一个ID转换,如下所示的代码:
library(org.Hs.eg.db)
ids=toTable(org.Hs.egSYMBOL)
head(ids)
tbs=merge(ids,tb,by='gene_id')
wordcloud(words = tbs$symbol, freq = tbs$Freq, min.freq = 1,
max.words=200, random.order=FALSE, rot.per=0.35,
colors=brewer.pal(8, "Dark2"))
重新出图如下:

是不是现在就顺眼很多了呢,不知道这个词云里面有没有你正在研究的基因呢?
更多拓展作为学徒作业
这个 gene2pubmed.gz 约50M文件里面的信息太丰富了,有1333万行信息,仅仅是人类就有159万行的文献,涉及到3万9千的基因数量,绝大部分基因都是如过眼云烟,很少人去研究它。
我们的TP53能拔得头彩也是不容易,但它也有自己的发展规律,希望大家可以更细致去探索 ftp://ftp.ncbi.nlm.nih.gov//gene 里面的文件。
比如这样的top 100的基因词云,其实可以做出来最近30年的变化规律,只需要你去找到文献的时间年份信息,进行拆分,每个年份独立统计绘图即可。