年前立下的flag,说要把明码标价专栏扩充到100个项目:
- ATAC-seq项目的标准分析仅收费1600
- 单细胞转录组的质控降维聚类分群和生物学注释仅收费800
- 普通转录组上游分析仅收费800
- 公共数据库的WGCNA分析仅需800
- 公共数据库的生存分析进需800
- 明码标价之转录组常规测序服务(仅需799每个样品)
- 明码标价之转录组下游分析仅需800元
转眼间2021都过去了快三分之一,我们的明码标价专栏建设的进度条却纹丝不动,感觉略微有点尴尬,得加油啦!根据大家在我们《生信技能树》,《生信菜鸟团》以及《单细胞天地》公众号后台关键词查询频率来看,甲基化差异分析是一个高频需求,就先把它明码标价哈!最高频的3个甲基化技术
这3个甲基化技术就是 甲基化测序的 WGBS和RRBS,还有 芯片:
- 全基因组DNA甲基化测序(Whole Genome Bisulfite Sequencing,WGBS)是 DNA 甲基化研究的金标准,它通过 Bisulfite 处理和全基因组 DNA 测序结合的方式,对整个基因组上的甲基化情况进行分析,具有单碱基分辨率,可精确评估单个 C 碱基的甲基化水平,构建全基因组精细甲基化图谱。数据量非常大。
- 简化甲基化测序 (Reduced representation bisulfite sequencing, RRBS)是一种准确、高效、经济的DNA甲基化研究方法,通过酶切 (Msp I) 富集启动子及CpG岛区域,并进行Bisulfite测序,同时实现DNA甲基化状态检测的高分辨率和测序数据的高利用率。作为一种高性价比的甲基化研究方法,简化甲基化测序在大规模临床样本的研究中具有广泛的应用前景。
- Illumina的Infinium BeadChip芯片,包括HumanMethyation450(450K)和MethylationEPIC(850K)。Infinium芯片存在染料偏差、不同探针化学和位置效应的问题,已知这些问题会影响结果,必须在数据处理过程中进行校正。Infinium 450K探针交叉反应和模糊比对到人类基因组中的多个位置影响了485,000个探测器中的约140,000个探针(29%),将可用探针的数量减少到约345,000个。这个问题在新发布850K仍然存在,其包括> 90%的450K探针。
有文章比较这3个技术:Empirical comparison of reduced representation bisulfite sequencing and Infinium BeadChip reproducibility and coverage of DNA methylation in humans,感兴趣的都是可以自由阅读,提高自己哈。甲基化信号的主要分析也是 差异甲基化区域(DMRs)与 DMR 相关差异基因。基因不同位置的甲基化的生物学意义不同
不同区域的甲基化位点信号值的生物学意义完全不一样,很多研究都喜欢混淆它们,我觉得有必要重点指出来大家的错误。
一般流行的分类是:(Up2kb, 5′-UTR, CodingExon, Intron, 3′-UTR, Down2kb and Intergenic regions) , 比如就是doi: 10.1093/nar/gkr053文章就是这样分类!甲基化芯片数据处理我是有视频课程的
首先需要阅读我在生信技能树的甲基化系列教程,目录如下:
- 01-甲基化的一些基础知识.pdf
- 02-甲基化芯片的一般分析流程.pdf
- 03-甲基化芯片数据下载的多种技巧.pdf
- 04-甲基化芯片数据下载如何读入到R里面.pdf
- 05-甲基化芯片数据的一些质控指标.pdf
- 06-甲基化信号值矩阵差异分析哪家强.pdf
- 07-甲基化芯片信号值矩阵差异分析的标准代码.pdf
- 08-TCGA数据库的各个癌症甲基化芯片数据重新分析.pdf
- 09-TCGA数据库的癌症甲基化芯片数据重分析.pdf
- 10-TCGA数据辅助甲基化区域的功能研究.pdf
- 11-按基因在染色体上的顺序画差异甲基化热图.pdf
- 850K甲基化芯片数据的分析.pdf
- 使用DSS包多种方式检验差异甲基化信号区域.pdf
然后就可以看我在B站免费分享的视频课程《甲基化芯片(450K或者850K)数据处理 》 - 教学视频免费在:https://www.bilibili.com/video/BV177411U7oj
- 课程配套思维导图:https://mubu.com/doc/1cwlFgcXMg
明码标价
无论你的甲基化数据是自己实验室产出的,还是公共数据库,只要是WGBS和RRBS或者甲基化芯片的,我们就可以进行简单(2个分组)的差异分析:
- WGBS收费1600
- RRBS收费1000
- 甲基化芯片(450K和850K)都是800元
2个分组样品量不能超过20个,主要的结果在:07-甲基化芯片信号值矩阵差异分析的标准代码.pdf
选择champ流程,进行一系列差异分析结果可视化,火山图,MA图, 热图等等!
有需要的小伙伴,可以在公众号后台留言咨询,或者在此推文下面留言均可,我们会有专门的生信工程师对接大家的课题哦!一个RRBS差异分析例子
于 2020 Aug 发表在cancer research 杂志的文章,标题是:《STAT3 and GR Cooperate to Drive Gene Expression and Growth of Basal-Like Triple-Negative Breast Cancer》, DOI: 10.1158/0008-5472.CAN-20-1379
如下所示: - To identify regulatory regions specific to basal-like breast cancer, reduced representation bisulfite sequencing (RRBS) was performed on 28 breast cancer cell lines (18 basal-like and 10 luminal) in order to measure DNA methylation across the genome.
- Of the 479,746 CG positions in the genome with at least 10x coverage in each cell line, 3,748 CGs were significantly differentially methylated (Linear Regression Benjamini-Hochberg adjusted p< 0.05) between basal-like and luminal cell lines
也就是说这个RRBS技术其实就测到了不到48万个位点,数据量来说有点类似于甲基化450K的芯片了,然后basal-like和 luminal组的差异分析,就可以区分上下调条讨论: - 在 luminal 组里面是unmethylated的1,300 CGs位点, 去和ENCODE计划的149个TF的ChIP-seq 的bed文件进行坐标交集,最显著的是 ER, FOXA1, and GATA3 这样的转录因子。
- 在 basal-like 组里面是unmethylated的1,793 CGs位点, , 去和ENCODE计划的149个TF的ChIP-seq 的bed文件进行坐标交集,最显著的是 JUN, STAT3, and GR 这样的转录因子。
然后还结合公共数据库的 ATAC-seq data from 59 primary breast tumors (15 basal-like and 44 luminal)
我们的标准差异分析只能说提供这样的文献的一张图,就是最简单的热图,如下所示:
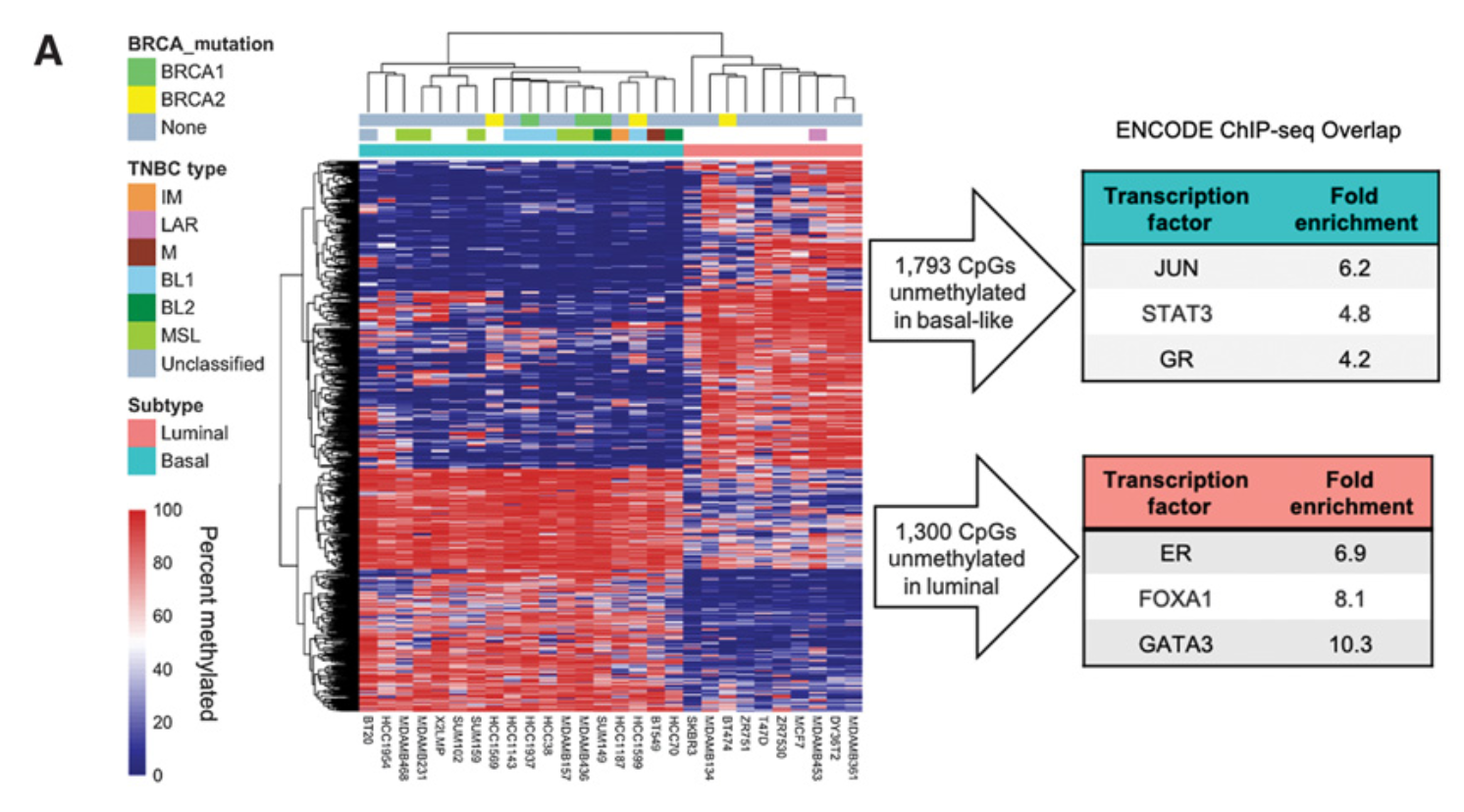
如果是这个示例文献的ChIP-seq 和ATAC-seq 结合,费用可能是需要几何倍数增加,所以建议自己学会生物信息学,自己摸索,或者说雇佣一个生信工程师来按照你的想法随心所欲的探索各式各样的数据分析可能性!