有人提问,他自己做单细胞的gsva, 细胞通讯,转录因子,拟时序, inferCNV这些分析,发现特别的消耗计算资源,因为项目很多,每个细胞亚群都是过万的细胞。希望可以这些单细胞亚群进行抽样,使得其细胞数量一致。
我们以 seurat 官方教程为例:
rm(list = ls())
library(Seurat)
# devtools::install_github('satijalab/seurat-data')
library(SeuratData)
library(ggplot2)
library(patchwork)
library(dplyr)
load(file = 'basic.sce.pbmc.Rdata')
DimPlot(pbmc, reduction = 'umap',
label = TRUE, pt.size = 0.5) + NoLegend()
sce=pbmc
如果你不知道 basic.sce.pbmc.Rdata 这个文件如何得到的,麻烦自己去跑一下 可视化单细胞亚群的标记基因的5个方法,自己 save(pbmc,file = ‘basic.sce.pbmc.Rdata’) ,我们后面的教程都是依赖于这个 文件哦!
比如subset函数就有downsample参数
使用起来超级方便,subset(sce, downsample = 15) 即可,全部的 代码如下:
features= c('IL7R', 'CCR7','CD14', 'LYZ', 'IL7R', 'S100A4',"MS4A1", "CD8A",'FOXP3',
'FCGR3A', 'MS4A7', 'GNLY', 'NKG7',
'FCER1A', 'CST3','PPBP')
DoHeatmap(subset(sce ),
features = features,
size = 3
)
table(Idents(sce))
DoHeatmap(subset(sce, downsample = 15),
features = features, size = 3)
抽样前后很容易看出来:
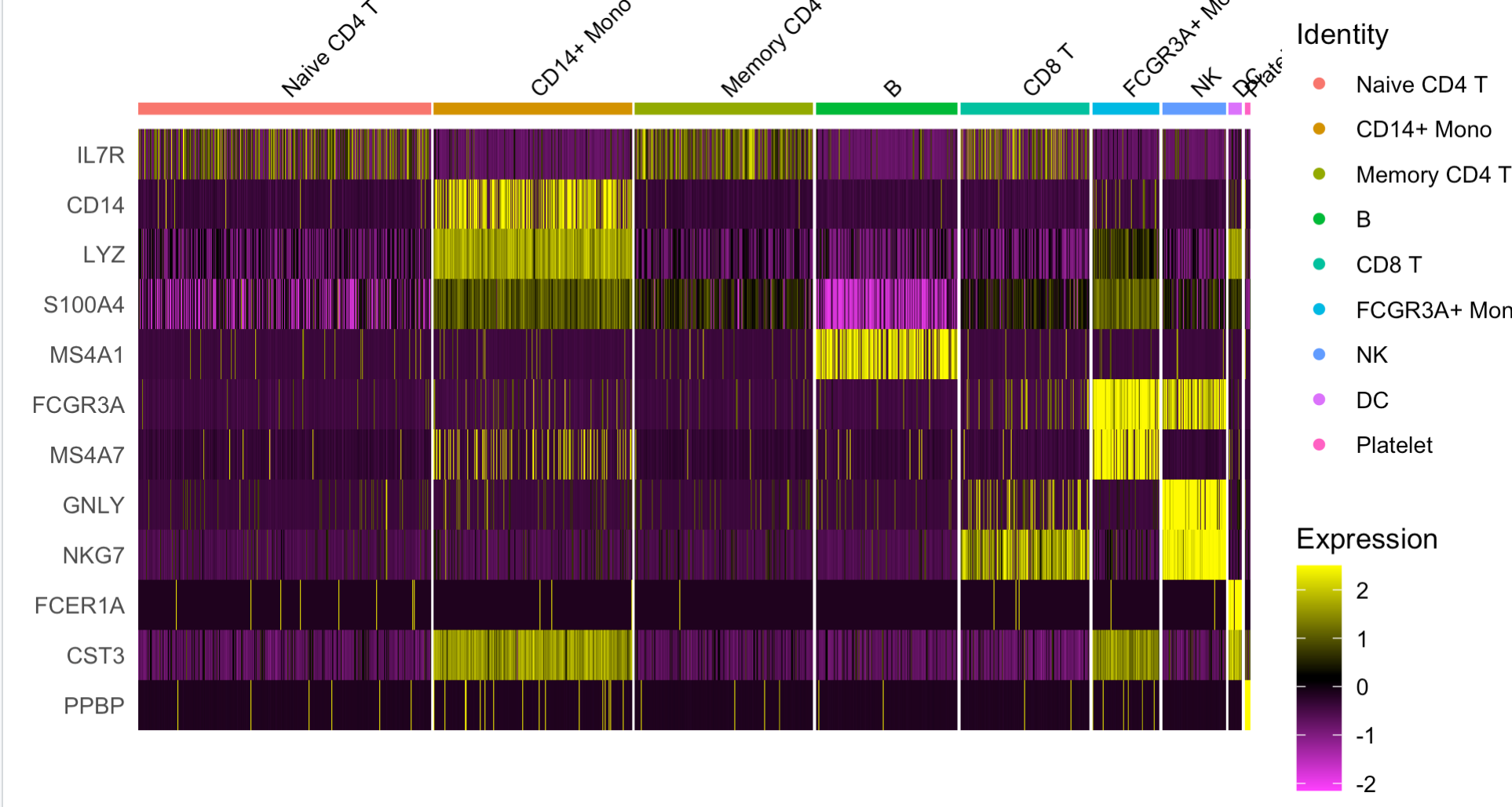
可以看到:
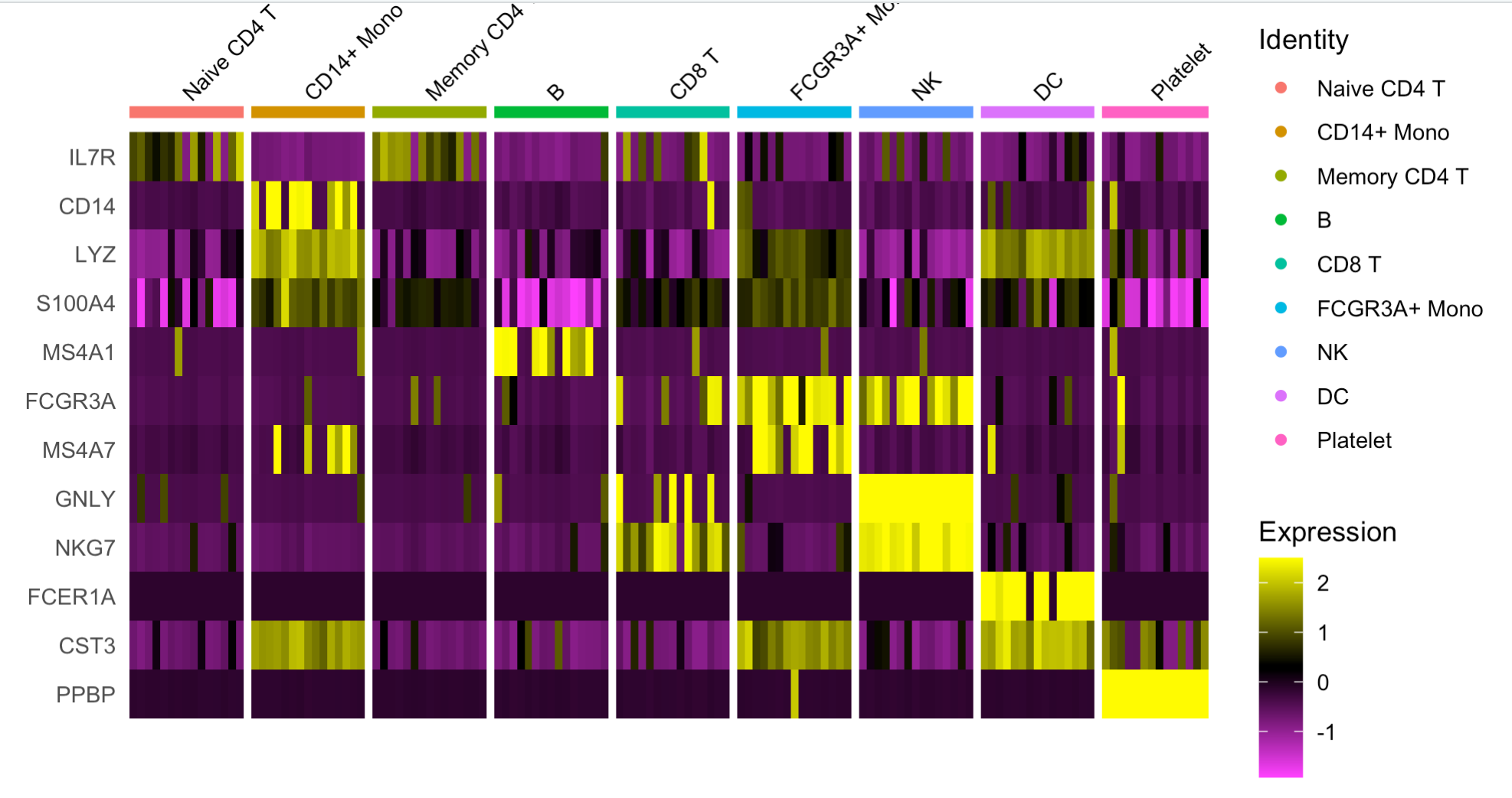
自己写函数进行抽样
# 每个细胞亚群抽10
allCells=names(Idents(sce))
allType = levels(Idents(sce))
choose_Cells = unlist(lapply(allType, function(x){
cgCells = allCells[Idents(sce)== x ]
cg=sample(cgCells,10)
cg
}))
cg_sce = sce[, allCells %in% choose_Cells]
cg_sce
table(Idents(cg_sce))
可以看到抽样很成功:
> as.data.frame(table(Idents(cg_sce)))
Var1 Freq
1 Naive CD4 T 10
2 CD14+ Mono 10
3 Memory CD4 T 10
4 B 10
5 CD8 T 10
6 FCGR3A+ Mono 10
7 NK 10
8 DC 10
9 Platelet 10