咱们《生信技能树》公众号一直缺乏宏基因组数据分析,还有蛋白质组学,代谢组的笔记,是时候补充起来了。主要是因为我们着重于基础知识体系建设,各个ngs组学或者其它生物信息学方向无非是背景知识的积累,万变不离其宗,统计可视化部分是相通的。
我们以一篇2019年的CELL杂志的文章为例,标题:《Stress-Induced Metabolic Disorder in Peripheral CD4+ T Cells Leads to Anxiety-like Behavior》,链接是:https://pubmed.ncbi.nlm.nih.gov/31675497/
首先看组间差异和组内差异
我们一直强调,看组间差异和组内差异主要是3张图,代谢组学和转录组都是一样,这个文章呢,主要是展现了 Partial Least-squares discrimination analysis (PLS-DA) :
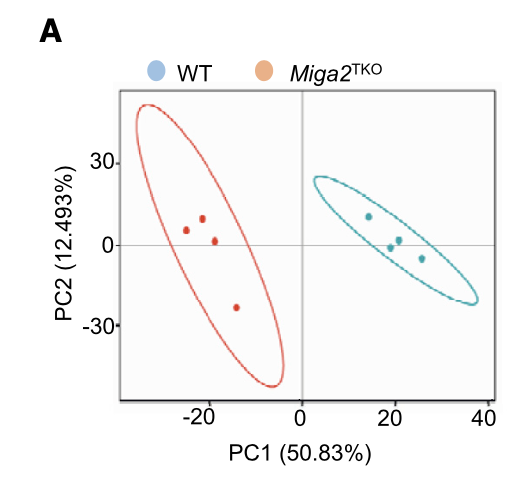
图例是:(A) Partial Least-squares discrimination analysis (PLS-DA) of the serum metabolome of WT and Miga2TKO mice (n = 4). Each symbol represents the data of an individual mouse.
如果你是第一次接触 Partial Least-squares discrimination analysis (PLS-DA) ,你会发现它跟我们一直强调的PCA非常类似,如果你并不是做算法研究,只需要知道这个图可以区分组间差异和组内差异即可。
然后是差异分析
可以使用热图和火山图的展示形式:
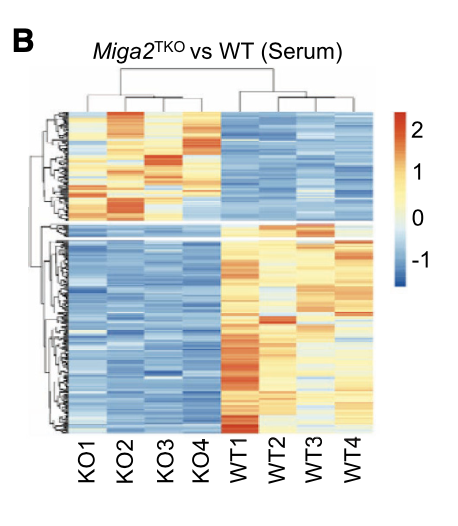
图例是:(B) Heatmap showing differential metabolic production (DMP) in the serum of WT and Miga2TKO mice. The DMPs were identified with a fold change of Miga2TKO/WT > 2.0 or < 0.5.
可以看到仍然是统计学指标P值和变化倍数来进行差异筛选,至于筛选到的是转录组数据的基因列表,还是我们这个代谢组的代谢物列表,不过是表明形式不一样而已!
最后是功能富集(R包 MetaboSignal )
需要注意的是,代谢组数据的差异分析结果,通常是以代谢物为标签,所以它的KEGG数据库的注释呢,也可以使用其专门的包,比如:https://bioconductor.org/packages/release/bioc/html/MetaboSignal.html
library(MetaboSignal)
MS_keggFinder(KEGG_database="organism", match = "rattus")
MS_keggFinder(KEGG_database ="pathway", match = c("glycol", "inositol phosphate","insulin signal", "akt"), organism_code = "rno")
metabo_paths <- c("rno00010","rno00562")
signaling_paths <- c("rno04910", "rno04151")
MetaboSignal_table <- MS_replaceNode(node1 = c("cpd:C00267", "cpd:C00221"),
node2 = "cpd:C00031", MetaboSignal_table)
更深入的用法建议自行研读 MetaboSignal 的文档!
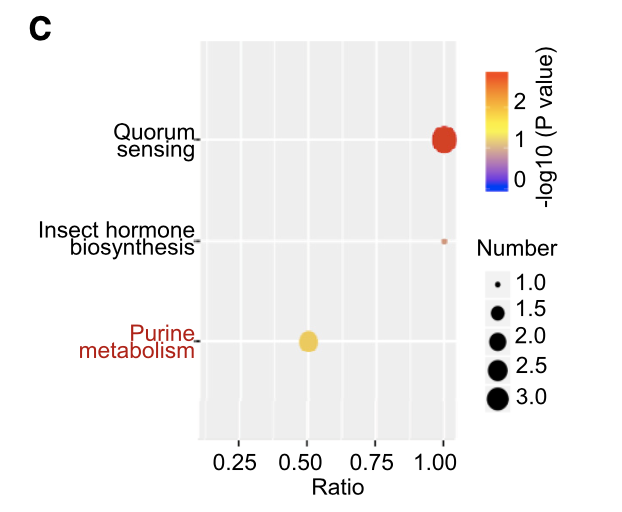
图例是:(C) KEGG analysis of these DMPs-enriched biological processes.
看到这里,你是不是发现这些基本上看我六年前的表达芯片的公共数据库挖掘系列推文即可;
- 解读GEO数据存放规律及下载,一文就够
- 解读SRA数据库规律一文就够
- 从GEO数据库下载得到表达矩阵 一文就够
- GSEA分析一文就够(单机版+R语言版)
- 根据分组信息做差异分析- 这个一文不够的
- 差异分析得到的结果注释一文就够
代谢组学方法描述
首先保证是从6 to 8-week-old mice 体内通过 FACS AriaII cell sorter (BD Biosciences) to obtain naive CD4+ T cells
The oxygen consumption rate (OCR) and extracellular acidification rate (ECAR) were measured in XFp extra- cellular flux analyzers (EFA) (Seahorse Bioscience) by using XFp Cell Mito Stress Test Kit and XFp Glycolysis Stress Test kit, respec- tively.
麻烦的是一般来说代谢组学数据的公开并不是领域惯例,所以这个文章并没有给出任何可以重复它分析图表的数据。学徒作业
前面我们提到过:蛋白质组学数据一般共享在proteome xchange网站,而代谢组学数据,比如文章是 Cell. 2019 May 2;177(4): 里面提到 Data and Software Availability Lipidomics data on liver fractions have been deposited in the EMBL-EBI under accession code MTBLS600. 我就留意了一下这个数据库和ID,是关于MetaboLights,简称为MTBLS。
- https://www.ebi.ac.uk/metabolights/MTBLS600
无需注册即可下载,超级小的文件:
简单读取:library(data.table) a=fread('m_lipidome_retmit_metabolite_profiling_mass_spectrometry_v2_maf.tsv', data.table = F) colnames(a) dat=a[,22:39] pheatmap::pheatmap(dat) b=fread('s_Lipidome RetMit.txt', data.table = F) table(b[,c(5,10)])总共也就是 410个代谢物的信号值,在18个样品:
```
Characteristics[Organism part] Knock-out Scientific Control
Mitochondria-associated Endoplasmic Reticulum Membrane 5 4
mitochondrion 5 4
```
这18个样品首先呢,是的mitochondrion和 Mitochondria-associated Endoplasmic Reticulum Membrane 两个组,各自是9个样品。
然后 各自独立去比较 Knock-out 和Scientific Control的差异 :
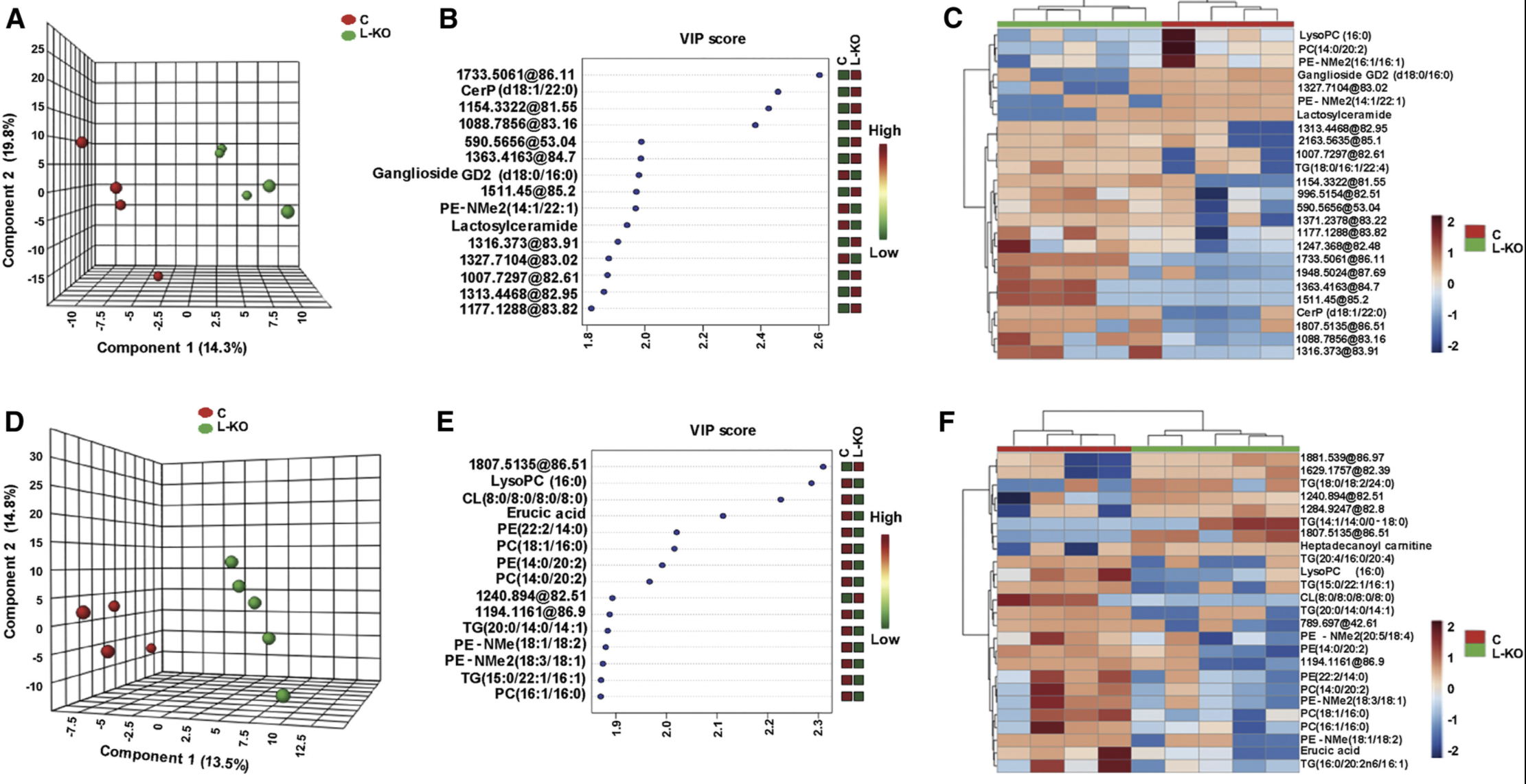
其中 Variable Importance in Projection (VIP) scores 应该是来源于 Partial Least-squares discrimination analysis (PLS-DA) 的分析。