转录组数据处理流程大家应该是耳熟能详了,拿到了测序fastq数据,质量控制,比对,然后定量拿到表达矩阵即可走差异分析流程,
初学者很容易在这个流程里面迷失,因为每个步骤都有十几款软件可以选择,都是好几个参数可以调整, 看起来非常的随心所欲。实际上如果你项目经验丰富,就会理解很多步骤确实是可以随心所欲,因为它并不会对数据分析的结果造成太大的影响。而只要不影响对生物学意义的判断,中间过程马虎一点对初学者来说是一个好事,极大的减少了学习负担。
但是人不可能永远是初学者,但凡有点追求的绝不可能满足于仅仅是跑流程,而且这样的跑流程现在大部分都是被云平台给替代了,如果你不想中年失业,就得想办法自救,突破自己的舒适区。比如可以多看看相关综述,这里给大家推荐一个2020年10月发表的,文章是《Impact of RNA-seq data analysis algorithms on gene expression estimation and downstream prediction》,链接: https://www.nature.com/articles/s41598-020-74567-y
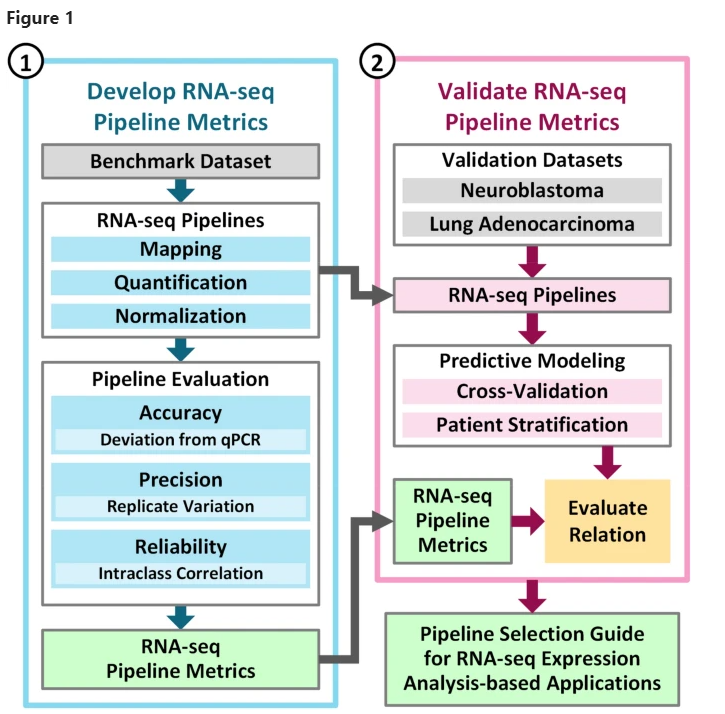
这个综述系统性评价了 美国FDA领导的的 Sequencing Quality Control (SEQC) 计划里面的 278个转录组测序数据的处理,涵盖了:
- 13 sequence mapping,
- three quantification,
- seven normalization methods
主要是3个评价维度,包括: accuracy, precision, and reliability,如果你也想做自己的软件评价课题,这是一个非常好的参考文献哦!
如果你对 Sequencing Quality Control (SEQC) 计划 比较陌生,可以先去看看 FDA-led microarray quality control project (MAQC-I),其实就是芯片表达技术替换成了转录组测序技术,相信在不久的将来也会出现单细胞的质量控制计划。
其实类似的不同转录组数据处理流程评价综述超级多了,随意列举几个如下所示 : - A. RNA-seq gene profiling-a systematic empirical comparison. PLoS ONE 9, e107026 (2014).
- A comprehensive comparison of RNA-Seq-based transcriptome analysis from reads to differential gene expression and cross-comparison with microarrays: A case study in Saccharomyces cerevisiae. Nucleic Acids Res. 40, 804 (2012).
- Gaining comprehensive biological insight into the transcriptome by performing a broad-spectrum RNA-seq analysis. Nat. Commun. 8, 59. https://doi.org/10.1038/s41467-017-00050-4 (2017).
本次测评,作者也给出来了一些软件列表:

可以看到,很多上古软件仍然是在测评范围,不可能是有一个软件可以满足全部的需求,但是如果仅仅是定量而已,我们仍然是推荐我一直使用的流程,超级简单:
希望优秀的你,看完了这个软件测评实验设计流程,也能照猫画虎提出来一个自己的课题,比如单细胞领域的!
另外,也不要错过b站的转录组测序数据分析免费课程啊
- 视频免费在B站:https://www.bilibili.com/video/BV12s41137HY 大家学习的时候记得发弹幕交流哈。
- 也有微云离线版本视频下载本地播放:
-
- 上游分析视频以及代码资料在:https://share.weiyun.com/5QwKGxi
- 下游主要是基于counts矩阵的标准分析的代码 https://share.weiyun.com/50hfuLi
- RNA-SEQ实战演练的素材:https://share.weiyun.com/5h1Z2QY ,包括一些公司PPT,综述以及文献以及测试数据
- RNA-SEQ 实战演练的思维导图:文档链接:https://mubu.com/doc/38y7pmgzLg 密码:p6fo