大家应该是看过了无数R绘图技巧代码,各种R包介绍。我们基本上不写这方面,主要是因为早就过了需要美图引流的阶段,分享自己实践过程的真实代码经验比一味地翻译画图博客和文档好很多。
而且最近看到很多人在朋友圈转发这个: r-graph-gallery ,感觉就这一个网站就可以养活一个公众号了:
网址:http://www.r-graph-gallery.com/
r-garp-gallery收入了大量利用R语言绘制的图形,这些图形包含了很多方面,通过这个网站,我们可以方便直观观察到R语言所能做的一些图形。
Distribution
- Violin
- Density
- Histogram
- Boxplot
- Ridgeline
Correlation
- Scatter
- Heatmap
- Correlogram
- Bubble
- Connected scatter
- Density 2d
Ranking
- Barplot
- Spider / Radar
- Wordcloud
- Parallel
- Lollipop
- Circular Barplot
Part of a whole
- Grouped and Stacked barplot
- Treemap
- Doughnut
- Pie chart
- Dendrogram
- Circular packing
Evolution
- Line plot
- Area
- Stacked area
- Streamchart
- Time Series
Map
- Map
- Choropleth
- Hexbin map
- Cartogram
- Connection
- Bubble map
Flow
- Chord diagram
- Network
- Sankey
- Arc diagram
- Edge bundling
General knowledge
- Ggplot2
- Animation
- Interactivity
- 3D
- Caveats
- Data art
最重要的是每个图都配有示例数据和代码
比如:http://www.r-graph-gallery.com/294-basic-ridgeline-plot.html
出图如下:
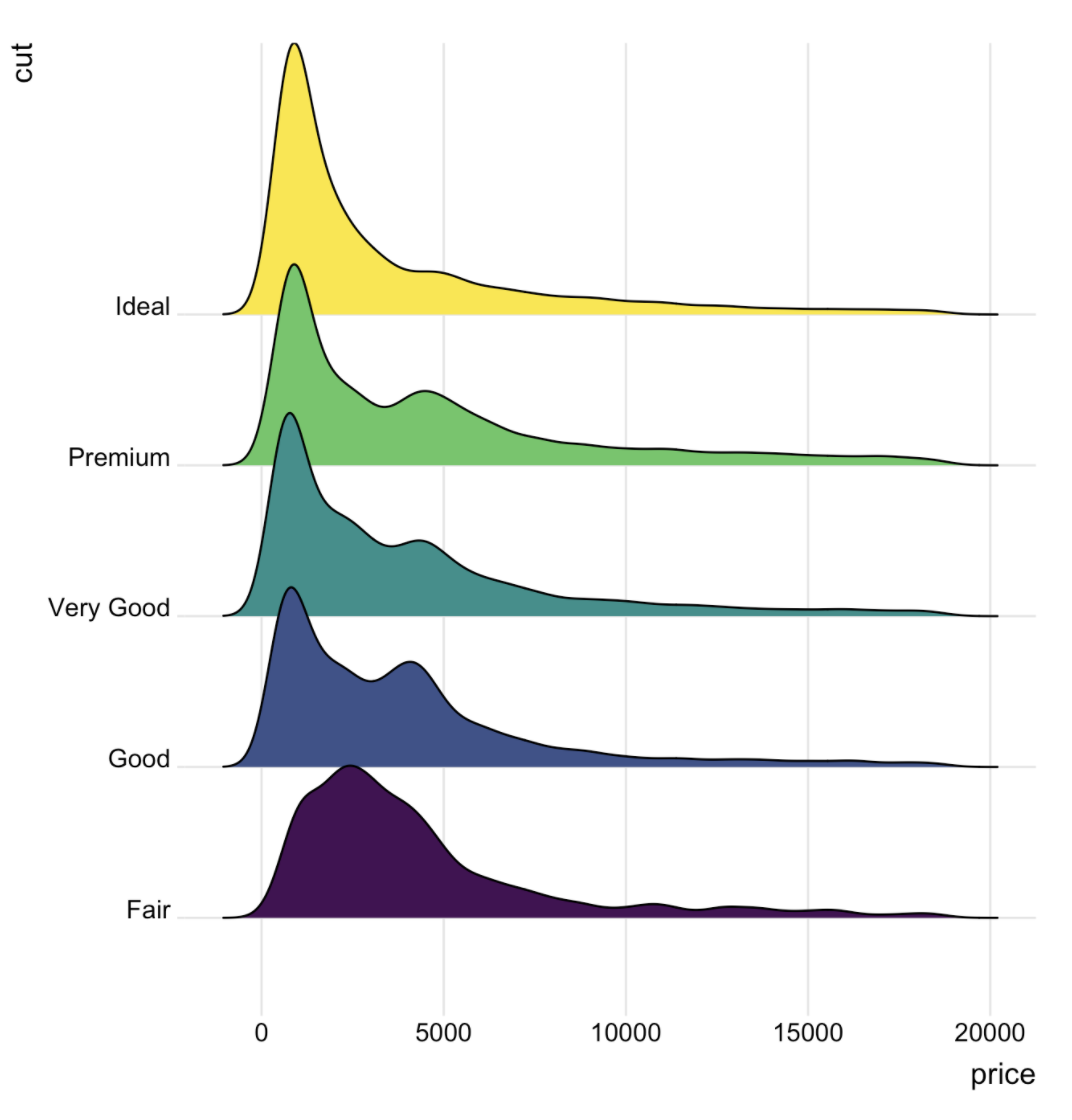
配套代码是:
# library
library(ggridges)
library(ggplot2)
# Diamonds dataset is provided by R natively
#head(diamonds)
# basic example
ggplot(diamonds, aes(x = price, y = cut, fill = cut)) +
geom_density_ridges() +
theme_ridges() +
theme(legend.position = "none")
是不是超级简单啊!
还有更多美图,比如小提琴图也是丰富多彩:http://www.r-graph-gallery.com/violin.html
前提是R语言基础过关啊!
再怎么强调生物信息学数据分析学习过程的计算机基础知识的打磨都不为过,我把它粗略的分成基于R语言的统计可视化,以及基于Linux的NGS数据处理:
把R的知识点路线图搞定,如下:
- 了解常量和变量概念
- 加减乘除等运算(计算器)
- 多种数据类型(数值,字符,逻辑,因子)
- 多种数据结构(向量,矩阵,数组,数据框,列表)
- 文件读取和写出
- 简单统计可视化
- 无限量函数学习
Linux的6个阶段也跨越过去 ,一般来说,每个阶段都需要至少一天以上的学习:
- 第1阶段:把linux系统玩得跟Windows或者MacOS那样的桌面操作系统一样顺畅,主要目的就是去可视化,熟悉黑白命令行界面,可以仅仅以键盘交互模式完成常规文件夹及文件管理工作。
- 第2阶段:做到文本文件的表格化处理,类似于以键盘交互模式完成Excel表格的排序、计数、筛选、去冗余,查找,切割,替换,合并,补齐,熟练掌握awk,sed,grep这文本处理的三驾马车。
- 第3阶段:元字符,通配符及shell中的各种扩展,从此linux操作不再神秘!
- 第4阶段:高级目录管理:软硬链接,绝对路径和相对路径,环境变量。
- 第5阶段:任务提交及批处理,脚本编写解放你的双手。
- 第6阶段:软件安装及conda管理,让linux系统实用性放飞自我。