前面的教程:[混合到同一个10X样品里面的多个细胞系如何注释](https://mp.weixin.qq.com/s/S5TJ2JDOYAOynhjXwTH-FQ),我们提到了可以使用细胞系的表达量矩阵去跟细胞亚群表达量矩阵进行相关性计算,然后就可以判断细胞亚群的生物学意义啦。当然了,我们也给出来了一个比较不错的可视化方法,见:[如果你觉得相关性热图不好看,或者太简陋](https://mp.weixin.qq.com/s/jrV5HER_bmWyvXGzi608Tw)。
如何很多朋友留言问,为什么不使用现成的工具呢,比如SingleR就构建自定义细胞亚群数据库。我们当然知道这样的工具很好用,但是我们要分享的是技术细节,如果一切都使用现成的工具,就都被包装起来了,成为了一个黑匣子。
而现成工具,其实就在于熟读文档罢了,SingleR构建自定义细胞亚群数据库,我这里也给大家演示一下:
```r
rm(list = ls())
library(SingleR)
library(Seurat)
library(ggplot2)
# 读入scRNA数据 -------
scRNA <- readRDS("../step1_聚类/sce_all.Rds")
table(Idents(scRNA) )
Idents(scRNA) <- "RNA_snn_res.0.2"
table(Idents(scRNA) )
# 读入参考数据集 -------
Ref <- read.csv("../step2_注释/processed_reference.csv")
Ref <- textshape::column_to_rownames(Ref, loc = 1)
head(Ref)
```
可以看到每个细胞系都有自己的表达量,如下所示的一个矩阵,在R里面就是一个数据框。

接下来才是SingleR构建自定义细胞亚群数据库,其实调用的是SingleCellExperiment这个对象构建的模式,主要是scater包需要学习一下,代码如下:
```r
ref_sce=SingleCellExperiment::SingleCellExperiment(assays=list(counts=Ref))
ref_sce=scater::logNormCounts(ref_sce)
library(SingleCellExperiment)
logcounts(ref_sce)[1:4,1:4]
colData(ref_sce)$Type=colnames(Ref)
ref_sce
```
有了SingleR构建自定义细胞亚群数据库,接下来我们只需要把自己的单细胞矩阵提取出来即可;
```r
testdata <- GetAssayData(scRNA, slot="data")
pred <- SingleR(test=testdata, ref=ref_sce,
labels=ref_sce$Type,
#clusters = scRNA@active.ident
)
table(pred$labels)
head(pred)
```
可以看到,两个矩阵使用SingleR函数处理一下,就可以拿到了 单细胞的亚群映射关系,如下所示:
```
> as.data.frame(table(pred$labels))
Var1 Freq
1 HEK293T 835
2 MCF10A.x 71
3 MCF10A.y 396
4 MCF7.x 30
5 MCF7.y 1177
6 MDAMB134VI 478
7 SUM44 316
8 T47D.x 966
9 T47D.y 345
```
这个时候的我们也可以把SingleR对细胞系表达量矩阵和我们单细胞矩阵的相关性矩阵提取出来:
```
pred@listData[["scores"]]
```
就是前面的 全部的细胞系和全部的具体的每个单细胞的表达相关性矩阵(Pearson correlation coefficient)
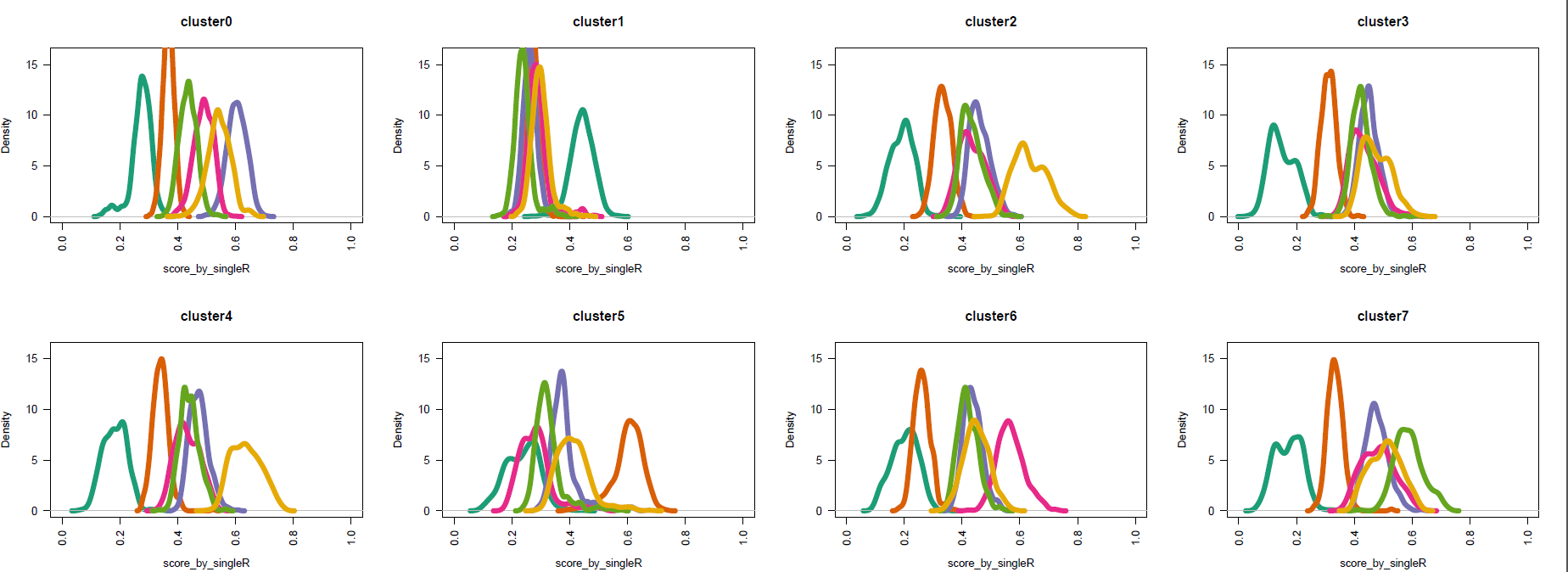
可以看到,殊路同归!