我们七月份的学徒培养专注于单细胞数据处理,第一个学徒选择的文章很有意思,标题是:《Single-cell transcriptomic heterogeneity in invasive ductal and lobular breast cancer cells》
这个单细胞文章仅仅是单个10X样品,但是测8个细胞系,流程如下所示:
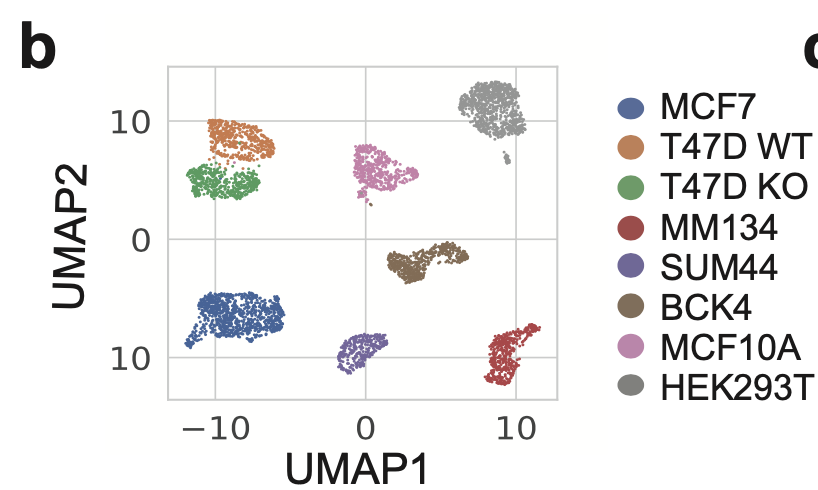
Number of cells:
- MCF7 (n=977)
- T47D WT (n=509)
- T47D KO (n=491)
- MM134 (n=439)
- SUM44 (n=314)
- BCK4 (n=512)
- MCF10A (n=491)
- HEK293T(n=881).
可以看到,不同细胞系,降维聚类分群后,泾渭分明;
数据在其GEO上面,可以很容易下载,然后走seurat流程,降维聚类分群就可以跟文章做得差不多的umap图,不过自己分群后的8个亚群需要跟文章里面的8个细胞系进行对应,这个过程就略微有点复杂。这个文献及其数据处理也会纳入我们的两个b站单细胞栏目,持续更新半年,基本上涵盖了大家需要的技能:
- https://www.bilibili.com/video/BV1DK4y1X7bb/ 更新至第8篇,「生信技能树」100个单细胞文献解读(8/100)
- https://www.bilibili.com/video/BV19Q4y1R7cu/ section 3已更新,「生信技能树」单细胞公开课2021
文末做一个小调研,你会混合多个样品进行一次10X单细胞转录组测序吗?因为10X样品拿到的细胞数量范围是5-10千,所以理论上并不能无限制混样品哦。
而且混合之后,如何把样品拆分开来,也是一个问题。
参考前面的例子:人人都能学会的单细胞聚类分群注释 ,我们演示了第一层次的分群。
如果你对单细胞数据分析还没有基础认知,可以看基础10讲: