在单细胞大行其道的如今,也有不少课题组选择做cytof数据,全称是:Cytometry + Time Of Flight,是Fluidigm公司的产品,可以检测单个细胞的多个设定好的抗体的信号强度。这些抗体 对应的基因通常是目前科研界认识比较清晰的,可以用来分群的,而且该仪器可以一次性检测数十万单细胞,有点类似于靶向单细胞转录组测序。因为很多情况下,并不需要全部的基因的转录水平的信息。
我在《生信技能树》发布了cytof这样的质谱流式数据处理系列文字版教程:
- 1.cytof数据资源介绍(文末有交流群)
- 2.cytofWorkflow之读入FCS文件(一)
- 3.cytofWorkflow之构建SingleCellExperiment对象(二)
- 4.cytofWorkflow之基本质量控制(三)
- 5.cytofWorkflow之聚类分群(四)
- 6.cytofWorkflow之人工注释生物学亚群(五)
- 7.cytofWorkflow之亚群比例差异分析(六)
只需自己准备好FCS格式的数据文件,后续全部的R里面的统计可视化。再怎么强调生物信息学数据分析学习过程的计算机基础知识的打磨都不为过,我把它粗略的分成基于R语言的统计可视化,以及基于Linux的NGS数据处理:
把R的知识点路线图搞定,如下:
- 了解常量和变量概念
- 加减乘除等运算(计算器)
- 多种数据类型(数值,字符,逻辑,因子)
- 多种数据结构(向量,矩阵,数组,数据框,列表)
- 文件读取和写出
- 简单统计可视化
- 无限量函数学习
发现自己的教程对原理介绍有点少
恰好最近学徒推荐了一个视频给我,里面对降维聚类分群的原理介绍的比较清晰,尤其是《3.1 Phenograph理论介绍》,以及《4.1 tSNE介绍》,非常值得听哦!
该视频的学习目录如下;
- 0.1 准备工作(1)
- 0.1 准备工作(2)
- 1.1 Data Cleaning
- 2.1 数据预处理
- 2.2 数据预处理(R)
- 3.1 Phenograph理论介绍
- 3.2 Phenograph(R)
- 4.1 tSNE介绍
- 4.2 tSNE(R)
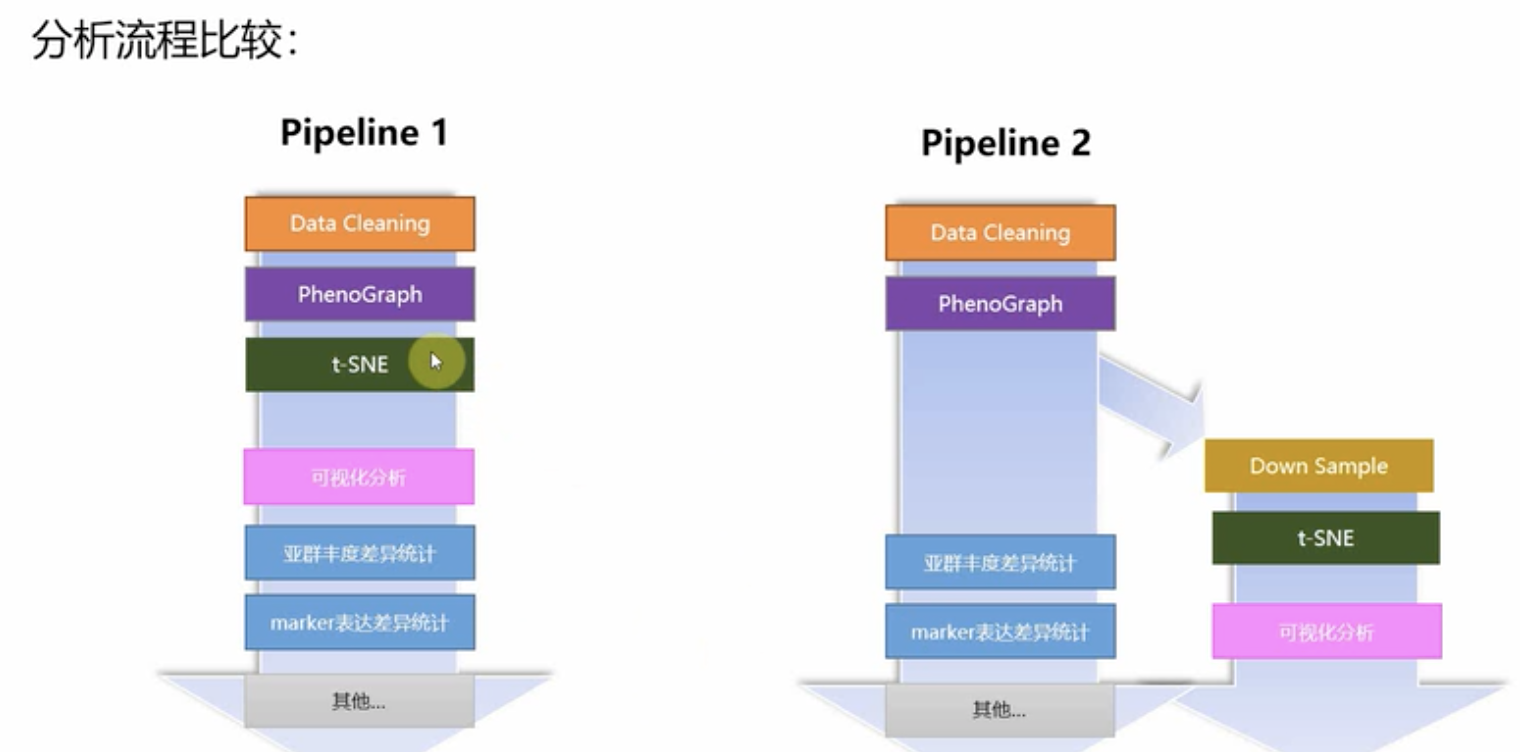
- 5.1 第五节 数据分析流程(Pipeline1)
- 5.2 Pipeline1(R):聚类和降维
- 5.3 Pipeline1(R):Cytofkit Shinny App(可视化)
- 5.4 Pipeline1(R):可视化分析
- 5.5 Pipeline1(R):统计画图前的准备
- 5.6 Pipeline1(R):Abundance统计
- 5.7 Pipeline1(R):Expression统计,Heatmap
- 6.1 第六节 数据分析流程(Pipeline2)
- 6.2 Pipeline2(R):Down-sample
组织形式非常好,而且提前全套的练习数据和代码哦(虽然代码写的很一般)
教程相关代码,Demo数据见以下链接:
链 接: https://pan.baidu.com/s/1pEtw-1R2KuOjJYFe_QGQ8g 提取码: sd32
压缩包解压密码:123
视频全部免费在b站哦,下面的二维码直达:

或者复制粘贴链接( https://www.bilibili.com/video/BV1cJ41137Pn?p=1 )到浏览器打开!
再次强调
该系列视频,对于数据处理的原理介绍比较好,但是代码其实并不值得推荐。如果是处理数据,仍然是推荐我的系列文字版教程:
- 1.cytof数据资源介绍(文末有交流群)
- 2.cytofWorkflow之读入FCS文件(一)
- 3.cytofWorkflow之构建SingleCellExperiment对象(二)
- 4.cytofWorkflow之基本质量控制(三)
- 5.cytofWorkflow之聚类分群(四)
- 6.cytofWorkflow之人工注释生物学亚群(五)
- 7.cytofWorkflow之亚群比例差异分析(六)
当然了,视频里面的一系列可视化方法也值得借鉴,不过对于掌握了R语言的小伙伴来说, 自己的写绘图函数简直就是小菜一碟啦。
另外值得一提的是,视频里面介绍的流程思维: